
扩展功能
文章信息
- 孙悦龙, 刘浩, 朱宝成, 张伟涛, 郭晓军
- SUN Yuelong, LIU Hao, ZHU Baocheng, ZHANG Weitao, GUO Xiaojun
- 枯草芽孢杆菌N2-10的基因组测序和比较基因组分析
- Genome sequencing and comparative genomics analysis of Bacillus subtilis N2-10
- 微生物学通报, 2023, 50(1): 131-147
- Microbiology China, 2023, 50(1): 131-147
- DOI: 10.13344/j.microbiol.china.220556
-
文章历史
- 收稿日期: 2022-06-09
- 接受日期: 2022-06-25
- 网络首发日期: 2022-07-27
2. 河北省饲用微生物技术创新中心, 河北 保定 071000;
3. 农业废弃物资源化利用河北省工程研究中心, 河北 保定 071000;
4. 河北省畜牧总站, 河北 石家庄 050000
2. Hebei Province Feed Microorganism Technology Innovation Center, Baoding 071000, Hebei, China;
3. Hebei Engineering Research Center of Agricultural Waste Resource Utilization, Baoding 071000, Hebei, China;
4. Hebei Animal Husbandry Station, Shijiazhuang 050000, Hebei, China
枯草芽孢杆菌(Bacillus subtilis)作为发酵饲料的常用菌株,在提高动物生产性能、增强动物机体免疫能力、改善动物机体肠道环境等方面效果显著[1]。枯草芽孢杆菌属于革兰氏阳性菌[2],是一种好氧化能异养型细菌,具有耐酸碱、耐高温、耐盐等特性[3],能够产生氨基酸、维生素、有机酸和生长因子等多种参与机体新陈代谢的产物,而且分泌多种消化酶如蛋白酶、淀粉酶等[4],有效促进蛋白质、淀粉、多糖等有机物的分解[5]。
研究表明,枯草芽孢杆菌可有效提高家禽饲料能量转化率,促进家禽生长生产,提高肉、蛋产量,改善肉、蛋品质,降低家禽疾病发生率[6]。李靖[7]在奶牛日粮中加入枯草芽孢杆菌BS041可降低瘤胃中弯曲菌属和葡萄球菌属的丰度水平;程连平[8]在湖羊日粮中添加枯草芽孢杆菌制剂后发现,免疫球蛋白、白细胞介素含量显著提高,瘤胃液氨态氮、有机酸含量显著提高;陈志敏等[9]应用枯草芽孢杆菌发酵肉鸡饲料,当发酵饲料替代10%基础日粮饲喂肉鸡后,可以显著提高生长性能,肉鸡肠道形态得到明显改善;王震[10]应用枯草芽孢杆菌发酵麸皮,当发酵饲料替代10%基础日粮饲喂育肥猪时,显著提高平均日增重,降低料重比。朱燕丽[11]将枯草芽孢杆菌发酵麸皮饲喂肉鸡发现,发酵麸皮在提高肉鸡生长生产性能等方面发挥巨大作用。
本实验室前期筛选到一株枯草芽孢杆菌N2-10,其具有较强的抑菌能力且还可以产木质纤维素酶、淀粉酶、蛋白酶等多种消化酶[12],该菌株发酵秸秆后饲喂绵羊,可明显改善绵羊瘤胃的微生物多样性[13]。此外,将其应用在家禽全价配合饲料上,显著提高了蛋鸡生长性能、生产性能,降低死淘率,有效抑制多种肠道病原菌[14]。为了更好地研究该菌株的抑菌机制和发酵饲料在畜禽养殖中的效果,通过二代联合三代测序对枯草芽孢杆菌N2-10进行了全基因组测序、功能注释和次级代谢产物分析,并将该菌株与NCBI数据库中下载得到的模式菌株枯草芽孢杆菌168进行比较基因组分析,以期为高效开发利用枯草芽孢杆菌N2-10提供生物信息学基础。
1 材料与方法 1.1 材料 1.1.1 菌株枯草芽孢杆菌(Bacillus subtilis) N2-10,保存于河北农业大学农牧微生物实验室。
1.1.2 培养基NB培养基(g/L):牛肉膏3.00,蛋白胨10.00,NaCl 5.00。NA培养基(g/L):牛肉膏3.00,蛋白胨10.00,NaCl 5.00,琼脂20.00。
1.2 方法 1.2.1 菌株基因组提取及测序将−80 ℃保存的枯草芽孢杆菌N2-10划线接种于NA培养基上,37 ℃培养24 h,挑取单菌落接种于100 mL NB培养基中,37 ℃、220 r/min培养24 h,将菌液12 000 r/min离心5 min收集菌体,送北京诺禾致源科技股份有限公司测序,库检合格后进行PacBio Sequel和Illumina NovaSeq PE150测序。
1.2.2 基因组组装及优化原始下机数据过滤质控后,利用SMRT Link V5.0.1软件(https://www.pacb.com/support/software-downloads/)对原始下机数据进行初步组装,将组装的数据校正后得到基因组组装数据。将组装数据优化后与组装结果进行比对分析,进行测序结果的矫正[15]。
1.2.3 基因组组分分析利用Glimmer V3.02 (http://ccb.jhu.edu/software/glimmer/index.shtml)和GeneMarkS V4.3软件(http://topaz.gatech.edu/GeneMark)预测枯草芽孢杆菌N2-10基因组的编码基因[16-17];利用tRNAscan-SE V2.0软件(http://trna.ucsc.edu/software/)预测tRNA[18];采用rRNAmmer V1.2软件(http://www.cbs.dtu.dk/services/RNAmmer/)对rRNA进行分析[19];使用BLAST+V2.3.0软件(ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/2.3.0/)对SnRNA进行预测[20];利用Minced V3.0软件(https://github.com/ctSkennerton/minced)预测CRISPR-Cas数量[21]。利用PHAST V2.3软件(https://sourceforge.net/projects/phage-finder/files/)预测前噬菌体个数[22];利用IslandViewer V4.0软件(https://www.pathogenomics.sfu.ca/islandviewer/resources/)预测基因岛数量[23]。
1.2.4 基因功能分析通过Blast2go V2.5软件(https://www.blast2go.com/)对GO (gene ontolog)数据库进行注释[24];通过eggNOG V4.5.1软件(http://eggnogdb.embl.de/#/app/home)对COG (clusters of orthologous groups of proteins)数据库进行注释[25];通过KEGG (Kyoto encyclopedia of genes and genomes)在线数据库(http://www.genome.jp/kegg/)对KEGG进行注释[26];通过CAZy (carbohydrate-active enzymes) V6.0软件(http://www.cazy.org/)对CAZy数据库进行注释[27];通过antiSMASH V4.0.2软件(https://dl.secondarymetabolites.org/releases/4.0.2/)对次级代谢产物进行注释[28]。
1.2.5 比较基因组分析利用IQ-TREE V1.6.12软件(http://www.iqtree.org/release/v1.6.12)构建同源单拷贝基因系统发育树,建树方法为maximum likelihood;使用Mummer V4.0分析软件(https://mummer4.github.io/)进行共线性分析,默认软件参数;基因组组分比较分析同1.2.3;基因组功能比较分析同1.2.4。
2 结果与分析 2.1 基因组测序及分析枯草芽孢杆菌N2-10基因组中包含一条染色体,大小为4 036 899 bp,GC含量为43.88%;共编码4 163个编码基因,所有编码基因总长度为3 594 369 bp,编码基因平均长度为863 bp,编码区总长度占基因组总长度的89.04%;含有85个tRNA,10个5S rRNA、10个16S rRNA、10个23S rRNA;2个CRISPR-Cas,CRISPR1所在区域为494 724−494 986,含有5个重复序列,重复序列平均长度为23 bp,间隔序列平均长度37 bp,CRISPR2所在区域为3 545 037−3 545 307,含有4个重复序列,重复序列平均长度为28 bp,间隔序列平均长度53 bp;1个前噬菌体位于1 313 578−1 348 279处,序列总长34 702 bp,包含46个基因;含有基因岛6个,基因岛总长度为192 610 bp,平均长度为19 261 bp。菌株N2-10的基因组圈图见图 1,其基因组序列已提交至GenBank数据库,登录号为CP098417。
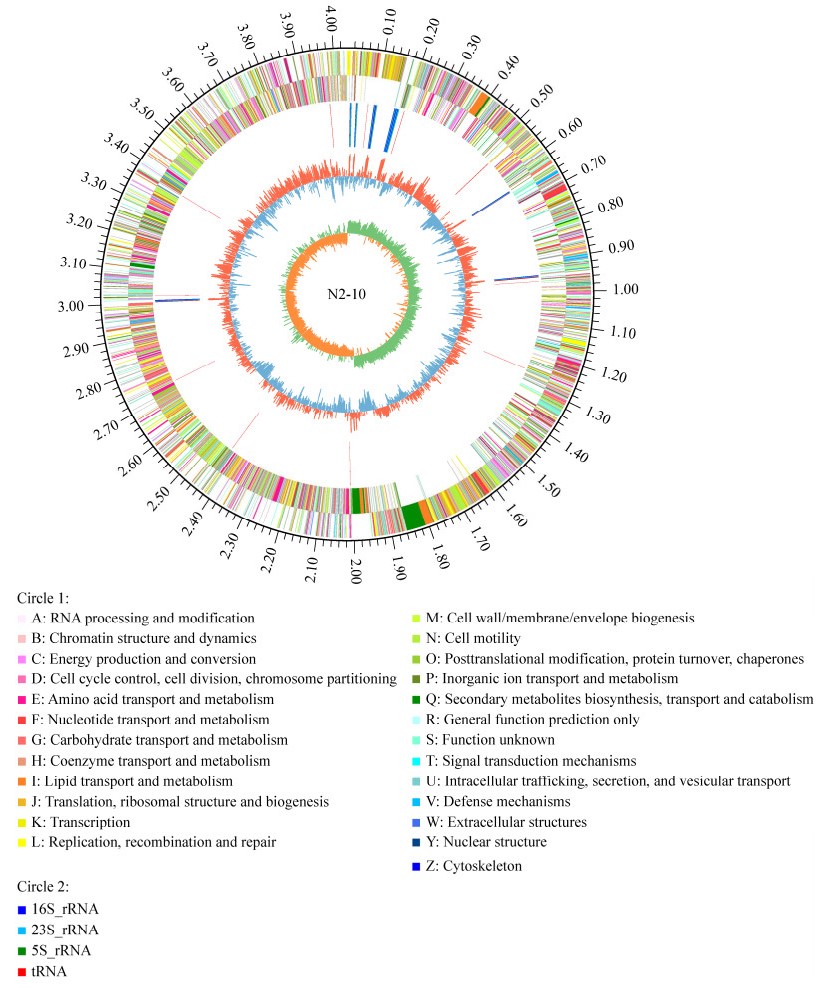
|
| 图 1 枯草芽孢杆菌N2-10基因组圈图 Figure 1 The circos of Bacillus subtilis N2-10. |
|
|
菌株N2-10的GO注释结果见图 2,共有3 048个基因注释到GO功能,占基因组的73.22%。其中与细胞组成(cellular component)相关的基因数为42.69%,与分子功能(molecular function)相关的基因数为57.03%,与生物过程(biological process)相关的基因数为56.40%。在生物过程方面,与氧化还原(oxidation reduction process)、转录调控(regulation of transcription)等方面相关的基因占比18.23%;在分子功能方面,与DNA结合(DNA binding)、ATP结合(ATP binding)、水解酶活性(hydrolase activity)等方面相关的基因占比20.20%;在细胞组成方面,与生物膜组成(integral component of membrane)等方面相关的基因占比45.65%。
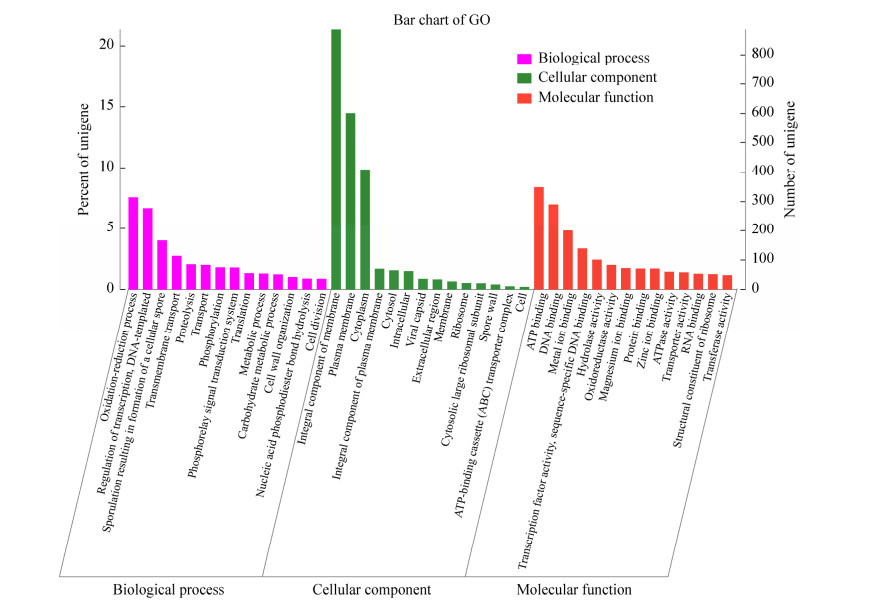
|
| 图 2 枯草芽孢杆菌N2-10的GO功能分类图 Figure 2 Gene ontology annotation functional classification chart of Bacillus subtilis N2-10. |
|
|
菌株N2-10的COG注释结果见图 3,共有3 177个基因注释到4类COG功能,20种类型,占基因组的76.32%。除19.96%的基因未注释到具体功能,菌株N2-10的功能主要集中在氨基酸的转运与代谢(amino acid transport and metabolism) (7.30%)、转录(transcription) (6.27%)、碳水化合物转运与代谢(carbohydrate transport and metabolism) (6.22%)、无机离子转运与代谢(inorganic ion transport and metabolism) (4.78%)等方面。此外,有1.37%的基因被注释到与次级代谢产物转运和代谢(secondary metabolites biosynthesis, transport and catabolism)相关。
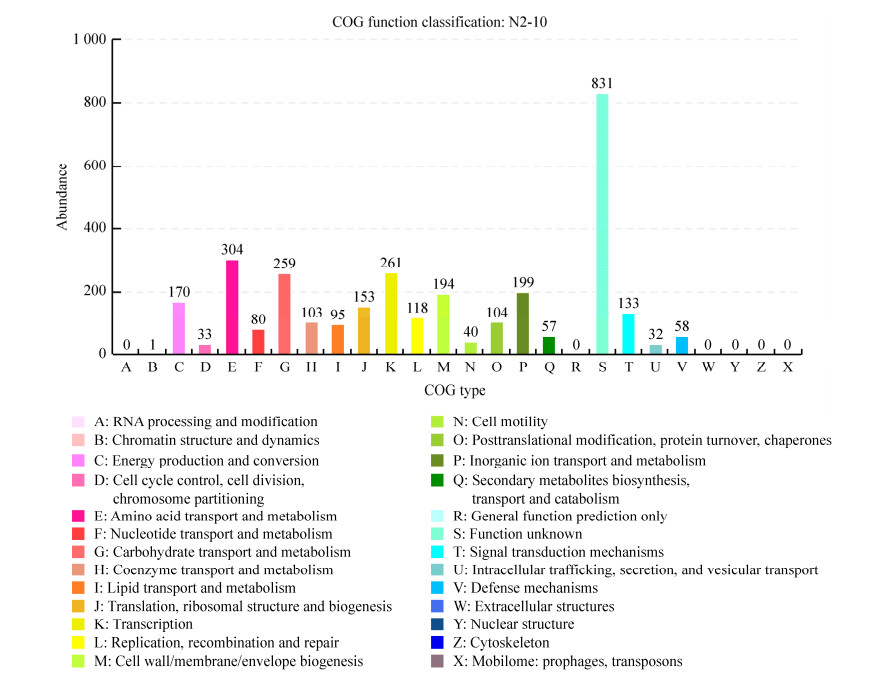
|
| 图 3 枯草芽孢杆菌N2-10的COG功能分类图 Figure 3 COG annotation functional classification chart of Bacillus subtilis N2-10. |
|
|
N2-10菌株的KEGG注释结果见图 4,共有3 894个基因注释到功能信息,占基因组的93.54%。与细胞过程(cellular process)具有相关性的基因数为3.89%,与代谢(metabolism)具有相关性的基因数为31.40%,与人类疾病(human diseases)具有相关性的基因数为2.02%,与遗传信息处理(genetic information processing)具有相关性的基因数为4.42%,与生物系统(organismal systems)具有相关性的基因数为0.89%,与环境信息处理(environmental information processing)具有相关性的基因数为7.76%。在KEGG功能注释中,与代谢通路具有相关性的基因数最多,其中碳水化合物代谢(carbohydrate metabolism)占比6.78%,其次是氨基酸代谢(amino acid metabolism)占比4.83%。另外,发现有44个基因与萜类和聚酮化合物的代谢(metabolism of terpenoids and polyketides)相关,其中,包括表面活性素生物合成(surfactin family lipopeptide synthetase A, B, C)及丰原素生物合成(fengycin family lipopeptide synthetase A, B, C, D, E)。

|
| 图 4 菌株N2-10的KEGG功能分类图 Figure 4 KEGG annotation functional classification chart of the Bacillus subtilis N2-10. |
|
|
菌株N2-10的CAZy注释结果见图 5,共有145个基因注释到6类活性酶。其中,37.24%的基因被注释为糖苷水解酶(glycoside hydrolases),30.34%的基因被注释为糖基转移酶(glycosyl transferases),20.69%的基因被注释为碳水化合物酯酶(carbohydrate esterases),4.83%的基因被注释为多糖裂解酶(polysaccharide lyases),4.83%的基因被注释为辅助活性(auxiliary activity),还有2.07%的基因被注释为碳水化合物酶结合模块(carbohydrate-binding modules)。在糖苷水解酶中,覆盖度最高的主要是β-葡萄糖苷酶(EC 3.2.1.21)、β-1, 4-木聚糖内切酶(EC 3.2.1.8)、内切葡聚糖酶(EC 3.2.1.4)、几丁质酶(EC 3.2.1.14)、半乳糖苷酶(EC 3.2.1.103)等。在糖基转移酶中,UDP-葡萄糖醛酸转移酶(EC 2.4.1.11)、纤维素合酶(EC 2.4.1.12)、蔗糖合酶(EC 2.4.1.13)等具有较高的覆盖度。此外,乙酰木聚糖酯酶(EC 3.1.1.72)在碳水化合物酯酶中具有较高的覆盖度。
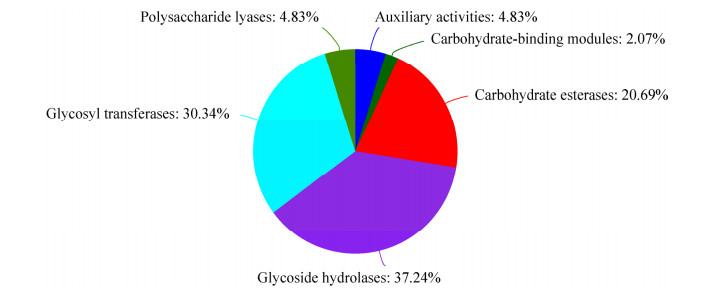
|
| 图 5 菌株N2-10的CAZy功能分类图 Figure 5 CAZy annotation functional classification chart of Bacillus subtilis N2-10. |
|
|
N2-10菌株的次级代谢产物注释结果见表 1,共注释到10个次级代谢产物合成基因簇,分别为3个非核糖体多肽合成酶(nrps)、2个萜烯(terpene)、2个塞克肽类(sactipeptide-headtotail)、1个nrps-transatpks-otherks、1个第三类聚酮合酶(t3pks)和1个尚不明确的代谢产物。在注释的基因簇中,根霉素(rhizocticin A)、表面活性素(surfactin)、bacillaene、丰原素(fengycin)、儿茶酸铁载体杆菌苷(bacillibactin)、芽孢杆菌素(subtilosin A)、溶杆菌素(bacilysin)生物合成基因簇为已知基因簇。此外,还有3个基因簇未注释到明确的代谢产物。
| Cluster ID | Type | Start | End | Similar cluster | Similarity (%) | Gene No. |
| Cluster1 | Sactipeptide-head_to_tail | 204 809 | 226 299 | Rhizocticin biosynthetic | 32 | 21 |
| Cluster2 | Nrps | 357 110 | 422 501 | Surfactin biosynthetic | 82 | 49 |
| Cluster3 | Terpene | 1 149 006 | 1 169 812 | Other | 0 | 24 |
| Cluster4 | Nrps-transatpks-otherks | 1 768 961 | 1 878 789 | Bacillaene biosynthetic | 100 | 56 |
| Cluster5 | Nrps | 1 939 075 | 2 022 487 | Fengycin biosynthetic | 100 | 45 |
| Cluster6 | Terpene | 2 088 267 | 2 110 165 | Other | 0 | 23 |
| Cluster7 | t3pks | 2 158 621 | 2 199 716 | Other | 0 | 47 |
| Cluster8 | Nrps | 3 090 909 | 3 140 702 | Bacillibactin biosynthetic | 100 | 47 |
| Cluster9 | Sactipeptide-head_to_tail | 3 659 514 | 3 681 125 | Subtilosin A biosynthetic | 100 | 21 |
| Cluster10 | Other | 3 684 111 | 3 725 527 | Bacilysin biosynthetic | 100 | 43 |
菌株N2-10基因组序列经与NCBI中GenBank比对后,与枯草芽孢杆菌168 (Bacillus subtilis AL009126.3)、杯状芽孢杆菌5L6 (Cytobacillus ciccensis CP041305.1)、地衣芽孢杆菌ATCC 14580 (Bacillus licheniformis CP034569.1)、解淀粉芽孢杆菌DSM 7 (Bacillus amyloliquefaciens FN597644.1)、死谷芽孢杆菌DSM 11031 (Bacillus vallismortis CP026362.1)具有较高的相似性。如图 6所示。N2-10与168菌株聚在一起,说明N2-10与168菌株进化距离最近。

|
| 图 6 同源单拷贝基因进化树标尺代表序列差异表现的分支长度;分支点上的数字代表自展值 Figure 6 Phylogenetic tree of homologous single copy genes. The bar shows the branch length represented by sequence differences; Numbers at the node represent the bootstrap values. |
|
|
在上一步分析中,N2-10与168菌株基因组相似性最高,将2株菌全基因组序列进行比对分析,通过共线性分析进一步识别了两基因组的一致性及变异性。菌株N2-10的共线性分析结果见图 7,N2-10与168菌株基因组中存在大量同源性基因,表明N2-10与168菌株具有良好的共线性;而非同源性基因代表了菌株N2-10具有自身特异性。
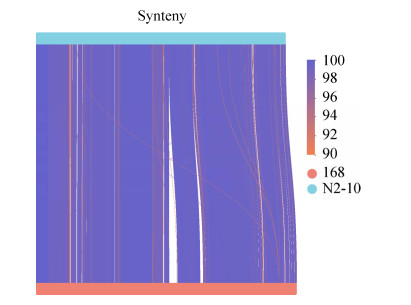
|
| 图 7 枯草芽孢杆菌N2-10和168共线性分析结果 Figure 7 Alignment results of Bacillus subtilis N2-10 and 168. |
|
|
为进一步探究两基因组的差异,对两基因组特征进行了比较(表 2)。由表 2可知,菌株N2-10基因组碱基数小于168菌株,GC含量大于168菌株。N2-10菌株基因组的编码基因数、基因岛数、前噬菌体数与168菌株存在差异。
| Items | Bacillus subtilis 168 |
Bacillus subtilis N2-10 |
| Genome size (bp) | 4 215 606 | 4 036 899 |
| Scaffold | 1 | 1 |
| GC content (%) | 43.51 | 43.88 |
| Coding sequences | 4 417 | 4 153 |
| tRNA | 85 | 85 |
| rRNA | 30 | 30 |
| Gene islands | 5 | 6 |
| Prophage | 3 | 1 |
| CRISPR | 2 | 2 |
菌株N2-10与168在GO功能数据库中的数值比较结果具有较高的相似性,均注释到三类生物学功能,但菌株N2-10注释到的总基因数少于168菌株,仅为3 046个基因。N2-10基因组中与细胞组成、分子功能、生物过程相关的基因数均少于168菌株。菌株N2-10与168菌株在COG功能数据库中的数值结果具有较高的相似性,菌株N2-10注释到的基因数少于168菌株基因组注释到的基因数,但在COG类型上无差异。根据KEGG注释结果,在碳水化合物代谢通路中,相比于168菌株,N2-10菌株缺失了1个UTP-1-磷酸葡萄糖尿酸转移酶编码基因(EC 2.7.7.9)、UDP-葡萄糖-4-异构酶编码基因(EC 5.1.3.2)、N-乙酰氨基葡萄糖专用组件编码基因(EC 2.7.1.193);增加了1个果糖激酶编码基因(EC 2.7.1.4)、β-果糖呋喃糖苷酶编码基因(EC 3.2.1.26)。在氨基酸代谢通路中,相较于168菌株,菌株N2-10具有乙酰乙酸酯CoA-转移酶α亚基编码基因(EC 2.8.3.9)、缺失了单胺氧化酶编码基因(EC 1.4.3.4)。在碳水化合物降解酶注释方面,菌株N2-10注释的糖苷水解酶编码基因数少于168菌株,糖基转移酶、碳水化合物酯酶编码基因多于168菌株。在糖苷水解酶编码基因中,菌株N2-10缺失了1个多聚半乳糖醛酸内切酶编码基因(EC 3.2.1.15);在糖基转移酶编码基因中,相较于168菌株,菌株N2-10具有4-氨基-4-脱氧-1-阿拉伯糖基转移酶(EC 2.4.2.43)、脂质-A-二糖合酶编码基因(EC 2.4.1.182);在碳水化合物酯酶编码基因中,N2-10菌株相比168菌株增加了3个乙酰木聚糖酯酶编码基因(EC 3.1.1.72)。
如表 3所示,168菌株共注释到11个次级代谢产物合成基因簇,其中包括7个已知合成基因簇和4个未知合成基因簇。两个基因组中7个次级代谢产物合成基因簇相似性较高,每个代谢产物的编码基因数量和基因所在位置具有差异性。在N2-10菌株基因组中,根霉素(rhizocticin A)基因簇始末位置为204 809−226 299、表面活性素(surfactin)基因簇的始末位置为357 100−422 501、bacillaene基因簇的始末位置为1 768 961−1 878 789、丰原素(fengycin)基因簇的始末位置为1 939 075−2 022 487、儿茶酸铁载体杆菌苷(bacillibactin)基因簇的始末位置3 090 909−3 140 702、芽孢杆菌素(subtilosin A)基因簇的始末位置为3659 514−3 681 125、溶杆菌素(bacilysin)基因簇的始末位置为3 684 111−3 725 527;而在168菌株中,根霉素A基因簇始末位置为205 404−226 894、表面活性素基因簇的始末位置为356 968−422 359、bacillaene基因簇的始末位置为1 768 725−1 878 521、丰原素基因簇的始末位置为1 934 526−2 017 956、儿茶酸铁载体杆菌苷基因簇的始末位置为3 260 520−3 310 259、芽孢杆菌素基因簇的始末位置为3 826 058−3 847 669、溶杆菌素基因簇的始末位置为3 850 669−3 892 085。
| Name | Bacillus subtilis 168 |
Bacillus subtilis N2-10 |
| Rhizocticin A | 23 | 21 |
| Surfactin | 47 | 49 |
| Other | 24 | 24 |
| Bacillaene | 55 | 56 |
| Fengycin | 46 | 45 |
| Other | 23 | 23 |
| Other | 47 | 47 |
| Bacillibactin | 46 | 47 |
| Other | 41 | 0 |
| SubtilosinA | 21 | 21 |
| Bacilysin | 43 | 43 |
基因组测序技术广泛应用在基因高效、快速的探寻中。王莎等[29]利用全基因组测序技术对内生链霉菌(Streptomyces sp.) SAT1进行了测定,通过分析抗生素的合成途径解释了SAT1具有抗菌活性的原因。本试验利用Illumina和PacBio测序平台对菌株N2-10进行测序分析发现,菌株N2-10基因组大小、GC含量与模式菌株具有高度相似性。
在GO功能注释结果中,较多的基因与分子功能相关和生物过程相关,N2-10菌株所携带的基因参与了众多生物学过程。在COG功能注释结果中,N2-10菌株的基因功能主要集中在氨基酸的转运及代谢、碳水化合物的转运及代谢、无机离子的转运及代谢,这与GO功能注释结果相一致。蛋白质作为构成细胞生命的三大营养物质之一,是构成机体组织器官的基本成分,参与多种重要的生理活动,因此,饲料中必须提供足够的蛋白质才能供动物机体利用[30]。无论是单胃动物还是反刍动物,对于蛋白质的消化吸收都必须先将其分解为氨基酸,才能供机体所利用[31-32]。在KEGG功能注释结果中,与碳水化合物代谢相关的基因数最多,其次是氨基酸代谢,这与上述注释结果相一致。此外,还发现44个基因与萜类和聚酮化合物的代谢相关,可能参与抗菌活性物质的生物合成。例如,srfAA (Gene0413)、srfAB (Gene0414)、srfAC (Gene0415)均为表面活性素的生物合成基因[33];ppsA (Gene2052)、ppsB (Gene2051)、ppsC (Gene2050)、ppsD (Gene2049)等为丰原素的生物合成基因[34]。
碳水化合物在动物生长生产方面具有重要作用。碳水化合物可分为营养性多糖和结构性多糖[35]。结构性多糖主要分为纤维素、木质素等[36]。碳水化合物功能注释结果中显示,菌株N2-10基因组中含有大量与β-葡萄糖苷酶、β-1, 4-木聚糖内切酶、内切葡聚糖酶、乙酰木聚糖酶等相关的基因。β-葡萄糖苷酶、内切葡聚糖酶属于纤维素酶家族,在纤维素分解过程中扮演着重要角色。β-1, 4-木聚糖内切酶属于木聚糖降解酶家族,是木聚糖水解最关键的酶,乙酰木聚糖酶可协同木聚糖酶进行木聚糖的水解[37]。动物饲料中含有较高比例的纤维素等结构性多糖,难以被动物机体消化利用,但可以在微生物的分解作用下被机体吸收[38],菌株N2-10能够合成众多结构性多糖降解酶,将结构性多糖转化为可供动物机体消化利用的营养物质,进一步提高饲料转化率。
通过利用antiSMASH数据库进行基因比对,发现7个已经报道的抗菌活性物质合成基因簇,分别为根霉素、表面活性素、bacillaene、丰原素、儿茶酸铁载体杆菌苷、芽孢杆菌素和溶杆菌素。根霉素A具有广谱抗真菌作用,在致病菌的防治中具有较大的生物潜力[39],bacillaene是由芽孢杆菌属产生的一类多聚烯类抑菌物质。有研究表明,bacillaene可以介导枯草芽孢杆菌影响空肠弯曲菌(Campylobacter jejuni)生物膜的形成,从而抑制此类致病菌[40]。丰原素也是由芽孢杆菌属代谢产生的一种环状脂质肽类物质,具有强大的抗真菌活性,在碱性和酸性等条件下均能展现出较强的抗真菌特性和化学稳定性[41]。儿茶酸铁载体杆菌苷对植物病原菌具有潜在抗菌活性,可以在体内或体外抑制丁香假单胞菌(Pseudomonas syringae)的生长。另有研究发现,儿茶酸铁载体杆菌苷还能够通过上调特定基因介导微生物间的竞争[42]。芽孢杆菌素A对某些革兰氏阳性菌具有较强抗菌活性,研究表明,随着菌株发生突变,代谢产生的芽孢杆菌素A相应出现突变,形成芽孢杆菌素A1,并展现出更强的抑菌能力[43]。溶杆菌素是一类双肽抗菌物质,不同于其他抗菌物质,此类物质可以在除地衣芽孢杆菌(Bacillus licheniformis)外多个菌种中产生,对革兰氏阴性菌具有较强抑菌活性,目前研究证明溶杆菌素在抑制食源性病原菌中发挥主导作用[44]。表面活性素是由芽孢杆菌属产生的一种环状脂肽类表明活性剂,是最有效的表明活性剂之一,由于其具有良好的抗菌和抗病毒活性,在病原菌防治等方面具有突出作用,值得注意的是,研究发现表面活性素有望成为治疗癌症和治理环境的新武器[45]。以上也进一步证明了菌株N2-10基因中含有的7个抗菌活性物质基因簇能够为揭示菌株对多种致病菌具有抑菌作用提供理论依据。
微生物是复杂且多样的,比较基因组学的研究能够帮助人们更清晰地了解微生物物种间的差异,进一步探究菌株基因组基因在进化过程中发生的变化,从而增强其对环境的适应能力[46]。Kwon等[47]通过比较基因组学开发了用于食品检测枯草芽孢杆菌的引物探针,基于此技术研发的引物探针具有较高的灵敏性和选择性。Abdelhamid等[48]的研究表明,双歧杆菌属是生命早期肠道微生物菌群中最丰富的物种之一,与人类健康具有重要联系,通过比较基因组分析,挖掘了双歧杆菌(Bifidobacterium dentium)基因组中存在的碳水化合物降解酶系统,解密了不同部位分离的双歧杆菌的糖酵解能力,强调了比较基因组在揭示双歧杆菌糖酵解能力和糖酵解能力对人类肠道中碳水化合物消化利用的影响。菌株在进化过程中,因环境选择,易造成基因的缺失、获得和变异等[49]。通过比较基因组学对菌株基因组进行比较分析,更有助于加深对菌株进化能力的揭示。
本实验以枯草芽孢杆菌168模式菌株为参考,对比了N2-10与168菌株的基因组结构及基因组功能。结果发现,N2-10与168菌株两基因组编码的氨基酸具有同源性[50]。在基因组组成中,菌株N2-10基因组的编码基因数、基因岛数、前噬菌体数与168菌株存在差异,N2-10菌株可能具有更强的环境适应能力。在碳水化合物酶方面,N2-10菌株较168菌株多了3个乙酰木聚糖酯酶。乙酰木聚糖酯酶在20世纪80年代被发现[51],乙酰木聚糖酯酶分为不同家族,具有不同的底物特异性[52],其作用位点为木糖残基的O-乙酰基[53],木聚糖是构成半纤维素的主要成分,木聚糖主链骨架和侧链的降解需多种酶完成降解,其中乙酰基是阻碍木聚糖降解的限制因素之一,解除乙酰基的限制,可帮助提高木聚糖的有效降解[54]。目前,乙酰木聚糖酯酶的催化机制研究较为透彻。此外,乙酰木聚糖酯酶的酶协同作用也被众多研究者关注,乙酰木聚糖酯酶与阿拉伯呋喃糖苷酶发挥协同作用,能够有效降解木聚糖。乙酰木聚糖酯酶与纤维素酶协同作用能够促进β-1, 4糖苷键的水解效率,进而大幅提高纤维素降解率[55-56]。由此可见,菌株N2-10基因组中较多的乙酰基木聚糖酯酶编码基因在提高饲料碳水化合物消化率方面具有重要作用。此外,菌株N2-10比168菌株增加了β-果糖呋喃糖苷酶编码基因和果糖激酶编码基因。β-果糖呋喃糖苷酶又可称为蔗糖转化酶,能够催化蔗糖转化为葡萄糖和果糖[57]。从本质上讲,β-果糖呋喃糖苷酶是一种水解酶,在低聚乳果糖的生物合成中至关重要,低聚乳果糖是一种功能性三糖,具有低热量等特点,可有效促进肠道双歧杆菌的生长繁殖,抑制肠道病原菌增殖和改善肠道功能等[58]。在本研究中,与168菌株相比,N2-10菌株催化d-葡萄糖转化d-果糖的效率更高。果糖激酶是果糖进一步转化分解过程中重要的酶,可将果糖进一步磷酸化为果糖-6-磷酸。研究表明,果糖激酶是一种双功能酶,不仅具有催化活性,还具有调节功能[59]。目前已经证实果糖激酶参糖代谢过程,特别是果糖必须经过磷酸化后才可以进一步进行代谢;此外,果糖激酶还参与糖感受和信号转导过程[60]。
在抗菌物质合成方面,N2-10菌株含有10个合成基因簇。168菌株具有11个合成基因簇,包括7个已知基因簇和4个未知基因簇。两株菌基因组中,根霉素A、表明活性素、bacillaene、丰原素、儿茶酸铁载体杆菌苷、芽孢杆菌素A、溶杆菌素合成基因簇和3个未知基因簇具有较高相似性。尽管N2-10与168菌株合成抗菌物质的基因簇具有相似性,但N2-10相对168菌株的抑菌能力强弱还需进一步研究。
4 结论本文通过Illumina NovaSeq PE150和PacBio Sequel测序技术对枯草芽孢杆菌N2-10进行了全基因组测序,N2-10基因组大小为4 036 899 bp,GC含量为43.88%;共编码4 163个编码基因;通过分析发现,菌株N2-10基因组含有根霉素A、表明活性素、bacillaene、丰原素、儿茶酸铁载体杆菌苷、芽孢杆菌素A、溶杆菌素等多个次级代谢产物合成基因簇,以及β-葡萄糖苷酶、β-1, 4-木聚糖内切酶、内切葡聚糖酶、几丁质酶、半乳糖苷酶、乙酰木聚糖酶等多种水解酶编码基因,这对进一步解释菌株N2-10具有较强抑菌能力和能够产生多种水解酶提供了宝贵的依据。此外,比较基因组学分析表明,菌株N2-10与模式菌株枯草芽孢杆菌168具有较高相似性,说明该菌在长期的进化中仍然保持高度的水平基因转移。本研究为进一步阐明菌株N2-10的作用机制和进一步开发利用N2-10菌株提供了坚实有力的依据。
| [1] |
KIM HB, BOREWICZ K, WHITE BA, SINGER RS, SREEVATSAN S, TU ZJ, ISAACSON RE. Longitudinal investigation of the age-related bacterial diversity in the feces of commercial pigs[J]. Veterinary Microbiology, 2011, 153(1/2): 124-1330. |
| [2] |
BEGG DP, STEINBRECHER KA, MUL JD, CHAMBERS AP, KOHLI R, HALLER A, COHEN MB, WOODS SC, SEELEY RJ. Effect of guanylate cyclase-C activity on energy and glucose homeostasis[J]. Diabetes, 2014, 63(11): 3798-3804. DOI:10.2337/db14-0160 |
| [3] |
POURABEDIN M, ZHAO X. Prebiotics and gut microbiota in chickens[J]. FEMS Microbiology Letters, 2015, 362(15): fnv122. DOI:10.1093/femsle/fnv122 |
| [4] |
CAI D, RAO Y, ZHAN Y, WANG Q, CHEN S. Engineering Bacillus for efficient production of heterologous protein: current progress, challenge and prospect[J]. Journal of applied microbiology, 2019, 126(6): 1632-1642. DOI:10.1111/jam.14192 |
| [5] |
DU XY, ZHAO K, ZHAO ZM, ZHAO J. Progress on Bacillus subtilis microbial ecological agents by fermentation[J]. Microbiology China, 2020, 47(3): 903-914. (in Chinese) 杜晓雨, 赵恺, 赵志敏, 赵吉. 枯草芽孢杆菌微生态制剂发酵研究进展[J]. 微生物学通报, 2020, 47(3): 903-914. DOI:10.13344/j.microbiol.china.190586 |
| [6] |
SEN S, INGALE SL, KIM YW, KIM JS, KIM KH, LOHAKARE JD, KIM EK, KIM HS, RYU MH, KWON IK, CHAE BJ. Effect of supplementation of Bacillus subtilis LS 1-2 to broiler diets on growth performance, nutrient retention, caecal microbiology and small intestinal morphology[J]. Research in Veterinary Science, 2012, 93(1): 264-268. DOI:10.1016/j.rvsc.2011.05.021 |
| [7] |
LI J. Effects of Bacillus subtilis on growth performance and rumen microflora in Holstein cows[D]. Taiʼan: Masterʼs Thesis of Shandong Agricultural University, 2019 (in Chinese). 李靖. 枯草芽孢杆菌对荷斯坦育成牛生长性能和瘤胃微生物区系的影响[D]. 泰安: 山东农业大学硕士学位论文, 2019. |
| [8] |
CHENG LP. Effect of selenium-enriched yeast and Bacillus subtilis on growth performance, digestion and immune function of hu lambs[D]. Hefei: Masterʼs Thesis of Anhui Agricultural University, 2018 (in Chinese). 程连平. 富硒酵母和枯草芽孢杆菌对湖羊羔羊生长性能、消化和免疫机能的影响[D]. 合肥: 安徽农业大学硕士学位论文, 2018. |
| [9] |
CHEN ZM, ZHENG AJ, CHANG WH, CAI HY, LIU GH. Effects of Lactobacillus and Bacillus subtilis mixed fermentation feed on growth performance, slaughter performance and intestinal morphology of broilers[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(12): 3909-3916. (in Chinese) 陈志敏, 郑爱娟, 常文环, 蔡辉益, 刘国华. 植物乳杆菌和枯草芽孢杆菌混菌发酵饲料对肉鸡生长性能、屠宰性能和肠道结构的影响[J]. 中国畜牧兽医, 2020, 47(12): 3909-3916. DOI:10.16431/j.cnki.1671-7236.2020.12.013 |
| [10] |
WANG Z. Effects of different strains fermented bran on the production performance, antioxidant and fecal microorganism of pigs[D]. Taiʼan: Masterʼs Thesis of Shandong Agricultural University, 2018 (in Chinese). 王震. 不同菌种发酵麸皮对育肥猪生产性能、抗氧化以及粪便微生物的影响[D]. 泰安: 山东农业大学硕士学位论文, 2018. |
| [11] |
ZHU YL. Study on the effects of bran fermentation on performance, nutrient utilization rate and granule production of broilers[D]. Taiʼan: Masterʼs Thesis of Shandong Agricultural University, 2019 (in Chinese). 朱燕丽. 麸皮发酵物对肉鸡生产性能、养分利用率和颗粒制作影响的研究[D]. 泰安: 山东农业大学硕士学位论文, 2019. |
| [12] |
ZHANG YR, ZHANG WT, WANG S, LI SN, GUO WT, WANG SX, LI HY. Evaluation of in vitro probiotic properties of Bacillus subtilis N2-10[J]. Feed Industry, 2020, 41(24): 12-17. (in Chinese) 张亚茹, 张伟涛, 王硕, 李术娜, 郭伟婷, 王树香, 李红亚. 枯草芽孢杆菌N2-10体外益生特性评价[J]. 饲料工业, 2020, 41(24): 12-17. |
| [13] |
GUO W, GUO XJ, ZHOU X, LI SN, ZHU BC. Effect of corn stalk fermented by complex bacterial on rumen bacteria diversity in sheep[J]. Chinese Journal of Animal and Veterinary Sciences, 2018, 49(4): 736-745. (in Chinese) 郭威, 郭晓军, 周贤, 李术娜, 朱宝成. 复合菌剂发酵玉米秸秆对绵羊瘤胃液细菌多样性的影响[J]. 畜牧兽医学报, 2018, 49(4): 736-745. |
| [14] |
WEI YB. Fermentation of compound feed by probiotics and application in laying hens breeding without antibiotic[D]. Baoding: Masterʼs Thesis of Hebei Agricultural University, 2020 (in Chinese). 魏越波. 益生菌发酵配合饲料在蛋鸡无抗养殖上的应用[D]. 保定: 河北农业大学硕士学位论文, 2020. |
| [15] |
REINER J, PISANI L, QIAO WQ, SINGH R, YANG Y, SHI LS, KHAN WA, SEBRA R, COHEN N, BABU A, EDELMANN L, JABS EW, SCOTT SA. Cytogenomic identification and long-read single molecule real-time (SMRT) sequencing of a Bardet–Biedl Syndrome 9 (BBS9) deletion[J]. Npj Genomic Medicine, 2018, 3: 3. DOI:10.1038/s41525-017-0042-3 |
| [16] |
DELCHER AL, BRATKE KA, POWERS EC, SALZBERG SL. Identifying bacterial genes and endosymbiont DNA with Glimmer[J]. Bioinformatics, 2007, 23(6): 673-679. DOI:10.1093/bioinformatics/btm009 |
| [17] |
BESEMER J, BORODOVSKY M. GeneMark: web software for gene finding in prokaryotes, eukaryotes and viruses[J]. Nucleic Acids Research, 2005, 33(suppl_2): W451-W454. |
| [18] |
LOWE TM, EDDY SR. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence[J]. Nucleic Acids Research, 1997, 25(5): 955-964. DOI:10.1093/nar/25.5.955 |
| [19] |
LAGESEN K, HALLIN P, RØDLAND EA, STAERFELDT HH, ROGNES T, USSERY DW. RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Research, 2007, 35(9): 3100-3108. DOI:10.1093/nar/gkm160 |
| [20] |
GARDNER PP, DAUB J, TATE JG, NAWROCKI EP, KOLBE DL, LINDGREEN S, WILKINSON AC, FINN RD, GRIFFITHS-JONES S, EDDY SR, BATEMAN A. Rfam: updates to the RNA families database[J]. Nucleic Acids Research, 2008, 37(suppl_1): D136-D140. |
| [21] |
BLAND C, RAMSEY TL, SABREE F, LOWE M, BROWN K, KYRPIDES NC, HUGENHOLTZ P. CRISPR recognition tool (CRT): a tool for automatic detection of clustered regularly interspaced palindromic repeats[J]. BMC Bioinformatics, 2007, 8: 209. DOI:10.1186/1471-2105-8-209 |
| [22] |
ZHOU Y, LIANG YJ, LYNCH KH, DENNIS JJ, WISHART DS. PHAST: a fast phage search tool[J]. Nucleic Acids Research, 2011, 39(suppl_2): W347-W352. |
| [23] |
BERTELLI C, LAIRD MR, WILLIAMS KP, SIMON Fraser University Research Computing Group, LAU BY, HOAD G, WINSOR GL, BRINKMAN FS. IslandViewer 4: expanded prediction of genomic islands for larger-scale datasets[J]. Nucleic Acids Research, 2017, 45(W1): W30-W35. DOI:10.1093/nar/gkx343 |
| [24] |
ASHBURNER M, BALL CA, BLAKE JA, BOTSTEIN D, BUTLER H, CHERRY JM, DAVIS AP, DOLINSKI K, DWIGHT SS, EPPIG JT, HARRIS MA, HILL DP, ISSEL-TARVER L, KASARSKIS A, LEWIS S, MATESE JC, RICHARDSON JE, RINGWALD M, RUBIN GM, SHERLOCK G. Gene Ontology: tool for the unification of biology[J]. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556 |
| [25] |
JENSEN LJ, JULIEN P, KUHN M, von MERING C, MULLER J, DOERKS T, BORK P. eggNOG: automated construction and annotation of orthologous groups of genes[J]. Nucleic Acids Research, 2007, 36(suppl_1): D250-D254. |
| [26] |
KANEHISA M, GOTO S. KEGG: Kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27 |
| [27] |
LOMBARD V, GOLACONDA RAMULU H, DRULA E, COUTINHO PM, HENRISSAT B. The carbohydrate- active enzymes database (CAZy) in 2013[J]. Nucleic Acids Research, 2013, 42(D1): D490-D495. |
| [28] |
BLIN K, WOLF T, CHEVRETTE MG, LU XW, SCHWALEN CJ, KAUTSAR SA, SUAREZ DURAN HG, de los SANTOS ELC, KIM HU, NAVE M, DICKSCHAT JS, MITCHELL DA, SHELEST E, BREITLING R, TAKANO E, LEE SY, WEBER T, MEDEMA MH. antiSMASH 4.0—improvements in chemistry prediction and gene cluster boundary identification[J]. Nucleic Acids Research, 2017, 45(W1): W36-W41. DOI:10.1093/nar/gkx319 |
| [29] |
WANG S, DOU GM, MA YC. Genome sequencing and comparative genome analysis of Streptomyces sp. SAT1[J]. Microbiology China, 2021, 48(9): 3039-3053. (in Chinese) 王莎, 窦桂铭, 马玉超. 植物内生链霉菌Streptomyces sp. SAT1的基因组测序和比较基因组分析[J]. 微生物学通报, 2021, 48(9): 3039-3053. |
| [30] |
LI LY. The regulatory mechanisms of mitochondrial fatty acid β-oxidation in energy homeostasis maintenance of fish[D]. Shanghai: Doctoral Dissertation of East China Normal University, 2020 (in Chinese). 李玲玉. 线粒体脂肪酸β-氧化对鱼类能量代谢稳态的维持及调控机制研究[D]. 上海: 华东师范大学博士学位论文, 2020. |
| [31] |
ZONG XY, ZHANG HW, SHI HP. Advance in research on digestive physiology and gastrointestinal microflora of lambs[J]. Animal Husbandry & Veterinary Medicine, 2022, 54(4): 124-129. (in Chinese) 宗学阳, 张华文, 史怀平. 羔羊消化生理及消化道微生物研究进展[J]. 畜牧与兽医, 2022, 54(4): 124-129. |
| [32] |
WU SJ, BHAT ZF, GOUNDER RS, MOHAMED AHMED IA, AL-JUHAIMI FY, DING Y, BEKHIT AEDA. Effect of dietary protein and processing on gut microbiota-a systematic review[J]. Nutrients, 2022, 14(3): 453. DOI:10.3390/nu14030453 |
| [33] |
RAHMAN FB, SARKAR B, MONI RP, RAHMAN MS. Molecular genetics of surfactin and its effects on different sub-populations of Bacillus subtilis[J]. Biotechnology Reports, 2021, 32: e00686. DOI:10.1016/j.btre.2021.e00686 |
| [34] |
STELLER S, VOLLENBROICH D, LEENDERS F, STEIN T, CONRAD B, HOFEMEISTER J, JACQUES P, THONART P, VATER J. Structural and functional organization of the fengycin synthetase multienzyme system from Bacillus subtilis b213 and A1/3[J]. Chemistry & Biology, 1999, 6(1): 31-41. |
| [35] |
DASHTY M. A quick look at biochemistry: carbohydrate metabolism[J]. Clinical Biochemistry, 2013, 46(15): 1339-1352. DOI:10.1016/j.clinbiochem.2013.04.027 |
| [36] |
MENG M, CHENG D, HAN LR, CHEN YY, WANG CL. Isolation, purification, structural analysis and immunostimulatory activity of water-soluble polysaccharides from Grifola frondosa fruiting body[J]. Carbohydrate Polymers, 2017, 157: 1134-1143. DOI:10.1016/j.carbpol.2016.10.082 |
| [37] |
ZHU TD, HE Y, YANG B, LIU JC, WU MC. Combinative degradation of xylan with acetyl xylan esterase and xylanase[J]. Journal of Food Science and Biotechnology, 2016, 35(4): 375-380. (in Chinese) 朱天地, 何瑶, 杨标, 刘加驰, 邬敏辰. 乙酰木聚糖酯酶协同木聚糖酶降解木聚糖的研究[J]. 食品与生物技术学报, 2016, 35(4): 375-380. DOI:10.3969/j.issn.1673-1689.2016.04.007 |
| [38] |
MOITA VHC, DUARTE ME, KIM SW. Functional roles of xylanase enhancing intestinal health and growth performance of nursery pigs by reducing the digesta viscosity and modulating the mucosa-associated microbiota in the jejunum[J]. Journal of Animal Science, 2022, 100(5): skac116. DOI:10.1093/jas/skac116 |
| [39] |
VILLA-RODRIGUEZ E, MORENO-ULLOA A, CASTRO-LONGORIA E, PARRA-COTA FI, de LOS SANTOS-VILLALOBOS S. Integrated omics approaches for deciphering antifungal metabolites produced by a novel Bacillus species, B. cabrialesii TE3T, against the spot blotch disease of wheat (Triticum turgidum L. subsp. durum)[J]. Microbiological Research, 2021, 251: 126826. DOI:10.1016/j.micres.2021.126826 |
| [40] |
EREGA A, STEFANIC P, DOGSA I, DANEVČIČ T, SIMUNOVIC K, KLANČNIK A, SMOLE MOŽI NA S, MANDIC MULEC I. Bacillaene mediates the inhibitory effect of Bacillus subtilis on Campylobacter jejuni biofilms[J]. Applied and Environmental Microbiology, 2021, 87(12): e0295520. DOI:10.1128/AEM.02955-20 |
| [41] |
GIMENEZ D, PHELAN A, MURPHY CD, COBB SL. Fengycin A analogues with enhanced chemical stability and antifungal properties[J]. Organic Letters, 2021, 23(12): 4672-4676. DOI:10.1021/acs.orglett.1c01387 |
| [42] |
DIMOPOULOU A, THEOLOGIDIS I, BENAKI D, KOUKOUNIA M, ZERVAKOU A, TZIMA A, DIALLINAS G, HATZINIKOLAOU DG, SKANDALIS N. Direct antibiotic activity of bacillibactin broadens the biocontrol range of Bacillus amyloliquefaciens MBI600[J]. mSphere, 2021, 6(4): e0037621. DOI:10.1128/mSphere.00376-21 |
| [43] |
HUANG T, GENG H, MIYYAPURAM VR, SIT CS, VEDERAS JC, NAKANO MM. Isolation of a variant of subtilosin A with hemolytic activity[J]. Journal of Bacteriology, 2009, 191(18): 5690-5696. DOI:10.1128/JB.00541-09 |
| [44] |
NANNAN C, VU HQ, GILLIS A, CAULIER S, NGUYEN TTT, MAHILLON J. Bacilysin within the Bacillus subtilis group: gene prevalence versus antagonistic activity against Gram-negative foodborne pathogens[J]. Journal of Biotechnology, 2021, 327: 28-35. DOI:10.1016/j.jbiotec.2020.12.017 |
| [45] |
SANTOS VSV, SILVEIRA E, PEREIRA BB. Toxicity and applications of surfactin for health and environmental biotechnology[J]. Journal of Toxicology and Environmental Health, Part B, 2018, 21(6/7/8): 382-399. |
| [46] |
ZHANG YZ, JALAN N, ZHOU XF, GOSS E, JONES JB, SETUBAL JC, DENG XL, WANG N. Positive selection is the main driving force for evolution of citrus canker-causing Xanthomonas[J]. The ISME Journal, 2015, 9(10): 2128-2138. DOI:10.1038/ismej.2015.15 |
| [47] |
KWON EA, LEE JI, PARK JW, KIM SS. Application of comparative genomics in the development of DNA probes to detect Bacillus cereus and Bacillus subtilis[J]. LWT, 2021, 142: 110996. DOI:10.1016/j.lwt.2021.110996 |
| [48] |
ABDELHAMID AG, EL-DOUGDOUG NK. Comparative genomics of the gut commensal Bifidobacterium bifidum reveals adaptation to carbohydrate utilization[J]. Biochemical and Biophysical Research Communications, 2021, 547: 155-161. DOI:10.1016/j.bbrc.2021.02.046 |
| [49] |
WANG GL, WANG MY, LIU LX, MA K, YANG XP. Progress of adaptive laboratory evolution for industrial strain breeding[J]. Microbiology China, 2022, 49(1): 306-322. (in Chinese) 王光路, 王梦园, 刘兰茜, 马科, 杨雪鹏. 适应性实验室进化在工业生产菌株选育中应用的进展[J]. 微生物学通报, 2022, 49(1): 306-322. |
| [50] |
CABAL A, JUN SR, JENJAROENPUN P, WANCHAI V, NOOKAEW I, WONGSURAWAT T, BURGESS MJ, KOTHARI A, WASSENAAR TM, USSERY DW. Genome-based comparison of Clostridioides difficile: average amino acid identity analysis of core genomes[J]. Microbial Ecology, 2018, 76(3): 801-813. DOI:10.1007/s00248-018-1155-7 |
| [51] |
BIELY P. Microbial xylanolytic systems[J]. Trends in Biotechnology, 1985, 3(11): 286-290. DOI:10.1016/0167-7799(85)90004-6 |
| [52] |
UHLIARIKOVÁ I, VRŠANSKÁ M, MCCLEARY BV, BIELY P. Positional specifity of acetylxylan esterases on natural polysaccharide: an NMR study[J]. Biochimica et Biophysica Acta: BBA - General Subjects, 2013, 1830(6): 3365-3372. DOI:10.1016/j.bbagen.2013.01.011 |
| [53] |
POUTANEN K, SUNDBERG M, KORTE H, PULS J. Deacetylation of xylans by acetyl esterases of Trichoderma reesei[J]. Applied Microbiology and Biotechnology, 1990, 33(5): 506-510. |
| [54] |
WANG JQ, ZHANG HM, WU MC, TANG CD. Cloning and sequence analysis of a novel xylanase gene, Auxyn10A, from Aspergillus usamii[J]. Biotechnology Letters, 2011, 33(5): 1029-1038. DOI:10.1007/s10529-011-0524-9 |
| [55] |
MORIYOSHI K, KOMA D, YAMANAKA H, SAKAI K, OHMOTO T. Expression and characterization of a thermostable acetylxylan esterase from Caldanaerobacter subterraneus subsp. tengcongensis involved in the degradation of insoluble cellulose acetate[J]. Bioscience, Biotechnology, and Biochemistry, 2013, 77(12): 2495-2498. DOI:10.1271/bbb.130568 |
| [56] |
RAWEESRI P, RIANGRUNGROJANA P, PINPHANICHAKARN P. Α-l-arabinofuranosidase from Streptomyces sp. PC22: purification, characterization and its synergistic action with xylanolytic enzymes in the degradation of xylan and agricultural residues[J]. Bioresource Technology, 2008, 99(18): 8981-8986. DOI:10.1016/j.biortech.2008.05.016 |
| [57] |
HUANG HW, LIU YL, LI C. Progress in glycosidase and modification of strategies[J]. Biotechnology Bulletin, 2010(5): 55-60. (in Chinese) 黄红卫, 刘艳丽, 李春. 糖苷酶的研究及其改造策略[J]. 生物技术通报, 2010(5): 55-60. |
| [58] |
DING WL, ZHANG QM, MA W, WU B, HE BF. Immobilization of β-fructofuranosidase for the synthesis of lactosucrose[J]. Microbiology China, 2016, 43(1): 9-16. (in Chinese) 丁为龙, 张庆明, 马旺, 吴斌, 何冰芳. β-呋喃果糖苷酶的固定化及其在低聚乳果糖合成中的应用[J]. 微生物学通报, 2016, 43(1): 9-16. |
| [59] |
LORETI E, de BELLIS L, ALPI A, PERATA P. Why and how do plant cells sense sugars?[J]. Annals of Botany, 2001, 88(5): 803-812. DOI:10.1006/anbo.2001.1526 |
| [60] |
LEÓN P, SHEEN J. Sugar and hormone connections[J]. Trends in Plant Science, 2003, 8(3): 110-116. DOI:10.1016/S1360-1385(03)00011-6 |
 2023, Vol. 50
2023, Vol. 50




