扩展功能
文章信息
- 陈潇潇, 曹雪松, 吴楠, 李喜梅, 王丽艳, 荆瑞勇
- CHEN Xiaoxiao, CAO Xuesong, WU Nan, LI Ximei, WANG Liyan, JING Ruiyong
- 两株聚球藻伴生细菌分离纯化与促生功能鉴定
- Isolation and growth-promoting function identification of two associated bacterial strains of Synechococcus sp.
- 微生物学通报, 2023, 50(5): 1826-1839
- Microbiology China, 2023, 50(5): 1826-1839
- DOI: 10.13344/j.microbiol.china.220621
-
文章历史
- 收稿日期: 2022-07-02
- 接受日期: 2022-11-19
- 网络首发日期: 2023-01-02
2. 农业农村部东北平原农业绿色低碳重点实验室, 黑龙江 大庆 163319
2. Key Laboratory of Low-carbon Green Agriculture in Northeast Plain, Ministry of Agriculture and Rural Affairs, Daqing 163319, Heilongjiang, China
聚球藻是地球上数量最丰富、分布最广泛的蓝藻之一,其适应性强、多样性丰富,广泛存在于淡水、河口、海岸、海洋水域环境,在海洋环境中约有7.0×1026个聚球藻细胞,它们提供了近海洋净初级生产量的16.7%[1],对于河口和海洋环境的聚球藻已有大量报道[2-4],但有关淡水生态系统中聚球藻的研究较少。目前大多数分离的聚球菌菌株均保持为非无菌培养状态[1],藻细胞向周围分泌代谢产物形成一种独特的微环境,称为“藻际环境” (phycosphere)[5],蓝藻藻际异养细菌由自由漂浮的游离细菌和在蓝藻上附着的伴生细菌组成[6],附着在蓝藻上的细菌称为伴生细菌[7]。
伴生细菌跟随蓝藻传代培养,长期共存,两者之间存在一定的平衡关系[8],Christie-Oleza等和Kaur等[9-11]从蛋白质组学的角度对聚球藻和伴生细菌之间的关系进行研究,发现在长期培养过程中添加了异养细菌的聚球藻培养体系相较于无菌的聚球藻培养体系维持得更久;并且Hayashi等[12]发现,Synechococcus leopoliensis CCAP1405/1与伴生细菌共同培养时可在普通的固体琼脂营养基平板上生长,而无菌时仅能在该营养条件的液体培养基中生长,说明伴生细菌对聚球藻不可或缺,能够促进其生长。目前,对于伴生细菌的研究大多利用高通量测序明确其种属,对分离株研究较少[13]。
藻菌之间的比例是影响蓝藻生长的一个重要因素,细菌将蓝藻产生的难降解的有机大分子物质分解为水、二氧化碳、铵盐、硝酸盐和磷酸盐等,为蓝藻提供无机盐[14-15];细菌还可以分泌蓝藻自身无法合成的生长因子,如吲哚乙酸(indole-3-acetic acid, IAA)[16-17]、B12[18-19]、铁载体[14],适宜浓度IAA对大多数蓝藻均有促生长作用,因此适宜藻菌比例可促进蓝藻的生长[20-21],明确促进蓝藻的最适藻菌比例关系,对深入理解藻菌关系更具意义,有利于蓝藻生物量的增加[21],但伴生细菌对蓝藻以及其他作物发芽是否均具有促生作用鲜有报道。
本研究基于对淡水聚球藻-伴生细菌相互作用机制的探索,对伴生细菌群落多样性进行研究并分离纯化伴生细菌,探索伴生细菌对淡水聚球藻生长的影响,并利用水稻试验发芽试验探索伴生细菌脱离藻际环境是否产生影响,以期为研究淡水蓝藻与其伴生细菌相互关系奠定基础。
1 材料与方法 1.1 材料 1.1.1 供试藻种聚球藻(Synechococcus) 7942购买于中国科学院淡水藻种库(Freshwater Algae Culture Collection at the Institute of Hydrobiology, FACHB),培养基为BG-11培养基,在25 ℃、光照强度为25 μE/(m2·s)、光周期为L: D=14:10的条件下培养。
1.1.2 供试培养基BG-11培养基[7]、LB培养基[22]和NA培养基[23]。
1.1.3 主要试剂与仪器TIANgel Midi Purification Kit,天根生化科技(北京)有限公司;聚碳酸酯滤膜,Whatman公司。恒温培养箱,上海跃进医疗器械有限公司;PCR仪,Biometra公司;光学显微镜,奥兰巴斯公司;分光光度仪,上海元析仪器有限公司。
1.2 方法 1.2.1 聚球藻伴生细菌的生物多样性将购买的聚球藻扩大培养,在无菌条件下,取200 mL活化藻用0.4 μm微孔滤膜过滤除去游离细菌,再用无菌BG-11液体培养基将滤膜上藻菌混合物冲洗,再过0.4 μm微孔滤膜,连续过膜3次,将藻菌混合物收集到0.4 μm滤膜上。所得滤膜分别转移至3个2 mL无菌离心管,运用CTAB法[24]分别提取DNA作为模板,采用引物515F (5′-GTGCCAGCMGCCGCGG-3′)/907R (5′-CCGTCAATTCMTTTRAGTTT-3′)对细菌16S rRNA基因的V4–V5区进行PCR扩增[25],PCR产物进行Illumina高通量测序,测序获得序列经优化后划分OTU,剔除聚球藻序列后在属水平统计伴生细菌群落组成。高通量测序原始数据已提交至国家微生物科学数据中心(https://nmdc.cn/),登录号为NMDC40027515– NMDC40027517。
1.2.2 伴生细菌的分离纯化取1 mL聚球藻藻液装入盛有5 mL NA液体培养基的10 mL离心管中,置于恒温摇床富集培养,30 ℃、160 r/min黑暗培养3 d后培养液变混浊,取1 mL浑浊培养液加入盛有5 mL NA液体培养基的10 mL离心管中,继续富集培养。连续富集2次后进行分离和纯化。取富集液100 μL涂布于NA固体培养基,挑取形态不同的单菌落进行划线纯化培养,连续纯化3次,纯化的单菌落进行革兰氏染色,显微镜观察。根据张东艳等[22]方法测定菌株产IAA能力。
1.2.3 菌株的16S rRNA基因鉴定伴生细菌菌液采用引物27F (5′-AGAGTTT GATCCTGGCTCAG-3′)和1492R (5′-GGTTAC CTTGTTACGACTT-3′)扩增16S rRNA基因。PCR反应体系(50 μL):去离子水23 μL,上、下游引物(50 pmol/L)各0.5 μL,2×Taq PCR PreMix 25 μL,DNA模板1 μL;阴性对照包含除模板外的所有成分。PCR反应条件:94 ℃ 5 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 1 min,30个循环;72 ℃ 10 min。PCR产物进行琼脂糖凝胶电泳,从1.5%琼脂糖凝胶上切取约1 500 bp大小的PCR产物,用TIANgel Midi Purification Kit进行胶回收,纯化DNA片段采用pEASY-T1质粒进行TA连接,质粒转化导入大肠杆菌DH5α感受态细胞。每个转化平板中选出20个白色克隆,用M13-F (5′-TGTAAA ACGACGGCCAGT-3′)和M13-R (5′-CAGGAA ACAGCTATGACC-3′)引物进行PCR扩增,检测阳性克隆。PCR体系和循环数分别为10 μL和25其他同上。每个菌株选取5个阳性克隆菌液进行测序。测序获得的序列与NCBI和EzBioCloud网站(https://www.ezbiocloud.net/)的16S rRNA基因数据库中模式菌株基因序列进行BLAST比对,并与高通量测序菌群中同属的OTU序列进行比较分析(https://www.ebi.ac.uk/Tools/msa/muscle/),相似性在99.0%以上认为分离菌株为聚球藻伴生细菌。
1.2.4 藻菌不同比例对聚球藻生长速率的影响根据陈宇炜等[26]实验方法,利用叶绿素a的含量估算蓝藻生物量,将分离纯化的两种细菌分别接种至对数生长期(培养7 d)蓝藻菌液中,聚球藻初始菌浓为1×106 CFU/mL,选取菌藻比例为5:1、10:1和15:1,即JQ1和JQ2初始菌浓度均达到5×106、1×107和1.5×107 CFU/mL。每隔24 h取2 mL聚球藻藻液样品,冷冻12 h后迅速加入2 mL 90%的热乙醇(80 ℃)于热水浴中萃取2 min,超声振荡(800 W, 40 kHz)处理10 min,黑暗静置萃取2−6 h后,4 000 r/min离心10 min取清液定容至10 mL,在波长665 nm和750 nm处测吸光值,分别记为E665和E750。滴l mol/L盐酸酸化,于相同波长下再次测量,分别记为A665和A750,通过公式(1)计算聚球藻样品中叶绿素a浓度。
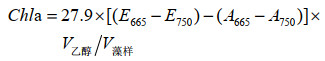
|
(1) |
式(1)中:Chla为叶绿素a含量(μg/mL);V乙醇为乙醇萃取液的体积(mL);V藻样为藻样的体积(mL)。
比生长率(μ, d–1)由叶绿素a的浓度和培养时间通过公式(2)确定。
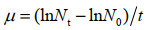
|
(2) |
式(2)中:N0为初始日叶绿素a浓度;Nt为第t天测得的叶绿素a浓度;t为培养时间。
以培养时间为横坐标、比生长率为纵坐标绘制生长曲线,以明确伴生细菌对聚球藻生长的影响。
1.2.5 伴生细菌对水稻种子发芽促生功能研究供试水稻品种为龙粳3040,水稻种子于40 ℃干燥48 h解除休眠,分别浸泡75%酒精和0.1%升汞进行消毒,消毒后种子接种组培瓶中,30粒/瓶。试验分为3组:JQ1菌液、JQ2菌液和NA培养基对照组,菌液终浓度分别为1×105 CFU/mL和5×106 CFU/mL,每组3次重复。在30 ℃暗处理48 h后,置于25 ℃恒温光照培养箱培养,每天统计发芽量,计算发芽势、发芽率、发芽指数和种子萌发指数。

|
式中:Dt为发芽天数;Gt为与Dt相对应的每天发芽种子数;∑为总和;GI值与活力成正相关;nd2、nd4、nd6、nd8分别为第2、第4、第6、第8天的种子萌发率。
以放入恒温光照培养箱记为第0天,到第14天剪下幼苗的地上部分及地下部分,统计其株高、地上部分干重、地下部分干重等秧苗素质指标和根长度、根体积、根表面积等根系参数。
1.3 数据处理将获得伴生细菌克隆测序获得的16S rRNA基因序列在NCBI和EzBioCloud网站上进行同源性比对,下载亲缘性最高的序列后利用ClustalX 1.81进行多序列比对,采用MEGA 7.0软件neighbor-joining法构建系统发育树,明确其种属关系;利用万深LA-S根系分析系统进行水稻根部扫描分析;利用SPSS 26.0软件对获得的叶绿素a含量数据和水稻发芽数据进行显著性分析;运用OriginPro2021及Excel 2016作图。
2 结果与分析 2.1 聚球藻伴生细菌的生物多样性聚球藻伴生细菌DNA样品,3次重复共获得高质量基因序列110 193条,平均36 731条/样品,基于OTU划分,剔除聚球藻后,伴生菌共检出34属37种细菌,隶属于6个门,分别为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidota)、放线菌门(Actinobacteriota)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteriota)和浮霉菌门(Planctomycetota),依次占59.74%、39.96%、0.05%、0.22%、0.02%和0.004%。在属水平上优势菌属分别为NS11-12 marine group_norank、产卟啉杆菌属(Porphyrobacter)、根瘤菌属(Rhizobium)、水单胞菌属(Aquimonas)和中慢生根瘤菌属(Mesorhizobium),占比分别为38.99%、22.28%、15.89%、8.19%和7.15% (图 1)。

|
| 图 1 属水平上聚球藻伴生细菌16S rRNA基因序列的相对丰度 Figure 1 Relative abundance of 16S rRNA gene sequence of associated bacteria with Synechococcus at genus level. Norank:分类学系谱中无这个层级的科学名称;Others:合并了丰度低于0.1%的菌属. Ⅰ、Ⅱ和Ⅲ代表 3次重复 Norank: No scientific name of this level in the taxonomic genealogy; Others: A bacteria genus with a abundance of less than 0.1%. Ⅰ, Ⅱ and Ⅲ indicate three replicates. |
|
|
利用NA培养基富集的聚球藻伴生细菌,平板划线纯化出两株细菌JQ1和JQ2。JQ1菌落呈现白色不透明圆形,表面光滑无褶皱,边缘不规则呈锯齿状,较为湿润;JQ2菌落白色不透明圆形,表面光滑边缘平整,微凸起且较为湿润。革兰氏染色发现菌株JQ1为G–,菌株JQ2为G+,均为杆菌(图 2)。产IAA含量分别为49.69 mg/L和29.91 mg/L。

|
| 图 2 JQ1和JQ2的菌落形态及显微照片 Figure 2 Colony pictures and micrograph of JQ1 and JQ2. A:JQ1菌落图. B:JQ2菌落图. C:JQ1显微图. D:JQ2显微图 A: Colony picture of JQ1. B: Colony picture of JQ2. C: Micrograph of JQ1. D: Micrograph of JQ2. |
|
|
聚球藻伴生细菌的16S rRNA基因序列经测序已获得并已登录至GenBank,菌株JQ1和JQ2的登录号分别为OP404838和OP404364。BLAST序列比对发现,与JQ1相似度最高(99.24%)的模式菌株为Rhizobium rosettiformans (CP032405),与JQ2相似度最高(99.93%)的模式菌株为Peribacillus frigoritolerans (MF800956)。为进一步明确其进化关系,采用邻接法构建系统发育树(图 3),初步确定菌株JQ1属于Rhizobium,JQ2属于Peribacillus,菌株重新命为Rhizobium sp. JQ1和Peribacillus sp. JQ2。与高通量测序菌群中同属的OTU序列进行比较分析,发现菌株JQ1和JQ2分别与OTU5和OTU24的相似度为100%,证明菌株JQ1和JQ2均为聚球藻伴生细菌。

|
| 图 3 采用邻接法构建JQ1和JQ2的系统发育树 Figure 3 Neighbor-joining phylogenetic tree was constructed with JQ1, JQ2 and closest relatives. 分支点上数字代表bootstrap值,bootstrap值< 50未显示;括号中的序号代表相应菌株在NCBI上的核酸序列登录号;标尺代表每个核苷酸替代物残基的数量 The numbers of branching points in phylogenetic tree indicate bootstrap value, bootstrap values < 50 are not shown; Serial number in bracket indicates nucleic acid sequence landing number of corresponding strain in NCBI website; The scale bar represents the number of nucleotide substitutions per residue. |
|
|
不同菌藻比对聚球藻叶绿素a含量和比生长率随时间变化见图 4,菌株JQ1与聚球藻浓度比例为5:1时,其对聚球藻具有明显的促进作用,第4天促进效果最明显(图 4A、4B),叶绿素a含量比对照组增加了121.21% (P < 0.05),比生长率[(0.624 0±0.01) d−1]比对照组显著增加了32.91% (P < 0.05);当比例为10:1时,与对照组的叶绿素a和比生长率无显著差异(图 4C、4D);当比例为15:1时,JQ1对聚球藻具有明显的抑制作用,第3天时抑制效果最明显(图 4E、4F),叶绿素a含量比对照组显著减少了55.56% (P < 0.05),比生长率[(0.094 9±0.05) d−1]比对照组显著降低了73.81% (P < 0.05)。
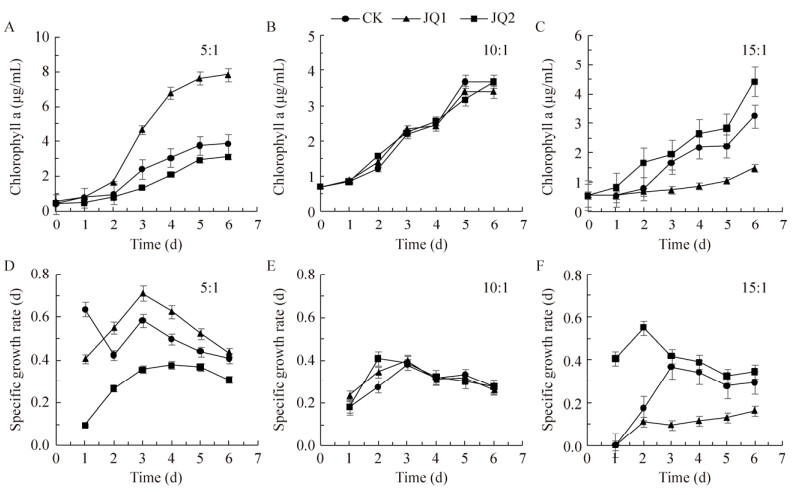
|
| 图 4 不同菌藻比对聚球藻叶绿素a和比生长率的影响 Figure 4 Effect of different ratios of bacteria to Synechococcus on Chlorophyll a and specific growth rate of Synechococcus. 图中5:1、10:1和15:1代表菌藻比 5:1, 10:1 and 15:1 in the figures indicate the ratio of bacteria and Synechococcus. |
|
|
JQ2与聚球藻浓度比例为5:1时,JQ2对聚球藻具有明显的抑制作用,第3天抑制效果最明显(图 4A、4B),叶绿素a含量比对照组显著降低了44.23% (P < 0.05),比生长率[(0.358 5±0.07) d−1]比对照组显著降低了35.78% (P < 0.05);比例为10:1时,叶绿素a含量和比生长率与对照无显著差异(图 4C、4D);比例为15:1时,JQ2对聚球藻具有促进作用,第2天时促进效果最明显(图 4E、4F),叶绿素a含量比对照组显著增加了111.76% (P < 0.05),比生长率[(0.549 3± 0.01) d−1]比对照组增加了215.42% (P < 0.05)。综上,JQ1与聚球藻的浓度比例为5:1时,对聚球藻具有促生效果;当JQ2与聚球藻的浓度比例为15:1时,对聚球藻具有促生效果;菌藻比为10:1时,JQ1和JQ2对聚球藻无促生效果。
2.5 发芽试验验证伴生细菌促生功能通过水稻发芽试验的相关指标验证两株伴生细菌的促生功能,结果表明JQ1能够显著增加水稻种子发芽率、发芽势、发芽指数和种子萌发指数,较对照组分别提高22.82%、18.30%、40.60%和24.40% (表 1);JQ2能显著促进水稻的发芽率,较对照组提高19.71%,发芽势、发芽指数及种子萌发指数也具有增加趋势(表 1)。结果表明JQ1和JQ2对水稻种子发芽都具有促生功能。
| 处理 Treatment |
发芽势 Germination potential (%) |
发芽率 Germination rate (%) |
发芽指数 Germination index |
种子萌发指数 Seed germination index |
| JQ1 | 77.78±0.01a | 93.33±0.02a | 62.89±3.62a | 56.08±2.29a |
| JQ2 | 72.22±0.04ab | 94.44±0.01a | 49.87±2.50b | 52.83±2.96ab |
| CK | 63.33±0.03b | 78.89±0.04b | 44.73±3.91b | 45.08±3.66b |
| 同列数据后不同小写字母表示不同处理间差异显著(P < 0.05). 下同 Different lowercase letters after the data within a column indicate significant differences (P < 0.05) among different treatments. The same below. |
||||
水稻秧苗素质指标见表 2,发现JQ1处理组株高显著高于JQ2处理组,但两者与CK组均无显著差异,JQ1和JQ2处理组的地下干重比CK组都显著增加26.67%,对地上鲜重、地上干重和地下鲜重的影响无显著性差异。可见,聚球藻伴生细菌JQ1与JQ2都对水稻根系干重具有促生作用。
| 处理 Treatment |
株高 Plant height (cm) |
地上鲜重 Aboveground fresh weight (g) |
地上干重 Aboveground dry weight (g) |
地下鲜重 Underground fresh weight (g) |
地下干重 Underground dry weight (g) |
| JQ1 | 7.89±0.43a | 0.095±0.003a | 0.013±0.003a | 0.201±0.003a | 0.019±0.005a |
| JQ2 | 4.62±0.39b | 0.090±0.002a | 0.035±0.025a | 0.194±0.006a | 0.019±0.004a |
| CK | 5.42±1.16ab | 0.083±0.002a | 0.012±0.001a | 0.213±0.008a | 0.015±0.001b |
水稻根系参数见表 3,JQ1较CK组的根表面积、根节点数和分叉数分别显著提高了56.87%、67.40%和83.88%,而根长度、根体积、平均直径、根尖数则无显著差异。JQ2较CK组的根表面积显著提高了66.46%,对于其他指标则与CK组无显著差异。可见,JQ1比JQ2更能显著提高水稻根系的生长,改善水稻秧苗素质。
| 处理 Treatment |
根长度 Root length (cm) |
表面积 Surface area (cm2) |
体积 Volume (cm3) |
平均直径 Average diameter (mm) |
节点数 Number of nodes |
根尖数 Apical number |
分叉数 Bifurcation number |
| JQ1 | 196.57±7.49a | 176.54±22.03a | 36.37±7.75a | 1.04±0.07a | 534±44.47a | 258.67±18.91a | 224.33±23.10a |
| JQ2 | 197.80±5.45a | 187.33±9.80a | 25.35±3.26a | 0.98±0.07a | 335±19.09b | 149.00±10.97a | 146.33±10.90b |
| CK | 177.89±15.74a | 112.54±12.14b | 43.90±22.11a | 0.81±0.05a | 319±28.31b | 161.67±19.13a | 122.00±9.87b |
藻际微生物主要包括细菌、放线菌、真菌、原生动物和噬藻体。目前已报道的藻类伴生细菌有假单胞菌属(Pseudomonas)、鞘氨醇单胞菌属(Sphingopyxis)、鞘脂单胞菌属(Sphingomonas)、黄杆菌属(Flavobacterium)、交替单胞菌属(Alteromonas)、玫瑰杆菌属(Roseobacter)、芽孢杆菌属(Bacillus)、枝芽孢杆菌属(Virgibacillus)、链霉菌属(Streptomyces)、气微菌属(Aeromicrobium)、微杆菌属(Microbacterium)、产卟啉杆菌属(Porphyrobacter)、柠檬球菌属(Citrococcus)和根瘤菌属(Rhizobium)[1, 4, 23, 27-28]。有关聚球藻伴生细菌的报道主要是海洋聚球藻,利用宏基因组学研究表明海洋聚球藻伴生细菌主要隶属于6个属:红杆菌属(Rhodobacter)、交替单胞菌属(Alteromonas)、黄杆菌属(Flavobacterium)、玫瑰杆菌属(Roseobacter)、假单胞菌属(Pseudomonas)及鞘脂单胞菌属(Sphingomonas)[8]。基于16S rRNA基因基因组内异质性会导致生物多样性被高估,而16S rRNA基因V4–V5区可将基因组内异质性对多样性的影响降至最低[29],本文通过高通量测序检测聚球藻伴生细菌16S rRNA基因V4–V5区的结果表明,淡水生态系统中的聚球藻伴生细菌有产卟啉杆菌属(Porphyrobacter)、根瘤菌属(Rhizobium)、芽孢杆菌属(Bacillus)、鞘脂单胞菌属(Sphingomonas)、水单胞菌属(Aquimonas)、假单胞菌属(Pseudomonas)、成对杆菌属(Dyadobacter)、鞘氨醇盒菌属(Sphingopyxis)、Aliihoeflea、Limnobacter和未进行分类的伴生菌等,与报道的海洋生态系统中伴生细菌种属略有不同,假单胞菌属、芽孢杆菌属和鞘脂单胞菌属为淡水和海洋生态系统共有的属,其他伴生细菌则可能为淡水生态环境专属的属,本实验分离的两株伴生细菌Rhizobium sp. JQ1和Peribacillus sp. JQ2,通过与高通量测序中相同属的OTU序列进行比对,匹配度为100%,基因组内异质性对其影响极小,证明JQ1和JQ2为淡水聚球藻伴生细菌,有学者在另一淡水藻类的伴生细菌中也发现了Porphyrobacter和Rhizobium[30],证明Rhizobium可能为淡水生态环境专属伴生细菌。
目前,已有较多有关蓝藻的相关异养细菌的报道[12, 31],但研究淡水聚球藻的较少且多采用宏基因组数据[8],分离纯化出淡水聚球藻伴生细菌并探索其对聚球藻生长影响的研究极少,蓝藻的伴生细菌群落组成与蓝藻物种、生理状态和营养限制[32]等因素有关。Xia等[1]发现聚球藻在沿海区域中的优势菌科为红杆菌科、黄杆菌科和畸单胞菌科,但未进行分离纯化;Zheng等[4, 8]发现海洋聚球藻中优势菌为黄杆菌和玫瑰杆菌,未进行分离纯化验证其功能;李孟珂等[23]分别从铜绿微囊藻和集胞藻中分离出Rhizobium,但未分离出Peribacillus;本研究丰富了蓝藻伴生细菌的种类。不同伴生细菌在藻菌共生系统中提供特定功能,分离单株伴生细菌有利于研究自身功能,例如:黄杆菌能够产生维生素K2[33]、分泌IAA[34]、溶磷、产铁载体和提高作物抗旱、抗盐的能力[35];玫瑰杆菌能够形成生物膜、降解硫化物,但对钠离子高度依赖[36];芽孢杆菌也能够分泌IAA,提供维生素B12、铁载体等物质[37-39],具有降解硫化物的能力[40],对蓝藻有促生功能;Limnobacter能将硫代硫酸盐氧化成硫酸盐,促进硫循环[41];Porphyrobacter能降解多环芳烃以及促进藻类生长[30, 42];Sphingopyxis能将氰氟草酯降解为氰氟草酸和氰氟草酚[43],免除农药对水生生态系统的影响。了解单株伴生菌的功能有利于对伴生细菌与蓝藻间相互关系的探索。
在淡水和海洋生态系统中,蓝藻与伴生细菌间的比例关系能推测蓝藻的生长状态,细菌的数量常随着水体中营养水平及藻类浓度进行波动[44],在水华期间蓝藻叶绿素a (Chla)浓度在4.9−16.1 μg/L之间进行浮动,而水体中具有代谢活性细菌的数量在0.48×106−1.68×106 cells/mL之间变动,伴生细菌占水体细菌的6%–40%左右[45],伴生细菌数量与Chla具有一定相关性,其动态变化滞后于Chla[46]。本研究发现当蓝藻与伴生细菌间的比例不同时,对蓝藻生长的影响有差异,选择适当的菌藻比有利于提高藻的生物量;有研究发现,在蓝藻中接种芽孢杆菌能明显提高蓝藻的叶绿素a含量和对苯酚降解效率,且随着菌藻接种比增加,蓝藻的生物量和对苯酚降解效率逐渐提高[47]。本研究也验证了藻菌间的比例关系对蓝藻生长有较大影响。
根据藻菌共培养以及水稻发芽试验的结果进行分析,伴生细菌JQ2对水稻发芽同样具有促生作用,说明伴生细菌在不同生境条件下也能发挥相同的作用,脱离其聚球藻藻际环境依旧可以发挥其促生功能。目前,已有通过施加蓝藻及伴生细菌复合菌系来提高水稻产量的研究[48-49],具有促进水稻发芽的蓝藻伴生细菌应用到水稻育秧生产上,有利于水稻壮秧增产。其中JQ1比JQ2对聚球藻和水稻的促进效果更好,仅添加少量菌体细胞便可促进蓝藻和水稻的生长,这可能是由于IAA分泌能力的差异导致,JQ1 (49.69 mg/L)分泌IAA的能力要高于JQ2 (29.91 mg/L),与促进蓝藻生长和水稻发芽能力相吻合,IAA含量的增加能使乙烯含量减少[50],从而促进宿主生长,因此,本实验认为IAA分泌能力是影响促生能力的重要因素,但有待获取更多菌株进一步明确蓝藻与伴生细菌之间的相互关系。
综上所述,淡水聚球藻伴生细菌与海洋聚球藻伴生细菌在群落组成上有明显不同,伴生细菌JQ1在藻菌比例为5:1时具有促生作用,伴生细菌JQ2在藻菌比例为15:1时具有促生作用,伴生细菌能促进蓝藻生长并提高其他作物发芽率,增强其秧苗素质和根系发育。
| [1] |
XIA XM, ZHENG Q, LEUNG SK, WANG Y, LEE PY, JING HM, JIAO NZ, LIU HB. Distinct metabolic strategies of the dominant heterotrophic bacterial groups associated with marine Synechococcus[J]. Science of the Total Environment, 2021, 798: 149208. DOI:10.1016/j.scitotenv.2021.149208 |
| [2] |
DUFRESNE A, OSTROWSKI M, SCANLAN DJ, GARCZAREK L, MAZARD S, PALENIK BP, PAULSEN IT, de MARSAC NT, WINCKER P, DOSSAT C, FERRIERA S, JOHNSON J, POST AF, HESS WR, PARTENSKY F. Unraveling the genomic mosaic of a ubiquitous genus of marine cyanobacteria[J]. Genome Biology, 2008, 9(5): R90. DOI:10.1186/gb-2008-9-5-r90 |
| [3] |
SCANLAN DJ, OSTROWSKI M, MAZARD S, DUFRESNE A, GARCZAREK L, HESS WR, POST AF, HAGEMANN M, PAULSEN I, PARTENSKY F. Ecological genomics of marine picocyanobacteria[J]. Microbiology and Molecular Biology Reviews: MMBR, 2009, 73(2): 249-299. DOI:10.1128/MMBR.00035-08 |
| [4] |
ZHENG Q, LU JY, WANG Y, JIAO NZ. Genomic reconstructions and potential metabolic strategies of generalist and specialist heterotrophic bacteria associated with an estuary Synechococcus culture[J]. FEMS Microbiology Ecology, 2019, 95(3): fiz017. |
| [5] |
MITCHELL BR. Chemotactic and growth responses of marine bacteria to algal extracellular products[J]. Biological Bulletin, 1972, 143(2): 265-277. DOI:10.2307/1540052 |
| [6] |
MAYALI X, AZAM F. Algicidal bacteria in the sea and their impact on algal blooms[J]. The Journal of Eukaryotic Microbiology, 2004, 51(2): 139-144. DOI:10.1111/j.1550-7408.2004.tb00538.x |
| [7] |
JING RY, WANG LY, SUN Q, LIU JJ, LIU JD, JIN J, LIU XB, WANG GH. Isolation and identification of bacteria associated with Anabaena from upland soils in northeast China[J]. Microbiology China, 2020, 47(1): 130-139. (in Chinese) 荆瑞勇, 王丽艳, 孙强, 刘俊杰, 刘居东, 金剑, 刘晓冰, 王光华. 东北旱田土壤中Anabaena伴生细菌的分离与鉴定[J]. 微生物学通报, 2020, 47(1): 130-139. |
| [8] |
ZHENG Q, WANG Y, XIE R, LANG AS, LIU YT, LU JY, ZHANG XD, SUN J, SUTTLE CA, JIAO NZ. Dynamics of heterotrophic bacterial assemblages within Synechococcus cultures[J]. Applied and Environmental Microbiology, 2018, 84(3): e01517-17. |
| [9] |
CHRISTIE-OLEZA JA, SCANLAN DJ, ARMENGAUD J. "You produce while I clean up", a strategy revealed by exoproteomics during Synechococcus-Roseobacter interactions[J]. Proteomics, 2015, 15(20): 3454-3462. DOI:10.1002/pmic.201400562 |
| [10] |
CHRISTIE-OLEZA JA, SOUSONI D, LLOYD M, ARMENGAUD J, SCANLAN DJ. Nutrient recycling facilitates long-term stability of marine microbial phototroph-heterotroph interactions[J]. Nature Microbiology, 2017, 2: 17100. DOI:10.1038/nmicrobiol.2017.100 |
| [11] |
KAUR A, HERNANDEZ-FERNAUD JR, del MAR AGUILO-FERRETJANS M, WELLINGTON EM, CHRISTIE-OLEZA JA. 100 Days of marine Synechococcus-Ruegeria pomeroyi interaction: a detailed analysis of the exoproteome[J]. Environmental Microbiology, 2018, 20(2): 785-799. DOI:10.1111/1462-2920.14012 |
| [12] |
HAYASHI S, ITOH K, SUYAMA K. Growth of the cyanobacterium Synechococcus leopoliensis CCAP1405/1 on agar media in the presence of heterotrophic bacteria[J]. Microbes and Environments, 2011, 26(2): 120-127. DOI:10.1264/jsme2.ME10193 |
| [13] |
IMEN L, NOÉMIE P, DIDIER D, CÉCILE B, JEAN-FRANÇOIS H, JULIE L. Structural diversity of bacterial communities associated with bloom-forming freshwater cyanobacteria differs according to the cyanobacterial genus[J]. PLoS One, 2017, 10(11): e0140614. |
| [14] |
AMIN SA, GREEN DH, HART MC, KÜPPER FC, SUNDA WG, CARRANO CJ. Photolysis of iron-siderophore chelates promotes bacterial-algal mutualism[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(40): 17071-17076. DOI:10.1073/pnas.0905512106 |
| [15] |
JIANG L, YANG LY, XIAO L, SHI XL, GAO G, QIN BQ. Quantitative studies on phosphorus transference occuring between Microcystis aeruginosa and its attached bacterium (Pseudomonas sp.)[J]. Hydrobiologia, 2007, 581(1): 161-165. DOI:10.1007/s10750-006-0518-0 |
| [16] |
DAO GH, WU GX, WANG XX, ZHUANG LL, ZHANG TY, HU HY. Enhanced growth and fatty acid accumulation of microalgae Scenedesmus sp. LX1 by two types of auxin[J]. Bioresource Technology, 2018, 247: 561-567. DOI:10.1016/j.biortech.2017.09.079 |
| [17] |
DAO GH, WU GX, WANG XX, ZHANG TY, ZHAN XM, HU HY. Enhanced microalgae growth through stimulated secretion of indole acetic acid by symbiotic bacteria[J]. Algal Research, 2018, 33: 345-351. DOI:10.1016/j.algal.2018.06.006 |
| [18] |
CRUZ-LÓPEZ R, MASKE H. The vitamin B1 and B12 required by the marine dinoflagellate Lingulodinium polyedrum can be provided by its associated bacterial community in culture[J]. Frontiers in Microbiology, 2016, 7: 560. |
| [19] |
CROFT MT, LAWRENCE AD, RAUX-DEERY E, WARREN MJ, SMITH AG. Algae acquire vitamin B12 through a symbiotic relationship with bacteria[J]. Nature, 2005, 438(7064): 90-93. DOI:10.1038/nature04056 |
| [20] |
MOUGET JL, DAKHAMA A, LAVOIE MC, de la NOÜE J. Algal growth enhancement by bacteria: is consumption of photosynthetic oxygen involved?[J]. FEMS Microbiology Ecology, 1995, 18(1): 35-43. DOI:10.1016/0168-6496(95)00038-C |
| [21] |
ZHANG JJ, DUAN LL, CHENG WL, JI CL, CUI HL, LI RZ. Algae-bacteria symbiosis increases biomass and oil production of Chlorella emersonii[J]. Biotechnology Bulletin, 2019, 35(5): 76-84. (in Chinese) 张靖洁, 段露露, 程蔚兰, 季春丽, 崔红利, 李润植. 菌藻共生提高小球藻生物量和产油率[J]. 生物技术通报, 2019, 35(5): 76-84. DOI:10.13560/j.cnki.biotech.bull.1985.2018-1098 |
| [22] |
ZHANG DY, LIU Y, WU Y, WANG GW, WAN BB, JIANG Y. Isolation and identification of IAA-producing strains from peanut rhizosphere and its promoting effects on peanut growth[J]. Chinese Journal of Oil Crop Sciences, 2016, 38(1): 104-110. (in Chinese) 张东艳, 刘晔, 吴越, 王国文, 万兵兵, 姜瑛. 花生根际产IAA菌的筛选鉴定及其效应研究[J]. 中国油料作物学报, 2016, 38(1): 104-110. |
| [23] |
LI MK, XIA YS, SHAN ZZ, JI T, CUI XL, XIAO W, ZHANG SY. Diversity of bacteria associated with three strains of cyanobacteria and their effects on the proliferation of Microcystis aeruginosa[J]. Journal of Yunnan University: Natural Sciences Edition, 2019, 41(6): 1238-1245. (in Chinese) 李孟珂, 夏运生, 单壮壮, 姬桐, 崔晓龙, 肖炜, 张仕颖. 三株蓝藻附着细菌多样性及其对铜绿微囊藻增殖的影响[J]. 云南大学学报(自然科学版), 2019, 41(6): 1238-1245. |
| [24] |
YANG ZM, ZHANG Q, XIE ST, WU R. Genomic DNA extraction and PCR amplication of haptophytes[J]. Journal of Biology, 2008, 25(1): 60-63. (in Chinese) 杨泽民, 章群, 谢数涛, 吴任. 金藻基因组DNA的提取与PCR扩增[J]. 生物学杂志, 2008, 25(1): 60-63. |
| [25] |
XU Y, MA DS, SONG WF, WEI XF. 16S rDNA-assisted high-throughput sequencing analysis of microbial diversity 16S rDNA-assisted high-throughput sequencing analysis of microbial diversity in oil reservoirs in oil reservoirs[J]. Chinese Journal of Applied and Environmental Biology, 2016, 22(3): 409-414. 许颖, 马德胜, 宋文枫, 魏小芳. 采用16S rDNA高通量测序技术分析油藏微生物多样性[J]. 应用与环境生物学报, 2016, 22(3): 409-414. |
| [26] |
CHEN YW, GAO XY. Comparison of two methods for phytoplankton chlorophyll-a concentration measurement[J]. Journal of Lake Sciences, 2000, 12(2): 185-188. (in Chinese) 陈宇炜, 高锡云. 浮游植物叶绿素a含量测定方法的比较测定[J]. 湖泊科学, 2000, 12(2): 185-188. DOI:10.3321/j.issn:1003-5427.2000.02.015 |
| [27] |
SHI LM, CAI YF, YANG HL, XING P, LI PF, KONG LD, KONG FX. Phylogenetic diversity and specificity of bacteria associated with Microcystis aeruginosa and other cyanobacteria[J]. Journal of Environmental Sciences, 2009, 21(11): 1581-1590. DOI:10.1016/S1001-0742(08)62459-6 |
| [28] |
ABDULAZIZ A, SAGEER S, CHEKIDHENKUZHIYIL J, VIJAYAN V, PAVANAN P, ATHIYANATHIL S, NAIR S. Unicellular cyanobacteria Synechocystis accommodate heterotrophic bacteria with varied enzymatic and metal resistance properties[J]. Journal of Basic Microbiology, 2016, 56(8): 845-856. DOI:10.1002/jobm.201500693 |
| [29] |
SUN DL, JIANG X, WU QL, ZHOU NY. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [30] |
ZHANG B, WEN R, SUN WX, SU YR, REN Q. Effect of Porphyrobacter on the flocculation harvest of Ochromonas[J]. China Oil and Fat, 2023, 48(1): 146-152. (in Chinese) 张波, 文然, 孙文昕, 苏琰儒, 任琴. 产卟啉杆菌Porphyrobacter对棕鞭藻絮凝收获的影响[J]. 中国油脂, 2023, 48(01): 146-152. |
| [31] |
KIM M, SHIN B, LEE J, PARK HY, PARK W. Culture-independent and culture-dependent analyses of the bacterial community in the phycosphere of cyanobloom-forming Microcystis aeruginosa[J]. Scientific Reports, 2019, 9(1): 20416. DOI:10.1038/s41598-019-56882-1 |
| [32] |
NAIR S, ZHANG ZH, LI HM, ZHAO HS, SHEN H, KAO SJ, JIAO NZ, ZHANG YY. Inherent tendency of Synechococcus and heterotrophic bacteria for mutualism on long-term coexistence despite environmental interference[J]. Science Advances, 2022, 8(39): eabf4792. DOI:10.1126/sciadv.abf4792 |
| [33] |
TAGVCHI H, SHIBATA T, TANI Y. Selective release of menaquinone-4 from cells of a mutant of Flavobacterium sp. 238-7 with a detergent[J]. Bioscience, Biotechnology, and Biochemistry, 1995, 59(6): 1137-1138. DOI:10.1271/bbb.59.1137 |
| [34] |
SOLTANI AA, KHAVAZI K, ASADI-RAHMANI H, OMIDVARI M, DAHAJI PA, MIRHOSEYNI H. Plant growth promoting characteristics in some Flavobacterium spp. isolated from soils of Iran[J]. Journal of Agricultural Science, 2010, 2(4): 8404-8404. |
| [35] |
ZHANG XF, SHE MZ, LI HY, JING S, JIANG HN, GAO H, ZHU YX, FU JJ. Growth promotion mechanisms of Flavobacterium succinicans and their physiological regulation on the growth and stress tolerance in Lolium perenne[J]. Acta Agrestia Sinica, 2021, 29(8): 1704-1711. (in Chinese) 张新飞, 佘木子, 李晗玉, 敬松, 姜惠娜, 高浩, 朱延晓, 付娟娟. 琥珀酸黄杆菌促生机理及其对多年生黑麦草生长和抗逆性的生理调控作用[J]. 草地学报, 2021, 29(8): 1704-1711. |
| [36] |
CHEN ZH, ZHANG YY, YANG SP. Research progresses of marine Roseobacter lineage[J]. Acta Ecologica Sinica, 2015, 35(5): 1620-1629. (in Chinese) 陈正浩, 张永雨, 杨素萍. 海洋玫瑰杆菌类群研究进展[J]. 生态学报, 2015, 35(5): 1620-1629. |
| [37] |
RUSHABH S, NATARAJAN A, PRITTESH P, HARDIK NJ, RAMAR K. Isolation and Characterization of Bacillus spp. endowed with multifarious plant growth-promoting traits and their potential effect on tomato (Lycopersicon esculentum) seedlings[J]. Arabian Journal for Science and Engineering, 2020, 45(2): 4579-4587. |
| [38] |
TILAK BR., REDDY |
| [39] |
HUANG YL, ZHENG LW, HUANG YY, JIA ZH, SONG SS, LI ZX. Effects of different application methods of Bacillus subtilis agent on soil microbial diversity and growth of muskmelon[J]. Chinese Journal of Biotechnology, 2020, 36(12): 2644-2656. (in Chinese) 黄亚丽, 郑立伟, 黄媛媛, 贾振华, 宋水山, 李再兴. 枯草芽孢杆菌菌剂不同施用方式对甜瓜土壤微生物多样性及生长的影响[J]. 生物工程学报, 2020, 36(12): 2644-2656. |
| [40] |
WANG W, DENG ZLY, JIANG JP, ZHU BC, LI J, WANG SY. Effects of Bacillus subtilis JA-14 preparation on ammonia release from the feces of broilers[J]. Journal of Henan Agricultural Sciences, 2017, 46(5): 136-139. (in Chinese) 王伟, 邓祖丽颖, 姜军坡, 朱宝成, 李佳, 王世英. 枯草芽孢杆菌JA-14菌剂对肉鸡粪便中氨气释放量的影响[J]. 河南农业科学, 2017, 46(5): 136-139. |
| [41] |
LI JY, SUN WM, SUN XX, KONG TL, LI BQ, LIU ZH, GAO P. Isolation, identification and functional verification of sulfur-oxidizing microorganisms in mine tailing[J]. Ecology and Environmental Sciences, 2022, 31(4): 785-792. (in Chinese) 李嘉义, 孙蔚旻, 孙晓旭, 孔天乐, 李宝琴, 刘振鸿, 高品. 尾矿硫氧化微生物的分离鉴定与功能验证[J]. 生态环境学报, 2022, 31(4): 785-792. |
| [42] |
WANG SL, CUI ZS, ZHENG L, GAO W, LI Q. Characterization of PAHs-degrading bacterium Porphyrobacter sp. D22F and its ecological niche in oil-degrading consortium D22-1[J]. Chinese Journal of Applied and Environmental Biology, 2012, 18(1): 122-127. (in Chinese) 王绍良, 崔志松, 郑立, 高伟, 李倩. Porphyrobacter sp. D22F的降解特性及其在石油降解菌群中的生态位[J]. 应用与环境生物学报, 2012, 18(1): 122-127. |
| [43] |
HOU Y, PEI T, WANG WY, YOU XY, NIU MF, HUANG TC. Isolation, identification, and characterization of a new cyhalofop-butyl (CyB)-degrading strain, Sphingopyxis sp. WP9[J]. Chinese Journal of Applied and Environmental Biology, 2019, 25(6): 1464-1469. (in Chinese) 侯颖, 裴韬, 王维宇, 尤晓颜, 牛明福, 黄天晨. 一株氰氟草酯降解新菌株Sphingopyxis sp. WP9的分离鉴定及降解特性[J]. 应用与环境生物学报, 2019, 25(6): 1464-1469. |
| [44] |
BIRD FDF, KALFF J. Empirical relationship between bacterial abundance and chlorophyll concentrations in fresh and marine waters[J]. Canadian Journal of Fisheries and Aquatic Sciences, 1984, 41: 1015-1023. |
| [45] |
ZHU LP, GAO G, TANG XM, CHAO JY, SHEN Y. Variation of bacteria in water and attached on Microcystis colonies and their activity during the algal blooms[J]. Journal of Lake Sciences, 2009, 21(3): 395-400. (in Chinese) 朱丽萍, 高光, 汤祥明, 晁建颖, 沈燕. 微囊藻水华期间水体及藻体上细菌的动态[J]. 湖泊科学, 2009, 21(3): 395-400. |
| [46] |
FENG S, GAO G, QIN BQ, CHEN M. Variability of bacterioplankton in the north zone of Lake Taihu[J]. Journal of Lake Sciences, 2006, 18(6): 636-642. (in Chinese) 冯胜, 高光, 秦伯强, 陈默. 太湖北部湖区水体中浮游细菌的动态变化[J]. 湖泊科学, 2006, 18(6): 636-642. |
| [47] |
YI T, SHAN Y, HUANG B, TANG T, GAO MT, WEI W. Effect of algae-bacteria co-culture on Chlorella sp. growth and phenol degradation[J]. Chinese Journal of Environmental Engineering, 2020, 14(6): 1679-1687. (in Chinese) 易涛, 山鹰, 黄渤, 唐涛, 高旻天, 魏伟. 藻菌共培养对小球藻生长及苯酚降解的影响[J]. 环境工程学报, 2020, 14(6): 1679-1687. |
| [48] |
SONG WM, ZHAO K, YANG F, WANG LY, GUO YX, WANG HZ, JING RY. Effects of nitrogen-fixing cyanobacteria and growth-promoting bacteria SM13 on yield and quality of rice[J]. Journal of Henan Agricultural Sciences, 2020, 49(10): 12-19. (in Chinese) 宋维民, 赵坤, 杨帆, 王丽艳, 郭永霞, 王海泽, 荆瑞勇. 固氮蓝藻和促生细菌SM13对水稻产量和品质的影响[J]. 河南农业科学, 2020, 49(10): 12-19. |
| [49] |
SONG WM, WANG LY, GUO YX, SUN Q, LYU YD, JING RY, WANG HZ. Effects of nitrogen-fixing cyanobacteria complex microbial inoculant and growth-promoting bacteria SM13 on yield and quality of rice with straw turnover[J]. Journal of Southern Agriculture, 2021, 52(3): 762-768. (in Chinese) 宋维民, 王丽艳, 郭永霞, 孙强, 吕艳东, 荆瑞勇, 王海泽. 秸秆还田条件下固氮蓝藻复合菌剂与促生细菌SM13对水稻产量及稻米品质的影响[J]. 南方农业学报, 2021, 52(3): 762-768. |
| [50] |
ZHOU YF, BAI YS, YUE T, LI QW, HUANG YN, JIANG W, HE C, W JB. Research progress on the growth-promoting characteristics of plant growth-promoting rhizobacteria[J]. Microbiology China, 2023, 50(2): 644-666. (in Chinese) 周益帆, 白寅霜, 岳童, 李庆伟, 黄艳娜, 蒋玮, 何川, 王金斌. 植物根际促生菌促生特性研究进展[J]. 微生物学通报, 2023, 50(2): 644-666. |
 2023, Vol. 50
2023, Vol. 50