扩展功能
文章信息
- 周凡凡, 姚虔, 刘文杰, 张凌娟
- ZHOU Fanfan, YAO Qian, LIU Wenjie, ZHANG Lingjuan
- 皮肤共生菌在皮肤健康和疾病中的作用研究进展
- Emerging roles of commensal bacteria in skin health and diseases
- 微生物学通报, 2022, 49(3): 1135-1146
- Microbiology China, 2022, 49(3): 1135-1146
- DOI: 10.13344/j.microbiol.china.210654
-
文章历史
- 收稿日期: 2021-07-19
- 接受日期: 2021-10-29
- 网络首发日期: 2021-12-28
皮肤是人体最大的器官,其表面定殖着众多微生物,据统计有104−109个/cm2微生物[1]。大部分微生物对宿主细胞无害且在某些情况下能够为宿主提供重要的保护作用,皮肤共生菌与宿主之间建立起的平衡有利于维持皮肤的健康稳态。然而,很多因素如紫外线、环境污染、机械性损伤和年龄等都会使皮肤共生菌的组成发生改变,严重失调时则会引发特应性皮炎、银屑病和痤疮等多种皮肤疾病,因此阐明皮肤共生菌的保护性作用对于皮肤疾病的防治显得尤为重要。
皮肤上天然存在着“数以亿计”的共生细菌,然而人们一直都不太清楚皮肤微生物群落如何影响人类健康。目前研究较多的肠道菌群主要通过维持全身免疫稳态来影响皮肤健康,并且肠道菌群与皮肤共生菌之间联系密切,两者有着极大的相似性。因此,本文对国内外关于肠道菌群与皮肤共生菌相关性及皮肤共生菌对宿主健康与皮肤疾病的关系进行回顾和展望,为“以菌治病”应用于皮肤疾病的防治提供更多的论据支持。
1 皮肤及皮肤共生菌概述 1.1 皮肤及共生菌介绍皮肤是人体最大的器官,是抵御外界环境侵袭和病原体入侵的第一道防线。首先,皮肤的角质层增加了皮肤的机械刚性,起到了“物理屏障”的作用。其次,一些由上皮细胞合成并在表皮不同部位表达的宿主防御分子组成宿主皮肤的“化学屏障”。其中,防御分子可以是诱导型或组成型表达的抗菌肽、蛋白酶和细胞因子,它们既可以直接抑制微生物生长,也可以作为先天性和适应性免疫反应的激活剂[2-3]。虽然皮肤中的多种细胞能保护机体免受外界有害微生物的侵袭,但截至目前,皮肤上天然存在的“数以亿计”的共生细菌与人体相互作用的机制尚不明确。皮肤是身体与外界环境产生直接接触的部位,聚集着环境中的大量微生物,所以皮肤有着独特的微生物环境稳态。在不同的皮肤部位生存着不同的共生微生物群落,这在人体感知外环境变化、预防病原菌定殖和感染、引导宿主免疫系统应对外来入侵等方面发挥着重要作用[4]。在皮肤上存在的微生物群落,主要包括细菌、病毒和真菌等,微生物群落的特性与人体宿主的防御机制共同参与了皮肤表面微环境的稳态[2]。在对皮肤及其皮肤共生菌进行研究时,应充分考虑皮肤上独特的微生物环境稳态,如微生物的多样性、对皮肤免疫力的影响及共生菌群与免疫细胞的相互作用等。
1.2 皮肤共生菌群落组成特异性的微生物物种与皮肤疾病之间存在怎样的关系及如何相互作用?回答这一问题对于理解一系列人类皮肤疾病具有重要的意义。通过皮肤微生物群的测序,可知人体皮肤微生物群包括大约113个种系型,属于6大菌门[2]。人体皮肤表层丰富的常驻型皮肤共生菌主要包括葡萄球菌属(Staphylococcus)、棒状杆菌属(Corynebacterium)和丙酸杆菌属(Propionibacterium)等[2]。其中,葡萄球菌属是一群革兰氏阳性球菌,表皮葡萄球菌(Staphylococcus epidermidis)和金黄色葡萄球菌(Staphylococcus aureus)是其中最常见的2种葡萄球菌[5]。表皮葡萄球菌是人类皮肤上最常见的一种凝固酶阴性葡萄球菌,广泛存在于皮肤上[2]。然而金黄色葡萄球菌是一种凝固酶阳性葡萄球菌,在受特应性皮炎影响的皮肤上可检测到金黄色葡萄球菌定殖增强,因此金黄色葡萄球菌被认为是导致特应性皮炎的一类病原体[6]。棒状杆菌属是一类革兰氏阳性需氧或兼性厌氧菌,与红癣和点状角化病等皮肤病有关[7]。丙酸杆菌属属于放线菌目,而痤疮丙酸杆菌(Propionibacterium acnes)是其中一种著名的革兰氏阳性厌氧菌,长期以来被认为是痤疮的重要致病因子,但其可以通过释放游离脂肪酸帮助皮肤维持较低的pH值,并阻止病原体(即金黄色葡萄球菌和链球菌)在皮肤上定殖,被认为是一种对人类健康有益的皮肤共生菌[2]。因此可见,皮肤共生菌在维持皮肤健康和正常功能方面发挥了巨大的作用。
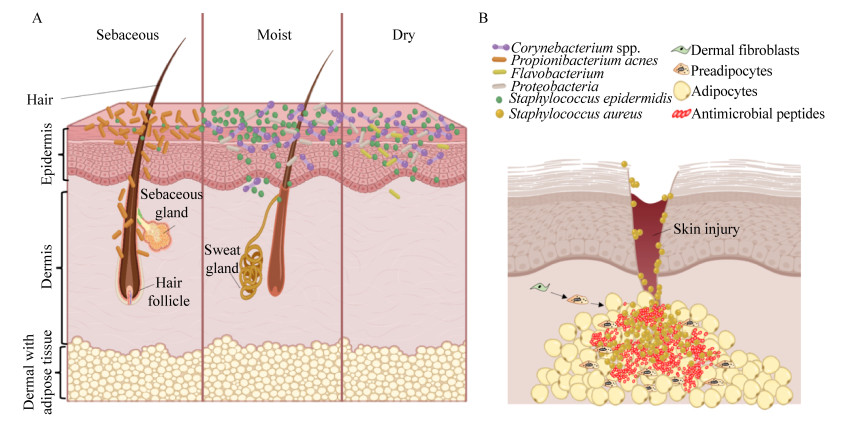
皮肤共生菌的组成情况与皮肤部位、年龄、性别等多种因素有关。多项研究证实了皮肤部位微生物组成存在特异性(图 1)。例如,丙酸杆菌属和葡萄球菌属主要分布在面部和上半身皮脂腺丰富的部位,在腋窝、肘内窝和膝内窝等潮湿部位则主要以棒状杆菌属、变形杆菌属如β-变形杆菌(Betaproteobacteria)和葡萄球菌属为主。相比之下,在前臂、手背等干燥部位,细菌种类变化较大,支持如β-变形杆菌、棒状杆菌属、葡萄球菌属和黄杆菌属(Flavobacterium)等细菌的生长[8]。最新研究发现,金黄色葡萄球菌可以通过毛囊深入皮肤,并且能高表达一类三酰甘油脂肪酶进而降解皮脂腺的皮脂从而进一步深入毛囊和真皮[9]。性别对皮肤微生物组成的影响可能与雌雄激素合成的下游效应有关,雄性激素能够促进皮脂腺分泌,使皮肤厚度增加。与男性相比,女性皮肤的pH值更偏酸,但差异并不显著,若酸性过强会导致微生物多样性降低[10]。女性和男性微生物多样性的差异还可能受到化妆品、工作环境等因素的影响[8]。此外,年龄也会影响共生菌的分布。经研究发现,新生儿皮肤的微生物菌群的复杂性显著低于成人皮肤微生物菌群,这提示了新生儿皮肤微生物菌群更易受到外界影响而改变;随着婴儿接触各种环境,以及皮肤不同部位的湿度、温度及皮脂腺汗腺密度的不同,皮肤微生物群落也随之变化,并且随着时间的推移而变得越来越多样化[11]。
|
| 图 1 正常皮肤菌群的组成及感染情况下的宿主反应 Figure 1 Composition of human skin microbiome and host response to bacterial infection. A:不同皮肤部位的微生物群落组成存在较大差异;B:金黄色葡萄球菌感染引起真皮脂肪增生及抗菌肽分泌 A: The microbiome diversity varies at different skin sites; B: Staphylococcus aureus invasion to skin dermis triggers a local dermal reactive adipogenesis response and secretion of antimicrobial peptides. |
|
|
随着皮肤共生菌的研究取得诸多突破性进展,近年来,越来越多的微生物组研究从被誉为人体“第二基因组”[12]的肠道菌群转向了皮肤共生菌。多项研究证实,肠道菌群与皮肤共生菌之间存在联系,共同影响着人类的健康与疾病[13]。肠道上皮面积约为400 m2,而成人皮肤如果算上毛囊、汗腺和皮脂腺的上皮总表面积至少为30 m2,肠道与皮肤巨大的表面积使其增加了与外界环境的接触[14],在与外界环境相互作用的同时也面临着感染的风险。与皮肤一样,肠道共生微生物群与免疫系统协同工作,保护宿主免受病原体侵袭,对于维持上皮组织内部的动态平衡及整体健康都非常重要。因此,借鉴肠道菌群研究的方法和思路有利于加快皮肤共生菌的研究进程。通过比较皮肤共生菌与肠道菌群可以让我们更全面地了解微生物组与宿主之间的互作如何影响人类身体健康。
肠道和皮肤之间存在联系,即肠道-皮肤轴(图 2)。Leyva-Castillo等[15]在小鼠模型中发现皮肤刮伤会引起小鼠肠道肥大细胞扩增、肠道通透性增加,进而推动食物过敏反应。Hill等[16]发现分娩方式对婴儿早期肠道菌群的组成有显著影响,也就是说婴儿肠道的微生物组成在一定程度上取决于顺产或剖腹产时在皮肤上定殖的微生物种类。这些研究都证实了肠道-皮肤轴的存在。
|
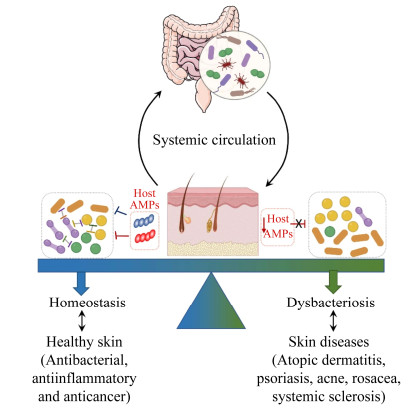
| 图 2 共生菌调控皮肤健康与疾病 Figure 2 The role of microbiota in skin health and diseases. |
|
|
肠道微生态充当着肠道-皮肤轴的主要调控因子,肠道微生物群的失调可能会改变系统免疫,导致体内平衡失调和皮肤功能受损[17]。许多研究表明,摄取益生菌会影响肠道,进而影响皮肤微生物区系。Arck等[18]饲喂小鼠乳酸杆菌可以减轻其神经源性皮肤炎症,显著延缓应激导致的毛囊退化,减少了毛囊周围MHC Ⅱ类分子阳性细胞的大量聚集。益生菌还可以改善免疫防御功能,例如乳酸杆菌可以加快紫外线诱导的免疫抑制后皮肤免疫稳态的恢复,这一过程与表皮内CD1a和DC-Lamp表达正常化、表皮细胞的同种异体刺激能力得到恢复有关[19]。肠道菌群失调能够通过肠道-皮肤轴影响多种皮肤疾病的发病,因此,服用益生菌可以调节肠道微生物菌群和全身免疫以恢复皮肤稳态,有望缓解紫外线造成的皮肤老化,改善特应性皮炎、银屑病和痤疮等皮肤疾病[20]。虽然对于共生菌群的研究目前仍主要集中于胃肠道中,但随着近年来皮肤微生物学逐渐受到重视,一些传统的观点开始发生变化。人体皮肤存在多种微生物,在一定程度上均能影响病原微生物的形成和皮肤疾病的进程。除了已知的皮肤屏障功能紊乱和免疫紊乱外,越来越多的证据表明,频发的皮肤疾病,如特应性皮炎,可能与微生物群落的失调和皮肤共生菌群的改变有关[21]。因此未来可从提高皮肤共生菌群的多样性和减少不良微生物的存在展开研究[22]。由于常驻皮肤细菌的高度多样化和复杂的影响因素,因此加强对皮肤微生物组的了解是必要的,一方面有利于充分发挥皮肤共生菌的抗菌、抗癌、抗炎等作用,另一方面有利于深入了解微生物参与人类皮肤疾病,并为开发新的抗菌治疗方法做铺垫。
2 皮肤共生菌对宿主的保护作用 2.1 抗菌共生细菌可以分泌具有生物活性的小分子多肽——抗菌肽(antimicrobial peptide,AMP),从而起到抗菌的作用[3],如表皮葡萄球菌可以通过产生酚可溶性蛋白(phenol-soluble modulin,PSM)等抗菌肽保护皮肤免受病原体侵害。此外,皮肤菌群还在塑造宿主免疫和协助刺激宿主免疫应答以防御病原微生物定殖方面发挥重要作用。Naik等将无菌小鼠(germ-free,GF)与无特定病原体(specific-pathogen-free,SPF)条件下饲养的小鼠进行比较,发现无菌小鼠的皮肤免疫反应减弱,IFN-γ和IL-17A表达水平显著降低,而IFN-γ和IL-17A能够在抵抗细菌定殖和感染的免疫应答中发挥关键作用。后续在无菌小鼠的皮肤上接种表皮葡萄球菌能观察到其皮肤上IL-17A的产生与恢复[23]。
2.2 抗炎和免疫调节在某些疾病状态下会导致微生物的平衡发生改变,这种状况被称为菌群失调。这种失衡状态可能会导致机体免疫稳态的破坏,产生某些炎症疾病症状,如特应性皮炎(atopic dermatitis,AD)。AD的发生主要与金黄色葡萄球菌的高丰度有关[24]。然而,在正常皮肤上发现的其他共生菌似乎有助于维持正常的免疫稳态,如存在于健康人体皮肤上的一种常见共生菌表皮葡萄球菌,就能减轻损伤后的炎症反应,促进抗菌肽的表达[24]。
2.3 抗癌Nakatsuji等[25]发现有一种独特的表皮葡萄球菌菌株会产生一种损害肿瘤生长的化合物6-N-羟基氨基嘌呤(6-N-hydroxyaminopurine,6-HAP),这种化合物可抑制肿瘤细胞DNA合成,并可以选择性抵抗转化的肿瘤细胞的增殖。这一结果表明,皮肤共生菌可能有助于保护宿主以防御皮肤瘤的形成[25]。科学家们认为[26] UVB引起的晒伤是导致黑色素瘤最有害的因素,因为UVB会诱导黑素细胞的DNA损伤。黑色素瘤是全球增长最快和最致命的癌症之一,尤其发生在30岁以下的年轻女性中。Wang等[26]发现皮肤共生菌如表皮葡萄球菌对UVB辐射后黑素细胞中DNA损伤的生成没有影响,同时黑素细胞还能够在承受DNA损伤的同时依旧进行生存和增殖,这是未来黑色素瘤发展的最高风险因素;然而,该研究还表明,痤疮丙酸杆菌在UVB辐射后可以通过增加细胞凋亡、产生粪卟啉和上调TNF-α抑制等方式来抑制黑素细胞的存活和增殖,最终导致大量黑素细胞死亡。这些结果表明,表皮葡萄球菌和痤疮丙酸杆菌共同作用所达到的平衡可以保护黑素细胞免受成为肿瘤细胞的风险,这些发现可能有助于预防和治疗黑色素瘤和其他与紫外线相关的皮肤癌。
3 皮肤共生菌与皮肤疾病的关系 3.1 耐药性金黄色葡萄球菌感染皮肤共生菌虽然对维持皮肤正常功能起了重要的作用,但当皮肤屏障功能受损,皮肤细菌深入真皮时可能会引发系统感染,其中金黄色葡萄球菌是皮肤和软组织上最常见的致病菌[27-28]。由于近年来抗生素滥用,抗生素耐药菌株逐渐出现,如耐甲氧西林金黄色葡萄球菌(methicillin-resistant Staphylococcus aureus,MRSA)自1960年以来一直在世界各地的医院流行,尤其是MRSA近年来肆虐全球,已经成为最致命的病原体之一,美国每年因MRSA感染的死亡人数超过2万,超过艾滋病、帕金森病、肺气肿等疾病的死亡人数[28-33],中国MRSA感染人数和死亡人数也在不断攀升[34-35]。Zhang等前期研究发现,给小鼠皮肤感染MRSA会引发真皮脂肪反应性增生(图 1),在这一过程中真皮成纤维细胞响应细菌感染后增生并往脂肪前体细胞和脂肪细胞分化,分化过程中的脂肪细胞能释放大量抗菌肽,这是宿主抵御黄色金葡萄球菌感染的一个重要机制,脂肪作为皮肤最深层次的屏障,能有效地预防细菌进入机体引发系统性感染[3, 36]。最新研究还发现真皮脂肪这一抗菌功能在TGFβ通路的介导下在老化或肥胖中丢失[37-38]。与传统的抗生素不同,抗菌肽不但能通过电荷作用直接破解细菌外膜,还能通过调控机体免疫系统间接杀伤细菌,不容易产生耐药性[3],因此开发抗菌肽药物或通过药物激活宿主抗菌免疫反应可能是治疗耐药性细菌感染的新方向。然而抗菌肽药物存在成本高及在常温下不稳定等问题,我们需要更多的研究来开发通过改变抗菌肽氨基酸序列或通过特殊药物载体(例如外泌体)而提高抗菌肽的稳定性并维持其抗菌活性的方法,这将推进抗菌肽更广泛地进入临床应用。
3.2 金黄色葡萄球菌与特应性皮炎和内瑟顿综合征特应性皮炎(AD)是最常见的免疫性疾病之一,临床上往往伴有多种并发症,如皮肤瘙痒、食欲减退、结膜炎及过敏性鼻炎等,严重影响患者的生活质量。在特应性皮炎患者中,微生物群多样性减少与疾病的严重程度增加有关,因此研究皮肤菌群在特应性皮炎中的作用有利于揭示其发病机制[39]。现代转录组学分析显示,特应性皮炎患者皮肤不同部位的基因表达谱存在差异性,大腿后部主要是角质化相关通路被激活,而上背部主要富集脂类代谢相关通路,背部皮脂腺丰富,产生具有抗菌活性的脂肪酸会抑制金黄色葡萄球菌定殖,这提示皮肤区域微环境和菌群之间存在密切联系[40]。研究证实特应性皮炎病情发作或加重与金黄色葡萄球菌在皮肤定殖有关[41],金黄色葡萄球菌形成了多种表面分子来黏附人的角质层,包括聚集因子A (clumping factors A,ClfA)和聚集因子B (clumping factors B,ClfB)、纤维连接蛋白结合蛋白(fibronectin-binding protein,FnBP)和铁调节的表面决定因子A (iron-regulated surface determinant A,ISDA)[42],同时通过分泌蛋白酶和PSMα肽损伤皮肤屏障,促进人角质形成细胞分泌IL-6、IL-1α和TNFα等促炎因子从而诱发炎症[41]。来自加州大学Dr. Gallo团队最新发表的一项临床1期研究成果中报道了一种新的针对特应性皮炎的菌群移植疗法,在此研究中研究人员为特应性皮炎患者皮肤移植一种来自健康人类皮肤并具有抗金黄色葡萄球菌活性的人葡萄球菌A9菌株,发现细菌移植治疗手段可以显著降低患者表皮金黄色葡萄球菌的数量,并进而有效改善特应性皮炎患者的临床症状,证明了细菌移植疗法的安全性和有效性[43]。与特应性皮炎类似,近期研究通过霰弹枪测序(shotgun sequencing)发现内瑟顿综合征(Netherton syndrome,NS;也称鱼鳞病样红皮病异型)病人的皮肤也大量定殖了金黄色葡萄球菌和表皮葡萄球菌,NS病人来源的金葡菌通过释放PSMα和表皮葡萄球菌释放的葡萄蛋白酶能协同破坏皮肤表皮屏障功能[44]。这些研究说明了维持宿主和细菌蛋白酶之间正常的平衡对预防皮肤病的发生至关重要。
3.3 银屑病银屑病是一种慢性炎症性疾病,病程较长且极易复发,给患者的身体健康和精神状况都带来了不良影响。据世界卫生组织统计,全球大约有1.25亿人患银屑病[45],但其病因至今还尚未研究清楚。早在50多年前,就有学者发现链球菌感染可能会诱发银屑病[46],进而有学者提出假设:银屑病是一种由链球菌超抗原诱导的T细胞介导的自身免疫性疾病[47]。近年来,越来越多的研究表明,银屑病的发生发展与皮肤共生菌紊乱从而引起的异常先天免疫反应有关[48]。目前对银屑病患者皮肤菌群多样性的研究尚无统一定论,有研究表明与健康皮肤相比,银屑病患者皮肤菌群表现出更高的多样性和异质性[49],但也有学者认为银屑病患者皮肤菌群的多样性降低[50]。Tett等[51]用高分辨率的宏基因组学方法描述了银屑病患者皮肤与健康皮肤的微生物组成差异。在门水平上,银屑病皮肤菌群主要包括放线菌门和厚壁菌门。在种水平上,金黄色葡萄球菌在银屑病患者皮损部位和非皮损部位的定殖与健康皮肤相比都有所增加,进一步研究发现金黄色葡萄球菌能够强烈诱发Th17反应,产生IL-17A、IL-17F和IL22等细胞因子[49];痤疮丙酸杆菌在健康皮肤中大量存在,通过调节CD4+ T细胞维持皮肤稳态[52],而银屑病皮损中其丰度降低,原因之一可能是银屑病斑块中皮脂腺含量降低不利于丙酸杆菌生长。与特应性皮炎不同,银屑病的皮损部位相对于健康皮肤表达更多抗菌肽[3, 53],包括cathelicidin和β defensins,我们需要更多的研究来明确抗菌肽表达失衡是否参与介导了银屑病患者的皮肤菌群失衡。
3.4 痤疮痤疮是一种发生于毛囊、皮脂腺的慢性炎症性皮肤病[54],好发于青春期,全世界大约有85%的青少年和年轻人都患过痤疮[55]。痤疮的发病原因复杂,多与皮脂分泌、毛囊角化、细菌定殖和炎症等因素有关[56]。长期以来,痤疮丙酸杆菌被认为是痤疮的主要致病菌[57],其在皮肤的定殖能够介导炎症反应,引发痤疮或加重痤疮的临床症状[56]。随着青春期激素水平的改变,皮脂分泌增加为痤疮丙酸杆菌的生长提供了适宜的环境,在出现痤疮的毛囊皮脂腺单元,痤疮丙酸杆菌丰度最高[58]。Sanford等发现痤疮丙酸杆菌释放的短链脂肪酸能通过抑制组蛋白去乙酰化酶8和9 (histone deacetylase,HDAC8和9)的表观遗传机制,来促进toll样天然免疫受体(TLR2和TLR3)介导的表皮角质形成细胞的炎症反应,从而诱发痤疮皮肤炎症[59]。然而,还有报道表明痤疮丙酸杆菌是一种有益的皮肤共生菌,可能对健康皮肤有保护作用[60],它可以产生丙酸维持酸性环境从而抵御条件致病菌金黄色葡萄球菌的繁殖。有研究对此进行了解释,痤疮丙酸杆菌及其他许多皮肤微生物在健康和疾病中都扮演双重角色,它们的功能差异可能与菌株或亚种有关[61]。Fitz-Gibbon等[58]发现痤疮丙酸杆菌的某些菌株在痤疮患者中大量存在,而其他一些菌株大多存在于健康皮肤中。在临床上,抗生素(包括红霉素和克林霉素)常被用于控制痤疮诱发的急性皮肤炎症,但是长期使用这些广谱抗生素会导致细菌产生耐药性,并且会进一步导致菌群失衡[56],因此开发更加特异性靶向痤疮丙酸杆菌乃至致病菌株的药物,或通过抗痤疮丙酸杆菌的有益皮肤共生菌的细菌移植疗法来恢复菌群平衡,是将来治疗痤疮的新方向。
3.5 玫瑰痤疮玫瑰痤疮,曾称为酒渣鼻,是一种好发于面中部、主要累及面部血管及毛囊皮脂腺单位的慢性炎症性疾病[62]。玫瑰痤疮在临床上可分为红斑毛细血管扩张型(erythematotelangiectatic rosacea,ETR)、丘疹脓疱型(papulopustular rosacea,PPR)、肥大增生型和眼型。有大量研究显示,螨类及皮肤共生菌在玫瑰痤疮的发生发展中起到重要作用。毛囊蠕形螨是目前研究最多的可能诱发玫瑰痤疮的致病因素,玫瑰痤疮患者单位面积内的毛囊蠕形螨密度明显高于正常人群,尤其是在丘疹脓疱型中[63]。临床中也发现抗蠕螨药物如甲硝唑、伊维菌素治疗玫瑰痤疮有效。除毛囊蠕形螨之外,皮肤共生菌也参与了玫瑰痤疮的发生发展。Rainer等采集了各19例玫瑰痤疮患者和健康人的鼻子和两侧脸颊的皮肤拭子,通过细菌16S rRNA基因测序发现红斑毛细血管扩张型玫瑰痤疮患者的粘液玫瑰单胞菌(Roseomonas mucosa)减少,而丘疹脓疱型与健康对照相比则富含溶脲弯曲杆菌(Campylobacter ureolyticus)、克氏棒状杆菌(Corynebacterium kroppenstedtii)和中间普雷沃菌(Prevotella intermedia)[64],这些共生菌在玫瑰痤疮中的作用还有待进一步研究。
3.6 系统性硬化症系统性硬化症也称为硬皮病,是一种免疫介导的风湿性疾病,以皮肤和内脏器官纤维化和血管病变为主要表现。系统性硬化症虽然不常见,但发病率和死亡率却很高[65]。目前已有大量研究证实,系统性硬化症与肠道菌群失调有关,但对皮肤共生菌的报道相对较少。近年来,有研究认为系统性硬化症患者的皮肤微生物组成发生了很大变化,Johnson等用RNA-Seq检测了23例系统性硬化症患者的皮肤活检样本[66],分析其微生物群落的组成与基因表达的关系,发现与健康皮肤相比,系统性硬化症患者的亲脂性共生菌(如丙酸杆菌和葡萄球菌)减少,脂质代谢通路下调,可能与系统性硬化症患者毛囊和皮脂腺萎缩有关;革兰氏阴性菌包括伯克霍尔德菌(Burkholderia)、柠檬酸杆菌(Citrobacter)和弧菌(Vibrio)的增加,与免疫激活途径密切相关,包括toll样受体和TGFβ信号,表明微生物群失调与炎性基因表达增加有关,宿主-微生物组研究对于揭示皮肤疾病的发病机制是极其必要的。
4 总结与展望笔者在本文中力求重新理解、完善和扩展皮肤及其共生菌的理念,这为认识皮肤病的微生物学提供了一个新的视角。皮肤共生菌是皮肤上存在的微生物群落,虽然肉眼难以察觉,容易被忽视,却是皮肤的重要组成部分。其中大部分微生物对宿主细胞无害并且在某些情况下能够为宿主提供重要的保护作用,若微生物菌群失调则会诱发皮肤疾病的产生。共生菌的有益作用为今后的“以菌治病”的皮肤疾病的微生物疗法带来了希望,有助于研究有效防治皮肤疾病的潜在途径。
对于未来的视角,在接下来的数年内,在理解皮肤共生菌群落的多样性和不良微生物的存在方面会取得明显的进步。同样,还将阐明有益皮肤共生菌及相应皮肤疾病有效的防治作用,包括共生菌是否作为一个独立于皮肤之外的存在的定位(皮肤-肠道),并有望能发现治疗的新靶点。
与此同时,当前其他的领域获得了迅猛发展。近年来,表征皮肤微生物群落的能力已通过测序技术而得到阐明,因此,我们对宿主和微生物之间相互作用的了解正在稳步增加。目前关于多种有益共生菌的发现已经深刻地改变了我们通过“以菌治病”内在的作用机制来调控健康和疾病的认识。现如今,研究者们已发现有多种共生菌参与了一些常见皮肤病的发病机制中,然而这些仅仅是揭开了皮肤微生物群落复杂多样性的冰山一角,还有很多的共生菌的作用还处于完全的未知状态。
最后,目前尚未明确不同的皮肤细胞类型如何与特定微生物群落富集并协作产生不同类型的皮肤疾病,以及在这些皮肤病细胞之间的相互作用影响是否影响皮肤共生菌的多样性,目前仍不清楚。随着这些见解的深入和发展,我们面临的一个主要挑战是如何改善皮肤的健康状态和提高皮肤病的诊疗技术[8]。未来的研究将需要探索不断变化的生活方式、环境乃至医疗卫生技术对人体皮肤共生菌群落的影响,该领域的进一步研究将有助于我们了解共生菌群落的改变对皮肤疾病的进展和症状的具体影响,以及我们该如何通过利用这些微生物群落来制定新的皮肤病治疗策略。
| [1] |
Weese JS. The canine and feline skin microbiome in health and disease[J]. Veterinary Dermatology, 2013, 24(1): 137-45.e31. DOI:10.1111/j.1365-3164.2012.01076.x |
| [2] |
Christensen GJM, Brüggemann H. Bacterial skin commensals and their role as host guardians[J]. Beneficial Microbes, 2014, 5(2): 201-215. DOI:10.3920/BM2012.0062 |
| [3] |
Zhang LJ, Gallo RL. Antimicrobial peptides[J]. Current Biology, 2016, 26(1): R14-R19. DOI:10.1016/j.cub.2015.11.017 |
| [4] |
Barnard E, Li HY. Shaping of cutaneous function by encounters with commensals[J]. The Journal of Physiology, 2017, 595(2): 437-450. DOI:10.1113/JP271638 |
| [5] |
Yong YY, Dykes GA, Choo WS. Biofilm formation by Staphylococci in health-related environments and recent reports on their control using natural compounds[J]. Critical Reviews in Microbiology, 2019, 45(2): 201-222. DOI:10.1080/1040841X.2019.1573802 |
| [6] |
Edslev SM, Agner T, Andersen PS. Skin microbiome in atopic dermatitis[J]. Acta Dermato Venereologica, 2020, 100(12): adv00164. DOI:10.2340/00015555-3514 |
| [7] |
Scharschmidt TC, Fischbach MA. What lives on our skin: ecology, genomics and therapeutic opportunities of the skin microbiome[J]. Drug Discovery Today: Disease Mechanisms, 2013, 10(3/4): e83-e89. |
| [8] |
SanMiguel A, Grice EA. Interactions between host factors and the skin microbiome[J]. Cellular and Molecular Life Sciences, 2015, 72(8): 1499-1515. DOI:10.1007/s00018-014-1812-z |
| [9] |
Nakamura K, Williams MR, Kwiecinski JM, Horswill AR, Gallo RL. Staphylococcus aureus enters hair follicles using triacylglycerol lipases preserved through the genus Staphylococcus[J]. The Journal of Investigative Dermatology, 2021, 141(8): 2094-2097. DOI:10.1016/j.jid.2021.02.009 |
| [10] |
Fierer N, Hamady M, Lauber CL, Knight R. The influence of sex, handedness, and washing on the diversity of hand surface bacteria[J]. PNAS, 2008, 105(46): 17994-17999. DOI:10.1073/pnas.0807920105 |
| [11] |
Grice EA, Kong HH, Conlan S, Deming CB, Davis J, Young AC, Program NCS, Bouffard GG, Blakesley RW, Murray PR, et al. Topographical and temporal diversity of the human skin microbiome[J]. Science, 2009, 324(5931): 1190-1192. DOI:10.1126/science.1171700 |
| [12] |
David LA, Maurice CF, Carmody RN, Gootenberg DB, Button JE, Wolfe BE, Ling AV, Devlin AS, Varma Y, Fischbach MA, et al. Diet rapidly and reproducibly alters the human gut microbiome[J]. Nature, 2014, 505(7484): 559-563. DOI:10.1038/nature12820 |
| [13] |
O'Sullivan JN, Rea MC, Hill C, Ross RP. Protecting the outside: biological tools to manipulate the skin microbiota[J]. FEMS Microbiology Ecology, 2020, 96(6): fiaa085. DOI:10.1093/femsec/fiaa085 |
| [14] |
Coates M, Lee MJ, Norton D, MacLeod AS. The skin and intestinal microbiota and their specific innate immune systems[J]. Frontiers in Immunology, 2019, 10: 2950. DOI:10.3389/fimmu.2019.02950 |
| [15] |
Leyva-Castillo JM, Galand C, Kam C, Burton O, Gurish M, Musser MA, Goldsmith JD, Hait E, Nurko S, Brombacher F, et al. Mechanical skin injury promotes food anaphylaxis by driving intestinal mast cell expansion[J]. Immunity, 2019, 50(5): 1262-1275.e4. DOI:10.1016/j.immuni.2019.03.023 |
| [16] |
Hill CJ, Lynch DB, Murphy K, Ulaszewska M, Jeffery IB, O'Shea CA, Watkins C, Dempsey E, Mattivi F, Tuohy K, et al. Evolution of gut microbiota composition from birth to 24 weeks in the infantmet cohort[J]. Microbiome, 2017, 5(1): 4. DOI:10.1186/s40168-016-0213-y |
| [17] |
Visser MJE, Kell DB, Pretorius E. Bacterial dysbiosis and translocation in psoriasis vulgaris[J]. Frontiers in Cellular and Infection Microbiology, 2019, 9: 7. DOI:10.3389/fcimb.2019.00007 |
| [18] |
Arck P, Handjiski B, Hagen E, Pincus M, Bruenahl C, Bienenstock J, Paus R. Is there a 'gut-brain-skin axis'?[J]. Experimental Dermatology, 2010, 19(5): 401-405. DOI:10.1111/j.1600-0625.2009.01060.x |
| [19] |
Peguet-Navarro J, Dezutter-Dambuyant C, Buetler T, Leclaire J, Smola H, Blum S, Bastien P, Breton L, Gueniche A. Supplementation with oral probiotic bacteria protects human cutaneous immune homeostasis after UV exposure-double blind, randomized, placebo controlled clinical trial[J]. European Journal of Dermatology: EJD, 2008, 18(5): 504-511. |
| [20] |
Zhang YL, You CP. Research progress on the correlation between gut microbiota and hair loss[J]. Microbiology China, 2021, 48(10): 3860-3871. (in Chinese) 张怡琳, 游春苹. 肠道微生物群与脱发的相关性研究进展[J]. 微生物学通报, 2021, 48(10): 3860-3871. |
| [21] |
Uberoi A, Bartow-Mckenney C, Zheng Q, Flowers L, Campbell A, Knight SaB, Chan N, Wei M, Lovins V, Bugayev J, et al. Commensal microbiota regulates skin barrier function and repair via signaling through the aryl hydrocarbon receptor[J]. Cell Host & Microbe, 2021, 29(8): 1235-1248.e8. |
| [22] |
Fyhrquist N, Salava A, Auvinen P, Lauerma A. Skin biomes[J]. Current Allergy and Asthma Reports, 2016, 16(5): 40. DOI:10.1007/s11882-016-0618-5 |
| [23] |
Naik S, Bouladoux N, Wilhelm C, Molloy MJ, Salcedo R, Kastenmuller W, Deming C, Quinones M, Koo L, Conlan S, et al. Compartmentalized control of skin immunity by resident commensals[J]. Science, 2012, 337(6098): 1115-1119. DOI:10.1126/science.1225152 |
| [24] |
Nakatsuji T, Chen TH, Narala S, Chun KA, Two AM, Yun T, Shafiq F, Kotol PF, Bouslimani A, Melnik AV, et al. Antimicrobials from human skin commensal bacteria protect against Staphylococcus aureus and are deficient in atopic dermatitis[J]. Science Translational Medicine, 2017, 9(378): eaah4680. DOI:10.1126/scitranslmed.aah4680 |
| [25] |
Nakatsuji T, Chen TH, Butcher AM, Trzoss LL, Nam SJ, Shirakawa KT, Zhou W, Oh J, Otto M, Fenical W, et al. A commensal strain of Staphylococcus epidermidis protects against skin neoplasia[J]. Science Advances, 2018, 4(2): eaao4502. DOI:10.1126/sciadv.aao4502 |
| [26] |
Wang ZP, Choi JE, Wu CC, Di Nardo A. Skin commensal bacteria Staphylococcus epidermidis promote survival of melanocytes bearing UVB-induced DNA damage, while bacteria Propionibacterium acnes inhibit survival of melanocytes by increasing apoptosis[J]. Photodermatology, Photoimmunology & Photomedicine, 2018, 34(6): 405-414. |
| [27] |
Gallo RL, Hooper LV. Epithelial antimicrobial defence of the skin and intestine[J]. Nature Reviews Immunology, 2012, 12(7): 503-516. DOI:10.1038/nri3228 |
| [28] |
Tong SYC, Davis JS, Eichenberger E, Holland TL, Fowler VG. Staphylococcus aureus infections: epidemiology, pathophysiology, clinical manifestations, and management[J]. Clinical Microbiology Reviews, 2015, 28(3): 603-661. DOI:10.1128/CMR.00134-14 |
| [29] |
DeLeo FR, Otto M, Kreiswirth BN, Chambers HF. Community-associated meticillin-resistant Staphylococcus aureus[J]. Lancet, 2010, 375(9725): 1557-1568. DOI:10.1016/S0140-6736(09)61999-1 |
| [30] |
Tenover FC, Goering RV. Methicillin-resistant Staphylococcus aureus strain USA300: origin and epidemiology[J]. The Journal of Antimicrobial Chemotherapy, 2009, 64(3): 441-446. DOI:10.1093/jac/dkp241 |
| [31] |
Otto M. MRSA virulence and spread[J]. Cellular Microbiology, 2012, 14(10): 1513-1521. DOI:10.1111/j.1462-5822.2012.01832.x |
| [32] |
Klevens RM, Morrison MA, Nadle J, Petit S, Gershman K, Ray S, Harrison LH, Lynfield R, Dumyati G, Townes JM, et al. Invasive methicillin-resistant Staphylococcus aureus infections in the United States[J]. JAMA, 2007, 298(15): 1763-1771. DOI:10.1001/jama.298.15.1763 |
| [33] |
Klein E, Smith DL, Laxminarayan R. Hospitalizations and deaths caused by methicillin-resistant Staphylococcus aureus, United States, 1999-2005[J]. Emerging Infectious Diseases, 2007, 13(12): 1840-1846. DOI:10.3201/eid1312.070629 |
| [34] |
Wushouer H, Guan XD, Han S, Shi LW. Clinical and economic burden of bloodstream infections caused by meticillin-resistant and meticillin-susceptible Staphylococcus aureus in Chinese tertiary hospitals from 2010 to 2017: a multicentre, retrospective cohort study[J]. The Lancet, 2018, 392: S67. |
| [35] |
Qiao F, Huang WZ, Cai L, Zong ZY, Yin WJ. Methicillin-resistant Staphylococcus aureus nasal colonization and infection in an intensive care unit of a university hospital in China[J]. The Journal of International Medical Research, 2018, 46(9): 3698-3708. DOI:10.1177/0300060518777812 |
| [36] |
Zhang LJ, Guerrero-Juarez CF, Hata T, Bapat SP, Ramos R, Plikus MV, Gallo RL. Dermal adipocytes protect against invasive Staphylococcus aureus skin infection[J]. Science, 2015, 347(6217): 67-71. DOI:10.1126/science.1260972 |
| [37] |
Zhang LJ, Guerrero-Juarez CF, Chen SX, Zhang XW, Yin MM, Li FW, Wu S, Chen J, Li M, Liu YZ, et al. Diet-induced obesity promotes infection by impairment of the innate antimicrobial defense function of dermal adipocyte progenitors[J]. Science Translational Medicine, 2021, 13(577): eabb5280. DOI:10.1126/scitranslmed.abb5280 |
| [38] |
Zhang LJ, Chen SX, Guerrero-Juarez CF, Li FW, Tong Y, Liang YQ, Liggins M, Chen X, Chen H, Li M, et al. Age-related loss of innate immune antimicrobial function of dermal fat is mediated by transforming growth factor beta[J]. Immunity, 2019, 50(1): 121-136.e5. DOI:10.1016/j.immuni.2018.11.003 |
| [39] |
Sun LX, Liu WJ, Zhang LJ. The role of toll-like receptors in skin host defense, psoriasis, and atopic dermatitis[J]. Journal of Immunology Research, 2019, 2019: 1824624. |
| [40] |
Ottman N, Barrientos-Somarribas M, Fyhrquist N, Alexander H, Wisgrill L, Olah P, Tsoka S, Greco D, Levi-Schaffer F, Soumelis V, et al. Microbial and transcriptional differences elucidate atopic dermatitis heterogeneity across skin sites[J]. Allergy, 2021, 76(4): 1173-1187. DOI:10.1111/all.14606 |
| [41] |
Williams MR, Costa SK, Zaramela LS, Khalil S, Todd DA, Winter HL, Sanford JA, O'Neill AM, Liggins MC, Nakatsuji T, et al. Quorum sensing between bacterial species on the skin protects against epidermal injury in atopic dermatitis[J]. Science Translational Medicine, 2019, 11(490): eaat8329. DOI:10.1126/scitranslmed.aat8329 |
| [42] |
Paller AS, Kong HH, Seed P, Naik S, Scharschmidt TC, Gallo RL, Luger T, Irvine AD. The microbiome in patients with atopic dermatitis[J]. Journal of Allergy and Clinical Immunology, 2019, 143(1): 26-35. DOI:10.1016/j.jaci.2018.11.015 |
| [43] |
Nakatsuji T, Hata TR, Tong Y, Cheng JY, Shafiq F, Butcher AM, Salem SS, Brinton SL, Rudman Spergel AK, Johnson K, et al. Development of a human skin commensal microbe for bacteriotherapy of atopic dermatitis and use in a phase 1 randomized clinical trial[J]. Nature Medicine, 2021, 27(4): 700-709. DOI:10.1038/s41591-021-01256-2 |
| [44] |
Williams MR, Cau L, Wang YC, Kaul D, Sanford JA, Zaramela LS, Khalil S, Butcher AM, Zengler K, Horswill AR, et al. Interplay of staphylococcal and host proteases promotes skin barrier disruption in netherton syndrome[J]. Cell Reports, 2020, 30(9): 2923-2933.e7. DOI:10.1016/j.celrep.2020.02.021 |
| [45] |
Organization WH. Global Report on Psoriasis[M]. Washington, DC: World Health Organization, 2016.
|
| [46] |
Norrlind R. Psoriasis following infections with hemolytic streptococci[J]. Acta Dermato Venereologica, 1950, 30(1): 64-72. |
| [47] |
Valdimarsson H, Baker BS, Jónsdóttir I, Powles A, Fry L. Psoriasis: a T-cell-mediated autoimmune disease induced by streptococcal superantigens?[J]. Immunology Today, 1995, 16(3): 145-149. DOI:10.1016/0167-5699(95)80132-4 |
| [48] |
Fry L, Baker BS, Powles AV, Engstrand L. Psoriasis is not an autoimmune disease?[J]. Experimental Dermatology, 2015, 24(4): 241-244. DOI:10.1111/exd.12572 |
| [49] |
Chang HW, Yan D, Singh R, Liu J, Lu XY, Ucmak D, Lee K, Afifi L, Fadrosh D, Leech J, et al. Alteration of the cutaneous microbiome in psoriasis and potential role in Th17 polarization[J]. Microbiome, 2018, 6(1): 154. DOI:10.1186/s40168-018-0533-1 |
| [50] |
Alekseyenko AV, Perez-Perez GI, De Souza A, Strober B, Gao Z, Bihan M, Li K, Methé BA, Blaser MJ. Community differentiation of the cutaneous microbiota in psoriasis[J]. Microbiome, 2013, 1(1): 31. DOI:10.1186/2049-2618-1-31 |
| [51] |
Tett A, Pasolli E, Farina S, Truong DT, Asnicar F, Zolfo M, Beghini F, Armanini F, Jousson O, De Sanctis V, et al. Unexplored diversity and strain-level structure of the skin microbiome associated with psoriasis[J]. NPJ Biofilms and Microbiomes, 2017, 3: 14. DOI:10.1038/s41522-017-0022-5 |
| [52] |
Agak GW, Kao S, Ouyang K, Qin M, Moon D, Butt A, Kim J. Phenotype and antimicrobial activity of Th17 cells induced by Propionibacterium acnes strains associated with healthy and acne skin[J]. The Journal of Investigative Dermatology, 2018, 138(2): 316-324. DOI:10.1016/j.jid.2017.07.842 |
| [53] |
Zhang LJ, Sen GL, Ward NL, Johnston A, Chun K, Chen YF, Adase C, Sanford JA, Gao NN, Chensee M, et al. Antimicrobial peptide LL37 and MAVS signaling drive interferon-β production by epidermal keratinocytes during skin injury[J]. Immunity, 2016, 45(1): 119-130. DOI:10.1016/j.immuni.2016.06.021 |
| [54] |
Zheng YM, Meng RL, Peng HY, Jiang R, Song LY, Dong K, He CF. Variation of skin microbial community in adolescent acne[J]. Microbiology China, 2019, 46(12): 3414-3423. (in Chinese) 郑玉梅, 孟若琳, 彭海悦, 蒋锐, 宋丽雅, 董坤, 何聪芬. 青少年痤疮面部皮肤微生物群落结构变化[J]. 微生物学通报, 2019, 46(12): 3414-3423. |
| [55] |
White GM. Recent findings in the epidemiologic evidence, classification, and subtypes of acne vulgaris[J]. Journal of the American Academy of Dermatology, 1998, 39(2): S34-S37. DOI:10.1016/S0190-9622(98)70442-6 |
| [56] |
Xu HX, Li HY. Acne, the skin microbiome, and antibiotic treatment[J]. American Journal of Clinical Dermatology, 2019, 20(3): 335-344. DOI:10.1007/s40257-018-00417-3 |
| [57] |
Leeming JP, Holland KT, Cuncliffe WJ. The microbial colonization of inflamed acne vulgaris lesions[J]. The British Journal of Dermatology, 1988, 118(2): 203-208. DOI:10.1111/j.1365-2133.1988.tb01775.x |
| [58] |
Fitz-Gibbon S, Tomida S, Chiu BH, Nguyen L, Du C, Liu M, Elashoff D, Erfe MC, Loncaric A, Kim J, et al. Propionibacterium acnes strain populations in the human skin microbiome associated with acne[J]. Journal of Investigative Dermatology, 2013, 133(9): 2152-2160. DOI:10.1038/jid.2013.21 |
| [59] |
Sanford JA, Zhang LJ, Williams MR, Gangoiti JA, Huang CM, Gallo RL. Inhibition of HDAC8 and HDAC9 by microbial short-chain fatty acids breaks immune tolerance of the epidermis to TLR ligands[J]. Science Immunology, 2016, 1(4): eaah4609. DOI:10.1126/sciimmunol.aah4609 |
| [60] |
Barnard E, Shi BC, Kang DZ, Craft N, Li HY. The balance of metagenomic elements shapes the skin microbiome in acne and health[J]. Scientific Reports, 2016, 6: 39491. DOI:10.1038/srep39491 |
| [61] |
Ramasamy S, Barnard E, Dawson TL, Li H. The role of the skin microbiota in acne pathophysiology[J]. The British Journal of Dermatology, 2019, 181(4): 691-699. DOI:10.1111/bjd.18230 |
| [62] |
Rosacea Research Center, Chinese Society of Dermatology, Rosacea Professional Committee, Chinese Dermatologist Association. Guidelines for the diagnosis and treatment of rosacea in China (2021 edition)[J]. Chinese Journal of Dermatology, 2021, 54(4): 279-288. (in Chinese) 中华医学会皮肤性病学分会玫瑰痤疮研究中心, 中国医师协会皮肤科医师分会玫瑰痤疮专业委员会. 中国玫瑰痤疮诊疗指南(2021版)[J]. 中华皮肤科杂志, 2021, 54(4): 279-288. |
| [63] |
Bonnar E, Eustace P, Powell FC. The Demodex mite population in rosacea[J]. Journal of the American Academy of Dermatology, 1993, 28(3): 443-448. DOI:10.1016/0190-9622(93)70065-2 |
| [64] |
Rainer BM, Thompson KG, Antonescu C, Florea L, Mongodin EF, Bui J, Fischer AH, Pasieka HB, Garza LA, Kang S, et al. Characterization and analysis of the skin microbiota in rosacea: a case-control study[J]. American Journal of Clinical Dermatology, 2020, 21(1): 139-147. DOI:10.1007/s40257-019-00471-5 |
| [65] |
Denton CP, Khanna D. Systemic sclerosis[J]. Lancet, 2017, 390(10103): 1685-1699. DOI:10.1016/S0140-6736(17)30933-9 |
| [66] |
Johnson ME, Franks JM, Cai GS, Mehta BK, Wood TA, Archambault K, Pioli PA, Simms RW, Orzechowski N, Arron S, et al. Microbiome dysbiosis is associated with disease duration and increased inflammatory gene expression in systemic sclerosis skin[J]. Arthritis Research & Therapy, 2019, 21(1): 49. |
 2022, Vol. 49
2022, Vol. 49




