扩展功能
文章信息
- 王雅琦, 张宇萌, 林小煌, 杨明坤, 葛峰
- WANG Yaqi, ZHANG Yumeng, LIN Xiaohuang, YANG Mingkun, GE Feng
- ClpP2蛋白失活对集胞藻PCC6803生长、光合作用和蛋白质组的影响
- Effects of ClpP2 protein inactivation on growth, photosynthesis, and proteome of Synechocystis sp. PCC6803
- 微生物学通报, 2023, 50(3): 1205-1219
- Microbiology China, 2023, 50(3): 1205-1219
- DOI: 10.13344/j.microbiol.china.220638
-
文章历史
- 收稿日期: 2022-07-09
- 接受日期: 2022-08-26
- 网络首发日期: 2022-11-16
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
蛋白质降解是维持细胞正常生理活动的重要环节[1]。在原核生物中,超过80%的待降解蛋白质通过蛋白酶体系统介导水解[2-3],酪蛋白裂解酶(Clp)复合物是其中重要的一类蛋白酶体系统。Clp复合物高度保守且广泛存在[4-5],最先在大肠杆菌中被鉴定,之后陆续在蓝细菌、高等植物叶绿体、哺乳动物线粒体中被发现。Clp复合物由蛋白酶和分子伴侣两部分组成,ClpP蛋白酶作为水解核心,并与Clp分子伴侣蛋白组装成多聚体如ClpC、ClpX等。通常,Clp分子伴侣蛋白识别并对底物进行去折叠化,将预处理后的底物转移到ClpP的蛋白水解室进行水解[6]。
作为水解核心的ClpP蛋白酶是一类典型的ATP依赖型丝氨酸蛋白酶,一般不是细菌生长必需蛋白质,但参与重要的细胞代谢活动,同时也是细菌适应环境胁迫必不可少的蛋白质。现有对光合自养生物原核生物的研究发现蓝细菌中存在复杂多样的ClpP蛋白,在集胞藻PCC 6803中已知4种ClpP蛋白酶,分别是ClpP1−ClpP4;此外,集胞藻中除了ClpX还有ClpC作为ClpP分子伴侣[7-8]。截至目前,对蓝细菌ClpP蛋白的研究较为浅显,相关的研究报道较少,对ClpP的底物、分子伴侣、功能等尚未明确。
越来越多的研究利用转录组、蛋白质组、代谢组学的方法对细菌中ClpP蛋白进行探究来了解其底物与分子机制,目前对蓝细菌ClpP蛋白相关的组学信息非常有限。为探究ClpP2蛋白对集胞藻的生长并揭示其功能和底物,本研究构建ClpP2蛋白失活的集胞藻突变株,进行生长实验并检测光合参数,结合相对和绝对定量同位素标记(isobaric tag for relative absolute quantitation, iTRAQ)与液相色谱-串联质谱(LC-MS/MS)技术,筛选并分析在ClpP2失活的影响下表达异常的蛋白质,利用平行反应监测(parallel reaction monitoring, PRM)技术验证部分iTRAQ实验结果,初步探究集胞藻ClpP2功能,并为ClpP2蛋白酶底物探究提供可靠底物簇。
1 材料与方法 1.1 材料实验所用集胞藻PCC6803来自中国科学院淡水藻种库,菌株接种在BG-11培养基[7],放置在30 ℃培养箱中接受白炽灯光照强度为40 μE,并充气培养。用于质粒克隆的菌为接种在LB培养基[9]中的大肠杆菌(Escherichia coli) DH5α,放置在37 ℃恒温培养箱中培养。实验所用质粒均为本实验室保存。
主要试剂和仪器:硫酸卡那霉素,Aladdin;二氯苯基二甲基[3-(3,4-dichlorophenyl)-1,1-dimethylurea, DCMU],奥默生物技术(上海)有限公司;三氟乙酸(trifluoroacetic acid, TFA)和乙腈,Acrosorganics;iTRAQs 6-Plex试剂盒,Sigma-Aldrich (上海)贸易有限公司。双通道调制叶绿素荧光仪,WALZ公司;LC色谱仪,Shimadzu公司;C18柱,Waters公司;色谱仪、质谱仪、色谱柱和RSLC C18柱,Thermo Fisher Scientific公司。
1.2 突变株的构建与菌株培养本研究使用集胞藻野生型(WT)与ClpP突变株(ΔClpP2)两种菌株进行实验。ΔClpP2的构建采用基因靶向诱变方法:在集胞藻基因组中定向插入卡娜霉素抗性基因序列,使编码ClpP2的基因(sll0534)发生靶向诱变。通过固体抗性培养基筛选培养,获得ΔClpP2单克隆后通过PCR与测序进行验证。其中固体抗性培养基为加入终浓度为12.5 μg/mL硫酸卡那霉素的BG-11固体培养基。
菌株挑取单克隆放入含150 mL液体BG-11培养基的250 mL规格锥形瓶中生长,瓶中匀速泵入过滤除菌的空气。其中ΔClpP2培养瓶中加入终浓度为25 μg/mL的硫酸卡那霉素。本研究所有实验均采用对数生长期菌种,即控制所用菌种液的光密度OD730为0.8±0.05。
正常培养条件(normal condition, NC)设置:温度设置为30 ℃,白炽灯光强为40。胁迫处理:强光(high light condition, HL)设置光强为200 μE,高温(high temperature condition, HT)设置温度为42 ℃[9],其他同正常条件。测定生长曲线时,将对数生长期菌株稀释至OD730为0.01,分别在NC、HT或HL条件下培养5 d,每12 h测量一次OD730。倍增时间通过对数生长期的菌株增长率进行计算。
1.3 叶绿素荧光参数分析为监测WT和ΔClpP2之间的光合生理变化,用双通道调制叶绿素荧光仪按照使用手册的指导测定叶绿素荧光和P700氧化还原动力学曲线[10]。菌株培养到对数期(OD730约为0.8–1.0)后收集,需要胁迫处理的则在收集前将对数期菌株放在HT或HL环境中1 h。
取收集到的菌液4 mL暗适应15 min后导入Dual-PAM-100配套的石英玻璃皿中,将玻璃皿插入PAM荧光计中。测定叶绿素荧光诱导曲线,饱和脉冲设置为10 000 μE,打开光化光,荧光信号出现峰值后打开测量光,每30 s给予一次饱和脉冲,当荧光曲线稳定后关闭光化光,同时对光照后关闭光化光后的叶绿素荧光瞬态进行监测并获得Post-illumination曲线[11-12]。光化学效率根据PSⅡ的最大光合效率(Fv/Fm)确定,公式为[13]:Fv/Fm=(Fm−F0)/Fm。
测定快速光曲线,每10 s给予逐步增加的饱和脉冲,饱和脉冲PAR从11–693 μE呈对数递增。荧光电子转移速率[rRealelectron transport rate of PSⅡ, ETR(Ⅱ)]和光化光下有效PSⅡ产率[actual photosynthetic quantum yield of PSⅡ, Y(Ⅱ)]的快速光曲线(rapid light curves, RLC)被软件自动记录生成文档。
为测量P700氧化还原动力学曲线,将菌液OD730调整为2.0±0.05,在15 min暗适应前加入浓度为10 μmol/L的电子阻断剂DCMU。双通道调制叶绿素荧光仪操作遵循Klughammer等[14]的方法并进行适当修改以适应集胞藻。吸光度范围为820−860 nm。打开测量灯,远红光持续30 s。测量光终止后得到曲线,曲线进行归一化处理,根据曲线拟合衰减函数计算半衰期[10]。
1.4 总蛋白的提取和酶解取200 mL对数生长期的WT和ΔClpP2菌液按照参考文献[15]的方法,直接提取菌株总蛋白并进行酶解。WT和ΔClpP2设置3个生物重复,共6组样品。酶解结束后加入0.1%的TFA终止反应,在4 ℃、2 500×g条件下离心10 min,保留上清液,放入真空干燥浓缩仪干燥,获得肽段样品,放入−80 ℃保存。
1.5 iTRAQ标记与肽段分离肽样品用TEAB和ddH2O各15 μL复溶,采用iTRAQs 6-Plex试剂盒按照操作手册进行标记。6组样品在室温孵育2 h后合并[16]。将样品通过上样器上样,使用梯度为2%−80%的缓冲液B (80%乙腈,5 mmol/L NH4OH)在色谱仪中以1 mL/min流速洗脱90 min,温度设置为40 ℃。按时间间隔收集90组样本,最后合并为12组。3.5 m×4.6 mm×250 mm XBridge Shield C18柱用缓冲液A (H2O, 5 mol/L NH4OH)洗涤后装入预分离的肽段进行除盐处理,真空离心干燥后用0.1%的TFA溶解,4 ℃、11 000×g条件下离心5 min,保留上清液用于LC-MS/MS分析。
1.6 LC-MS/MS分析LC-MS/MS分析结合色谱仪和Q质谱仪,质谱(MS)数据用数据依赖获取(data dependent acquisition, DDA)模式采集。2 μm×75 μm×20 mm规格的色谱柱用95%的缓冲液A (0.1%甲酸水溶液)平衡,除盐的肽段样品用2%−80%的缓冲液B (80%乙腈,0.1%甲酸),流速为250 μL/min,洗脱梯度:0、20% B;20% B,1 min;40% B,8 min;80% B,9 min;80% B,11 min;20% B,11.1 min;20% B,14 min。洗脱后将样品装入2 μm×75 μm×20 mm C18柱中,喷入质谱仪进行波普扫描。参数设置与实验室之前的研究[15]一致。
1.7 蛋白鉴定与定量分析将所有鉴定的质谱结果导入已有集胞藻蛋白质数据库的MaxQuant (version 1.3.0.5)软件进行蛋白质鉴定,注释信息从资源网站Cyanobase Synechocystis sp. PCC 6803 2015.10.16 (http://genome.microbedb.jp/cyanobase/GCA_001318385.1)下载。软件参数设置为:parent ion error为10 PPM,secondary ion error reference值为0.05 Da,trypsin omission site选择2,数据集的错误发现率(false discovery rate)小于0.01。
所注释到的蛋白质通过基因本体论(gene ontology, GO, http://www.geneontology.org/)与京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes, KEGG, http://www.genome.jp/kegg/)通路分析进行数据处理。
1.8 PRM验证差异表达蛋白为消除生物误差,选取iTRAQ实验同一批样品的肽段进行PRM验证实验。肽段样品从−80 ℃取出后用0.1% TFA复溶。采用色谱仪联用质谱仪对肽样品进行LC-MS/MS分析。MS数据采集采用DDA方式。用8%−95%缓冲液B (90%乙腈,0.1%甲酸水溶液)以350 nL/min的流速洗脱肽,洗脱时间设置为130 min。洗脱梯度设置为:0−91 min, 8%−25% B;92−117 min,25%−45% B;118−119 min,45%−90% B;120−130 min,90% B。洗脱后的样品喷入质谱仪进行扫描。结果用Skyline (3.5.1.9942)软件进行分析,定量结果归一化作为标准参考。根据iTRAQ的数据结果,选择45个蛋白进行PRM定量验证分析。
1.9 统计分析所有实验独立重复3次。所有实验数据均使用Origin 2021软件进行处理。数据采用t检验(单尾),并以标准差(SD)表示。P < 0.05为差异有统计学意义。
2 结果与分析 2.1 ΔClpP2的生长与应对胁迫情况 2.1.1 ΔClpP2构建成功并能光合自养根据拟南芥同源基因,在集胞藻中预测出4个Clp蛋白酶亚基,分别为ClpP2 (sll0534)、ClpP1 (slr0542)、Clp4/R (slr0164)和ClpP3 (slr0165)[17]。本研究通过同源重组原理将卡娜霉素抗性基因序列(Kmr)替换部分野生型集胞藻(WT) clpP1 (slr0542)、clpP2 (sll0534)序列,尝试构建ClpP1和ClpP2突变株[7],最终ClpP2突变株(ΔClpP2)成功构建并存活,利用clpP2两端引物确认Kmr片段替换成功(图 1B),并进行测序验证,所用引物见表 1;ClpP1突变株无法获得单克隆(数据未显示),构建失败。
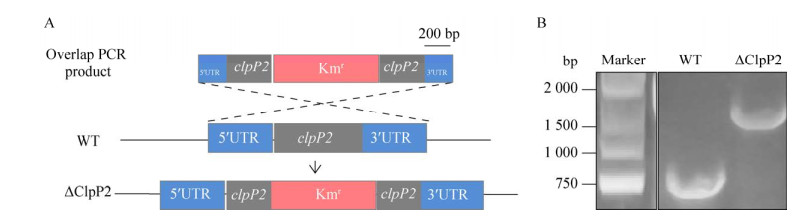
|
| 图 1 ClpP2敲除突变体的构建与验证 Figure 1 The construction schematic and verification. A:突变体构建示意图. B:ΔClpP2的PCR验证 A: Diagram of mutants construction. B: The verification of ΔClpP2 by PCR. |
|
|
| Primers name | Sequence (5′→3′) |
| Ha1-sllr0534-F | CAAATTACCCTTGGGGTGTC |
| Ha1-sll0534-R | GACTCTAGAGGATCCAAAATAGTAGCTGGGCCACA |
| Kana-F | GGATCCTCTAGAGTCGACCT |
| Kana-R | GATCTGATCCTTCAACTCA |
| Ha2-sll0534-F | TTGAAGGATCAGATCTGGACGCAGAGGACCCGGAA |
| Ha2-sll0534-R | CAGATGGGAGTCGTATTTGG |
将WT和ΔClpP2菌株接种到BG-11液体培养基中,分别置于正常(NC)、高光(HL)、高温(HT)条件下进行全日照振荡光合自养培养,每隔12 h测光密度OD730,进行120 h并绘制生长曲线。在正常条件下,经过120 h光合自养生长WT和ΔClpP2先后到达对数生长期(图 2A)。WT的OD730从0.01生长至2.26±0.015,24−96 h的倍增时间为18.17 h;ΔClpP2的OD730到达1.82±014,倍增时间为20.90 h。当面对高光或高温环境胁迫时,WT生长速度显著放缓,OD730从0.05分别增至0.73±0.46和0.51±0.000 77;ΔClpP2在这两种条件胁迫下OD730均未超过0.1,无法正常生长(图 2B、2C)。
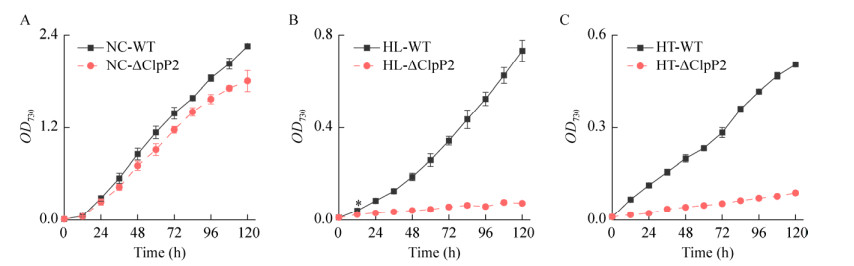
|
| 图 2 菌株在正常、高光和高温胁迫下的生长曲线 Figure 2 The gorwth curves under normal condition, high light stress and high temperature stress. A:30 ℃,40 μE. B:30 ℃,200 μE. C:42 ℃,40 μE A: 30 ℃, 40 μE. B: 30 ℃, 200 μE. C: 42 ℃, 40 μE. |
|
|
PSⅡ的最大光合效率(Fv/Fm)可以量化PSⅡ反应中心的最大光化学效率和PSⅡ电子转移情况,是PSⅡ光抑制或其他损伤的指标[18-19]。Fv/Fm测定值如图 3A所示,WT为0.63±0.007 2,显著高于ΔClpP2的0.58±0.016。ETR(Ⅱ)指在蓝细菌的稀释悬浮液中PSⅡ的电子传递速率[20];Y(Ⅱ)代表光化学反应吸收的能量部分,正常条件下范围是0−Fv/Fm[14]。快速光曲线结果如图 3B、3C所示,WT的ETR(Ⅱ)和Y(Ⅱ)的快速光曲线始终在ΔClpP2上方,与Fv/Fm结果有一致性,说明ΔClpP2菌株中PSⅡ受损,活性下降。
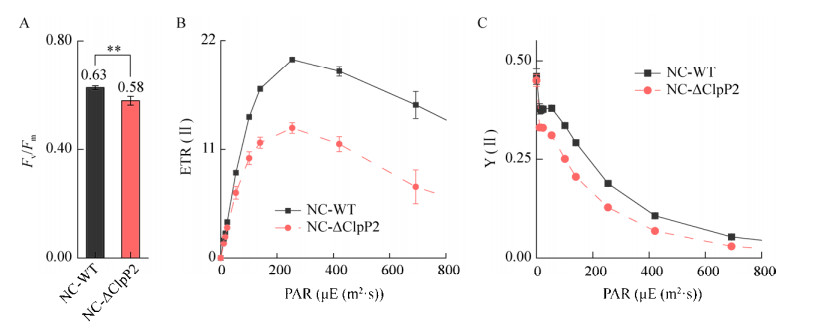
|
| 图 3 菌株的Fv/Fm与快速光曲线 Figure 3 Fv/Fm and rapid light curves of the strains. A:PSⅡ最大光化学量子产量(Fv/Fm),**: P < 0.01. B:PSⅡ真实电子传递速率[ETR(Ⅱ)]的快速光曲线. C:PSⅡ实际光合效率[Y(Ⅱ)]的快速光曲线 A: PSⅡ maximum photochemical quantum yield (Fv/Fm), **: P < 0.01. B: Rapid light curves of light-intensity dependence of the PSⅡ-mediated electron transport rate [ETR(Ⅱ)]. C: Rapid light curves of most measurements including quantum yield of PSⅡ [Y(Ⅱ)]. |
|
|
诱导动力学和光照后瞬态的典型叶绿素荧光轨迹,也称Post-illumination曲线如图 4A所示。在WT中光化光熄灭后荧光信号立即下降,经过约10 s荧光信号上升形成鼓包(图 4A WT-box),该过程中质体醌池在黑暗环境下发生氧化还原[12],PSⅠ中产生了光还原剂,光照后的这种荧光波动反映了NDH-依赖性活性,鼓包大小可以在一定程度上通过PSⅡ荧光反映围绕PSⅠ的环式电子传递[21-22]。在ΔClpP2中鼓包幅度减弱说明其环式电子传递受到影响但未完全消失(图 4A)。
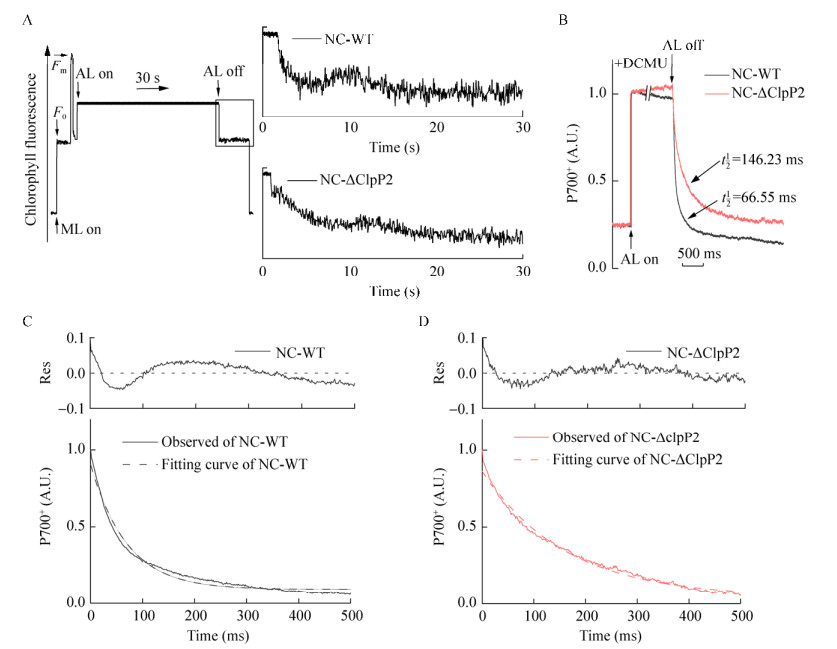
|
| 图 4 菌株的Post-illumination曲线与P700氧化还原动力曲线 Figure 4 The Post-illumination curves and P700 redox curves of strains. A:Post-illumination曲线. B:P700氧化还原动力曲线. C:WT的P700还原曲线. D:ΔClpP2的P700还原曲线 A: The curves of Post-illumination. The transient increase in chlorophyll fluorescence after termination of actinic light. B: Full redox curve of P700 of ΔClpP2 and WT. C : Reduction curve of P700 of WT. D: Reduction curve of P700 of ΔClpP2. |
|
|
P700是PSⅠ的反应中心,光照下通过细胞色素b6f复合物(Cytb6f)流向P700的电子流主要来自PSⅡ[23-24],作为一种PSⅡ抑制剂,DCMU抑制电子从PSⅡ上的醌向次级电子受体质醌的传递,将导致次级电子受体QA减少,可以切断蓝细菌中电子流向呼吸、流向环式电子传递途径[25]。DCMU存在时P700还原的电子主要来自NADH脱氢酶和PSⅠ周围的循环电子流[26-28]。在远红光诱导下P700会失去电子被瞬间氧化并稳定,关闭远红光P700开始被电子链上的电子还原,荧光信号逐渐降低,如图 4B所示。ΔClpP2的P700还原曲线比WT的下降更平缓。取还原曲线初始5 s信号进行衰减拟合,残差图显示拟合结果良好,见图 4C、4D。根据拟合结果计算P700还原半衰期,WT为(66.55±2.47) ms,ΔClpP2显著延长至(146.23±20.65) ms,是WT的2.20倍。Post-illumination曲线与P700还原曲线也具有一致性,表明ClpP2蛋白影响蓝细菌PSⅠ的环式电子转移速率。
2.3 ClpP2蛋白失活造成丰富的蛋白质表达变化借助iTRAQ方法可以监测蛋白质表达变化,工作流程如图 5所示。通过筛选(unique peptide≥1),最终6个实验组共有2 612个蛋白质被成功鉴定,这些蛋白约占集胞藻总基因组3 508个预测蛋白的74.46% (https://www.uniprot.org/proteomes/UP000011627),组学数据有较高的覆盖度。对所有蛋白信息进行主成分分析(principal components analysis, PCA),以检验生物学重复的重现性并初步了解样本之间的差异。PCA结果显示WT与ΔClpP2菌株的组别分别聚成两簇(图 6A),结果表明生物学重复良好,ΔClpP2中蛋白质表达情况发生了丰富的变化。
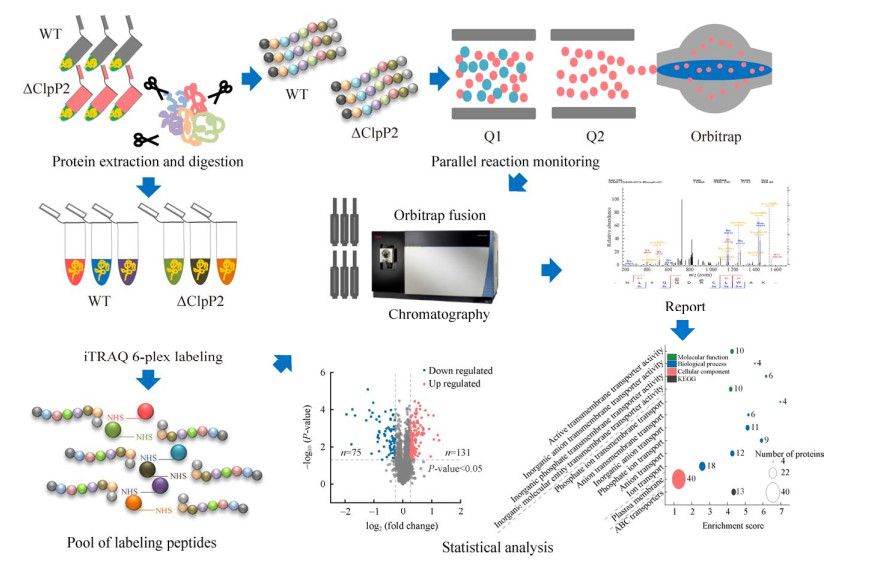
|
| 图 5 iTRAQ与PRM定量蛋白质组实验流程图 Figure 5 Proteomics experimental workflow. |
|
|
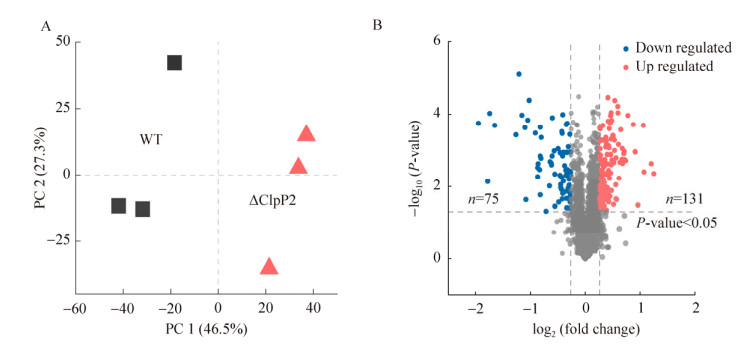
|
| 图 6 iTRAQ数据评估 Figure 6 Isobaric tags for relative and absolute quantitation (iTRAQ) data evaluation and subcellular localization. A:鉴定蛋白质的PCA分析. B:鉴定蛋白质的火山图 A: Principal components analysis (PCA) analysis of iTRAQ data. B: Volcano diagram of identified proteins. |
|
|
定义|log2[fold change(ΔClpP2/WT)]| > 0.263且P < 0.05为差异表达蛋白,共筛选出206个以WT为参考ΔClpP2菌株的差异表达蛋白,其中131个蛋白上调、75个下调,数据可视化为火山图,详见图 6B。上调的差异表达蛋白是下调的1.5倍,说明ClpP2蛋白异常导致更多的蛋白质在细胞中积累。这些上调差异表达蛋白是ClpP2蛋白的潜在底物群。表 2中选择性地列出一些已有注释的差异表达蛋白。
| Gene ID | Gene names | Fold change | Description | |
| iTRAQ-based | PRM | |||
| sll0247 | isiA | 0.653 | 0.001 | Iron-stress chlorophyll-binding protein, homologous to psbC (CP43) |
| sll0681 | pstC | 0.548 | 0.017 | Phosphate transport system permease protein PstC homolog |
| sll0534 | clpP2 | 0.466 | 0.020 | ATP-dependent Clp protease proteolytic subunit 2 |
| sll0683 | pstB1 | 0.299 | 0.021 | Phosphate transport ATP-binding protein PstB homolog |
| slr1250 | pstB3 | 0.748 | 0.150 | Phosphate transport ATP-binding protein PstB homolog |
| sll0684 | pstB2 | 0.416 | 0.158 | Phosphate transport ATP-binding protein PstB homolog |
| slr1672 | glpK | 0.551 | 0.264 | Glycerol kinase |
| sll0680 | pstS | 0.449 | 0.327 | Phosphate-binding periplasmic protein precursor (PBP) |
| sll0535 | clpX | 0.782 | 0.391 | ATP-dependent Clp protease ATPase subunit |
| slr1247 | pstS | 0.492 | 0.560 | Phosphate-binding periplasmic protein precursor (PBP) |
| sll0290 | ppk | 0.810 | 0.623 | Polyphosphate kinase |
| slr1853 | slr1853 | 0.658 | 0.789 | Carboxymuconolactone decarboxylase |
| slr0384 | slr0384 | 1.345 | 1.462 | Sulfoquinovosyldiacylglycerol biosynthesis protein SqdX |
| slr0549 | asd | 1.308 | 1.529 | Aspartate beta-semialdehyde dehydrogenese |
| sll0495 | asnS | 1.388 | 1.683 | Asparaginyl-tRNA synthetase |
| sll0900 | hisG | 1.243 | 1.734 | ATP phosphoribosyltransferase |
| sll1807 | rplX | 1.436 | 1.764 | 50S ribosomal protein L24 |
| sll0899 | glmU | 1.419 | 1.846 | UDP-N-acetylglucosamine pyrophosphorylase |
| sll0086 | sll0086 | 1.202 | 1.859 | Putative arsenical pump-driving ATPase |
| sll0502 | argS | 1.378 | 2.023 | Arginyl-tRNA-synthetase |
| sll0902 | argF | 1.345 | 2.209 | Ornithine carbamoyltransferase |
| sll0901 | purE | 1.319 | 2.209 | Phosphoribosylaminoimidazole carboxylase |
| slr0546 | trpC | 1.453 | 2.296 | Indole-3-glycerol phosphate synthase |
| slr1608 | slr1608 | 1.315 | 2.298 | Putative glucose dehydrogenase-B, periplasmic protein |
| sll1679 | hhoA | 1.248 | 2.407 | Periplasmic protease HhoA |
| sll0533 | tig | 1.257 | 2.728 | Trigger factor |
| slr0793 | slr0793 | 2.381 | 2.770 | Cation efflux system protein involved in nickel and cobalt tolerance |
| slr1331 | ymxG | 1.248 | 3.001 | Periplasmic processing protease |
| slr0544 | slr0544 | 1.270 | 3.050 | ATP-binding protein of ABC transporter |
| sll0897 | dnaJ1 | 1.627 | 3.273 | DnaJ protein, heat shock protein 40, molecular chaperone |
按照变化趋势分别对131个上调和75个下调差异表达蛋白进行GO分类,按照生物过程、细胞组分和分子功能注释成功,结果如图 7A所示。上调的差异表达蛋白具有结合(28.24%)、催化(28.24%)、转运(9.16%)活性,主要参与细胞的生理活动(28.24%)和代谢进程(20.61%),此外还参与细胞的应激反应、响应其他有机体、防御应答病毒等过程。在细胞的应激反应条目中有dfa4、isiA、tig、ruvC、ftsI、hslO、spoT蛋白,这些蛋白因ClpP2活性的缺失而堆积在细胞中,为ClpP2的潜在底物。ΔClpP2中PSⅡ的亚基psbY、psbL、psbH以及NADH脱氢酶的亚基ndhA、ndhB、ndhG也因ClpP2功能异常表达有所升高。
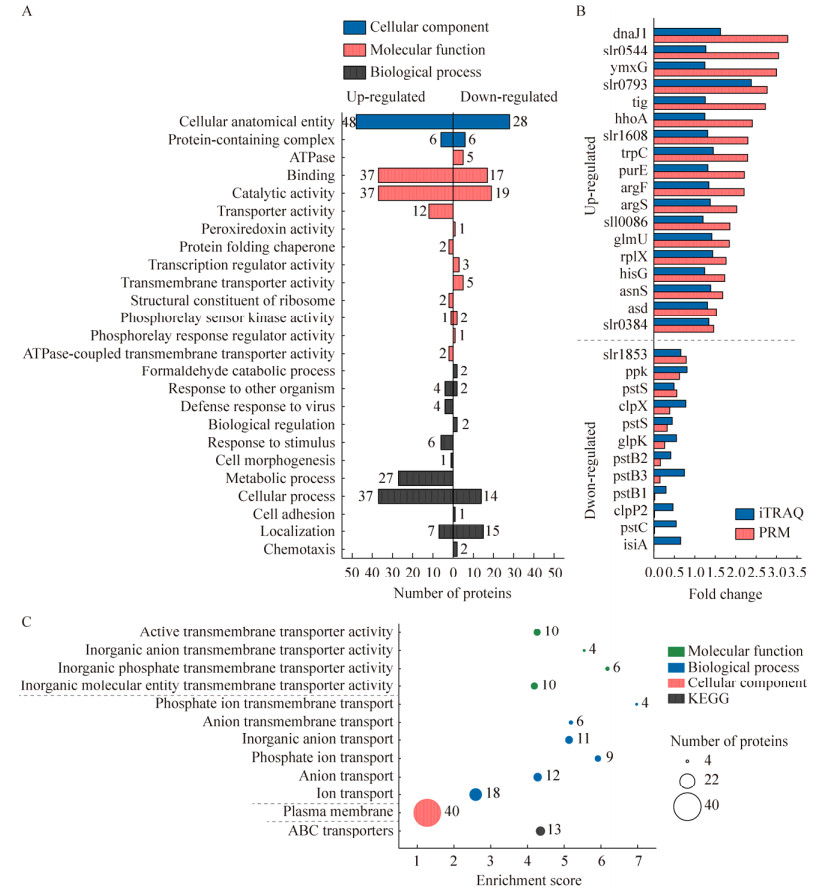
|
| 图 7 差异表达蛋白的功能分析与富集和PRM结果 Figure 7 Functional analysis and enrichenment for differentially expressed protein and the PRM results. A:GO分析细胞成分、分子功能和生物过程. B:GO和KEGG通路富集. C:将PRM的验证结果与iTRAQ进行对比 A: GO analysis for cellular component, molecular functions, and biological processes. B: GO and KEGG pathway enrichment. C: The verification results of parallel reaction monitoring (PRM) compared with iTRAQ. |
|
|
下调的差异表达蛋白除了具有催化(25.33%)、结合(22.67%)活性,还与ATP酶活性相关(6.7%),这些蛋白帮助细胞成分定位(20%)、参与细胞的生理活动(18.67%),也在生物调节、响应其他有机体、甲醛分解等过程中发挥作用。其中isiA蛋白参与铁离子的转运,ziaA蛋白与金属铜、锌、镉的转运密切相关[29-30]。
2.4.2 差异表达蛋白中的Clp家族蛋白尽管ΔClpP2菌株中ClpP2蛋白失活,在PRM和iTRAQ结果中仍能监测到降低表达的ClpP2信号,说明异常ClpP2蛋白肽段在突变株中有少量表达,但无法正常行使ClpP2蛋白功能(图 7C)。在iTRAQ数据中检测到ΔClpP2中显著降低表达的ClpX (sll0535)蛋白(图 7C),同时鉴定到其他Clp家族蛋白ClpP1 (sll0542)、ClpP3 (sll0165)和ClpC (sll0020)蛋白正常表达。
2.4.3 GO富集与KEGG富集分析206个差异表达蛋白的GO和KEGG富集分析在STRING 11.5 (https://cn.string-db.org/)的“Proteins with Values/Ranks”模块下进行,该模块的FDR stringency设置为5%。这一结果可以直接指出所有差异表达蛋白整体功能富集特征,并且根据富集结果重点关注高富集分数的条目所涉及到的蛋白及其注释生物功能。如图 7C所示,差异表达蛋白在各种跨膜转运蛋白活性密切相关的4项功能中显著富集,得分最高的是无机磷酸盐跨膜转运蛋白活性条目(6.18);有7项富集条目与各种分子和离子转运的生物进程相关,磷酸盐离子跨膜转运(6.97)得分最高。这些蛋白显著富集在质膜上,包含40个蛋白。差异表达蛋白的KEGG富集分析显著指向ABC转运途径,富集分数为4.35,包含Pst1系统中的pstS (sll0680)、pstC (sll0681)、pstB1、pstB2蛋白和Pst2系统中的pstS (slr1247)、pstC (slr1248)、pstB3蛋白。
2.5 PRM验证差异表达蛋白利用PRM技术进一步评估iTRAQ的定量数据,选择unique peptide > 1的差异表达蛋白进行验证,最终有34个差异表达蛋白验证成功(P < 0.01)。就结果而言,我们在iTRAQ和PRM中并未得出完全一致的结论:有4个蛋白在iTRAQ和PRM表现出不同的表达变化趋势,分别是slr1485、sll0654、ssl2559和slr1512;余下30个蛋白表达趋势一致,占总鉴定成功蛋白的88.24%,其中12个蛋白下调、18个蛋白上调。鉴定出的表达变化趋势相同的蛋白中,PRM定量结果相较于iTRAQ显示出更为剧烈的蛋白表达水平波动,详见图 7C。总体而言PRM验证结果表明iTRAQ定量结果可信度较高。
3 讨论与结论对于大部分细菌而言,ClpP蛋白不是生长必需蛋白质[31-32],如大肠杆菌中clpP的丢失不会产生明显的表型变化;枯草芽孢杆菌会因clpP的缺失而形态改变;在聚球藻PCC 7002中,ClpP1蛋白的失活会拉长细胞形态,同时明显降低菌株的生长速度。集胞藻ΔClpP2的成功构建(图 1B)同样证明在集胞藻中ClpP2蛋白质不是生长必需,在本研究中ΔClpP2可以光合自养至对数期,生长速度稍有减缓(图 1C),这与聚球藻PCC 7002的ClpP2突变株生长情况[33]相同。在面对高光或高温胁迫时WT的生长显著减缓说明集胞藻具有响应胁迫的机制(图 2),ΔClpP2无法正常生长则表明ClpP2蛋白在应对环境胁迫时不可或缺。Sokolenko等[7]研究指出ClpP2蛋白异常后集胞藻无法光合自养,必须依赖外部提供碳源,分析认为本研究与Sokolenko研究的差异是clpP2基因被替换序列不同所致。另外,集胞藻ClpP1敲除株构建失败与Panichkin等[17]的研究结果一致,提示ClpP1蛋白在集胞藻的生长过程中起不可或缺的作用。
集胞藻中存在两个Pst系统,当磷离子充足时Pst1系统蛋白表达量升高,同时增强细胞对磷离子的吸收;在磷离子缺陷条件下Pst2系统蛋白将显著上调以缓解磷胁迫[34]。ΔClpP2的Pst1与Pst2系统蛋白表达降低,说明因ClpP2蛋白异常菌株对磷离子的吸收和转运能力下降,进而影响集胞藻的各项生理活动。通过PRM验证检测到isiA蛋白表达量几乎为零(图 7C),isiA蛋白可以向PSⅠ光合反应中心传递光能,其下调将降低光合系统活性[35];ABC转运蛋白通路被显著富集,这些结果指向ClpP2蛋白的失活对光合系统带来的负面影响。结合表型实验,ΔClpP2的光合系统虽然受到损伤,但仍然可以完成光合作用并通过光合自养满足自身的生长,进一步说明ClpP2蛋白对集胞藻生长的影响是有限的。我们提出ClpP2蛋白通过影响物质的转运,特别是磷离子、铁离子、镉离子等的转运影响光合系统。另外,psbH可以稳定PSⅡ复合物中电子转移过程[36],ndhB在应对光抑制稳定PSⅠ循环电子传递速率上发挥重要作用[37],提示PSⅡ和NADH脱氢酶的部分亚基上调是抵御ClpP2蛋白失活带来的光损伤,这一猜测可以佐证Panichkin等[17]提出的ClpP2蛋白参与保护集胞藻光抑制的观点。
在念株菌PCC7942的Clp蛋白复合物中存在2个不同的Clp蛋白水解核心,即ClpP1与ClpP2形成多聚体再同ClpX结合形成蛋白酶复合物[8]。结合两种组学数据结果,ΔClpP2中ClpX蛋白表达显著降低,ClpP1蛋白表达无异常,我们认为ClpP2蛋白酶的分子伴侣是ClpX蛋白而非ClpC蛋白,即集胞藻中存在ClpXP2复合物。ΔClpP2中ClpP1蛋白的表达不受ClpP2失活的影响,初步认为集胞藻中不存在多核心的ClpXP1/P2复合物,但仍需进一步验证。
我们探究了ClpP2蛋白的失活对集胞藻带来的生长和光合作用的变化,并用蛋白质组学的方法获得差异表达蛋白,并通过结合生理表型数分析ClpP2蛋白直接影响集胞藻的离子转运,很可能因此影响其光合系统。下一步的工作将针对底物群与ClpP2进行相互作用筛选ClpP2蛋白的底物,并探索ClpP2蛋白调控的分子机制。
本研究筛选了WT和ΔClpP2的差异表达蛋白并进行了GO、KEGG分析,结合生长实验、光合参数阐述了ClpP2蛋白对集胞藻生长、光合系统的影响,揭示了ClpP2的分子伴侣和潜在生物学功能并提供了底物群。本研究填补了集胞藻中ClpP2蛋白研究的空白,为ClpP蛋白的研究提供了新的视角和强有力的数据支持,也为ClpP2蛋白的进一步功能研究和分子机制研究提供了重要基础。
| [1] |
NISHIMURA K, van WIJK KJ. Organization, function and substrates of the essential Clp protease system in plastids[J]. Biochimica et Biophysica Acta, 2015, 1847(9): 915-930. DOI:10.1016/j.bbabio.2014.11.012 |
| [2] |
REI LIAO, van WIJK KJ. Discovery of AAA+ protease substrates through trapping approaches[J]. Trends in Biochemical Sciences, 2019, 44(6): 528-545. DOI:10.1016/j.tibs.2018.12.006 |
| [3] |
SAUER RT, BAKER TA. AAA+ proteases: ATP-fueled machines of protein destruction[J]. Annual Review of Biochemistry, 2011, 80: 587-612. DOI:10.1146/annurev-biochem-060408-172623 |
| [4] |
BOUCHNAK I, van WIJK KJ. Structure, function, and substrates of Clp AAA+ protease systems in cyanobacteria, plastids, and apicoplasts: a comparative analysis[J]. Journal of Biological Chemistry, 2021, 296: 100338. DOI:10.1016/j.jbc.2021.100338 |
| [5] |
MABANGLO MF, HOURY WA. Recent structural insights into the mechanism of ClpP protease regulation by AAA+ chaperones and small molecules[J]. Journal of Biological Chemistry, 2022, 298(5): 101781. DOI:10.1016/j.jbc.2022.101781 |
| [6] |
KIM S, ZUROMSKI KL, BELL TA, SAUER RT. ClpAP proteolysis does not require rotation of the ClpA unfoldase relative to ClpP[J]. eLife, 2020, 9. |
| [7] |
SOKOLENKO A, POJIDAEVA E, ZINCHENKO V, PANICHKIN V, GLASER VM, HERRMANN RG, SHESTAKOV SV. The gene complement for proteolysis in the cyanobacterium Synechocystis sp. PCC 6803 and Arabidopsis thaliana chloroplasts[J]. Current Genetics, 2002, 41(5): 291-310. DOI:10.1007/s00294-002-0309-8 |
| [8] |
STANNE TM, POJIDAEVA E, ANDERSSON FI, CLARKE AK. Distinctive types of ATP-dependent Clp proteases in cyanobacteria[J]. Journal of Biological Chemistry, 2007, 282(19): 14394-14402. DOI:10.1074/jbc.M700275200 |
| [9] |
HAVAUX M, GUEDENEY G, HAGEMANN M, YEREMENKON, MATTHIJS HCP, JEANJEAN R. The chlorophyll-binding protein IsiA is inducible by high light and protects the cyanobacterium Synechocystis PCC6803 from photooxidative stress[J]. FEBS Letters, 2005, 579(11): 2289-2293. DOI:10.1016/j.febslet.2005.03.021 |
| [10] |
CHENG JF, CHEN GY, SHEN YG. Relational analysis of leaf characteristics and photosynthetic capacities of plants[J]. 中国生态农业学报, 2012, 20(4): 466-473. 程建峰, 陈根云, 沈允钢. 植物叶片特征与光合性能的关系[J]. 中国生态农业学报, 2012, 20(4): 466-473. |
| [11] |
SHIKANAI T, ENDO T, HASHIMOTO T, YAMADA Y, ASADA K, YOKOTA A. Directed disruption of the tobacco ndhB gene impairs cyclic electron flow around photosystem I[J]. Proceedings of National Academy of Sciences of the United States of America, 1998, 95(16): 9705-9709. DOI:10.1073/pnas.95.16.9705 |
| [12] |
GOTOH E, MATSUMOTO M, OGAWA K, KOBAYASHI Y, TSUYAMA M. A qualitative analysis of the regulation of cyclic electron flow around photosystem I from the post-illumination chlorophyll fluorescence transient in Arabidopsis: a new platform for the in vivo investigation of the chloroplast redox state[J]. Photosynthesis Research, 2010, 103(2): 111-123. DOI:10.1007/s11120-009-9525-0 |
| [13] |
GENTY B, BRIANTAIS JM, BAKER NR. The relationship between the quantum yield of photosynthetic electron transport and quenching of chlorophyll fluorescence[J]. Biochimica et Biophysica Acta, 1989, 990(1): 87-92. DOI:10.1016/S0304-4165(89)80016-9 |
| [14] |
KLUGHAMMER C, SCHREIBER U. Complementary PS Ⅱ quantum yields calculated from simple fluorescence parameters measured by PAM fluorometry and the Saturation Pulse method[J]. PAM Application Notes, 2008. |
| [15] |
LIN XH, YANG MK, LIU X, CHENG ZY, GE F. Characterization of lysine monomethylome and methyltransferase in model cyanobacterium Synechocystis sp. PCC 6803[J]. Genomics, Proteomics & Bioinformatics, 2020, 18(3): 289-304. |
| [16] |
YANG MK, YANG YH, CHEN Z, ZHANG J, LIN Y, WANG Y, XIONG Q, LI T, GE F, BRYANT DA, ZHAO JD. Proteogenomic analysis and global discovery of posttranslational modifications in prokaryotes[J]. Proceedings of National Academy of Sciences of the United States of America, 2014, 111(52): E5633-E5642. |
| [17] |
PANICHKIN VB, GLAZER VM, ZINCHENKO VV, SOKOLENKO A; HERRMANN RG, SHESTAKOV SV. clpP2 gene encoding peptidase in cyanobacteria Synechocystis sp. PCC 6803 controls the sensitivity of cells to photoinhibition[J]. Izvestiia Akademii Nauk Seriia Biologicheskaia, 2001(3): 312-317. |
| [18] |
BJÖRKMAN O, DEMMIG B. Photon yield of O2 evolution and chlorophyll fluorescence characteristics at 77 K among vascular plants of diverse origins[J]. Planta, 1987, 170(4): 489-504. DOI:10.1007/BF00402983 |
| [19] |
ROHÁČEK K, SOUKUPOVÁ J, BARTÁK M. Chlorophyll fluorescence: a wonderful tool to study plant physiology and plant stress[J]. Plant Cell Compartments-Selected Topics, 2008, 41-104. |
| [20] |
SCHREIBER U, KLUGHAMMER C, KOLBOWSKI J. High-end chlorophyll fluorescence analysis with the MULTI-COLOR-PAM. I. Various light qualities and their applications[J]. PAM Application Notes, 2011, 1: 1-21. |
| [21] |
DENG Y, YE JY, MI HL. Effects of low CO2 on NAD(P)H dehydrogenase, a mediator of cyclic electron transport around photosystem I in the cyanobacterium Synechocystis PCC6803[J]. Plant Cell Physiology, 2003, 44(5): 534-540. DOI:10.1093/pcp/pcg067 |
| [22] |
XU M, LÜ J, FU PC, Mi HL. Oscillation kinetics of post-illumination increase in chl fluorescence in cyanobacterium Synechocystis PCC 6803[J]. Frontiers in Plant Science, 2016, 7: 108. |
| [23] |
YU L, ZHAO J, MUHLENHOFF U, GOLBECK JH. PsaE is required for in vivo cyclic electron flow around photosystem I in the cyanobacterium Synechococcus sp. PCC 7002[J]. Plant Physiology, 1993, 103(1): 171-180. DOI:10.1104/pp.103.1.171 |
| [24] |
HUANG CH, YUAN XL, ZHAO JD, BRYANT DA. Kinetic analyses of state transitions of the cyanobacterium Synechococcus sp. PCC 7002 and its mutant strains impaired in electron transport[J]. Biochimica et Biophysica Acta, 2003, 1607(2): 121-130. |
| [25] |
FEILKE K, AJLANI G, KRIEGER-LISZKAY A. Overexpression of plastid terminal oxidase in Synechocystis sp. PCC 6803 alters cellular redox state[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2017, 372(1730). |
| [26] |
HOLLAND SC, ARTIER J, MILLER NT, CANO M, YU JP, GHIRARDI ML, BURNAPA RL. Impacts of genetically engineered alterations in carbon sink pathways on photosynthetic performance[J]. Algal Research, 2016, 20: 87-99. DOI:10.1016/j.algal.2016.09.021 |
| [27] |
MUNEKAGE Y, HASHIMOTO M, MIYAKE C, TOMIZAWA KI, ENDO T, TASAKA M, SHIKANAI T. Cyclic electron flow around photosystem I is essential for photosynthesis[J]. Nature, 2004, 429(6991): 579-582. |
| [28] |
KUSAMA S, MIYAKE C, NAKANISHI S, SHIMAKAWA G. Dissection of respiratory and cyclic electron transport in Synechocystis sp. PCC 6803[J]. Journal of Plant Research, 2022, 135(4): 555-564. |
| [29] |
BADARAU A, DENNISON C. Thermodynamics of copper and zinc distribution in the cyanobacterium Synechocystis PCC 6803[J]. Proceedings of National Academy of Sciences of the United States of America, 2011, 108(32): 13007-13012. |
| [30] |
TOTTEY S, PATTERSON CJ, BANCI L, BERTINI I, FELLI IC, PAVELKOVA A, DAINTY SJ, PERNIL R, WALDRON KJ, FOSTER AW, ROBINSON NJ. Cyanobacterial metallochaperone inhibits deleterious side reactions of copper[J]. Proceedings of National Academy of Sciences of the United States of America, 2012, 109(1): 95-100. |
| [31] |
TRENTINI DB, SUSKIEWICZ MJ, HEUCK A, KURZBAUER R, DESZCZ L, MECHTLER K, CLAUSEN T. Arginine phosphorylation marks proteins for degradation by a Clp protease[J]. Nature, 2016, 539(7627): 48-53. |
| [32] |
KIRSCH VC, FETZER C, SIEBER SA. Global inventory of ClpP- and ClpX-regulated proteins in Staphylococcus aureus[J]. Journal of Proteome Research, 2021, 20(1): 867-879. |
| [33] |
IMAI K, KITAYAMA Y, KONDO T. Elucidation of the role of clp protease components in circadian rhythm by genetic deletion and overexpression in cyanobacteria[J]. Journal of Bacteriology, 2013, 195(19): 4517-4526. |
| [34] |
BURUT-ARCHANAI S, EATON-RYE JJ, INCHAROENSAKDI A. Na+-stimulated phosphate uptake system in Synechocystis sp. PCC 6803 with Pst1 as a main transporter[J]. BMC Microbiology, 2011, 11: 225. |
| [35] |
ANDRIZHIYEVSKAYA EG, SCHWABE TME, GERMANO M, D'HAENE S, KRUIP J, van GRONDELLE R, DEKKER JP. Spectroscopic properties of PSI-IsiA super complexes from the cyanobacterium Synechococcus PCC 7942[J]. Biochimica et Biophysica Acta, 2002, 1556(2/3): 265-272. |
| [36] |
KOMENDA J, LUPÍNKOVÁ L, KOPECKÝ J. Absence of the psbH gene product destabilizes photosystem Ⅱcomplex and bicarbonate binding on its acceptor side in Synechocystis PCC 6803[J]. European Journal of Biochemistry, 2002, 269(2): 610-619. |
| [37] |
THOMAS DJ, THOMAS J, YOUDERIAN PA, HERBERT SK. Photoinhibition and light-induced cyclic electron transport in ndhB– and psaE– mutants of Synechocystis sp. PCC 6803[J]. Plant and Cell Physiology, 2001, 42(8): 803-812. |
 2023, Vol. 50
2023, Vol. 50




