扩展功能
文章信息
- 徐冬梅, 赵学金
- XU Dongmei, ZHAO Xuejin
- 基于CRISPR/Cas系统的天然产物生物合成基因簇克隆和编辑技术
- Cloning and refactoring of natural product biosynthetic gene clusters based on CRISPR/Cas system
- 微生物学通报, 2022, 49(9): 3933-3944
- Microbiology China, 2022, 49(9): 3933-3944
- DOI: 10.13344/j.microbiol.china.220106
-
文章历史
- 收稿日期: 2022-01-27
- 接受日期: 2022-04-25
- 网络首发日期: 2022-05-16
2. 中国科学院微生物研究所微生物生理与代谢工程重点实验室, 北京 100101
2. Key Laboratory of Microbial Physiological and Metabolic Engineering, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
随着基因组测序技术的快速发展,生物信息学分析揭示了微生物基因组中存在大量功能未知的生物合成基因簇,这些基因簇编码了潜在新型化学结构的天然产物[1]。然而绝大部分基因簇在常规条件下都沉默或未显著表达。因此,如何激活沉默基因簇的表达已成为挖掘天然产物的关键问题。天然产物基因簇沉默的生理学原因是宿主细胞仅仅需要这些产物帮助其应对恶劣环境因子和竞争者,正常实验室条件下则被严谨地抑制[2]。菌体生长、代谢、外界营养物质及环境压力等因素通过调控菌体内复杂的网络而控制次级代谢产物的合成。尽管多种基因簇激活方法已被开发,但仍然存在成功率低、发现新型分子少、杂分子干扰大、机制不清楚或遗传操作难等瓶颈问题[3-4]。如经典混菌培养刺激方法就经常重复地发现一些已有的药物分子;稀有金属等环境刺激方法则激活基因簇的随机性很强,成功率较低;物理化学诱变、核糖体工程等方法因改变了细胞全局的表达谱而产生太多杂分子,难以分析和检测,此外也存在一定的随机性;过表达正调控基因或敲除负调控基因也是目前激活沉默基因簇的有效手段,但因为很多工业菌种遗传操作难、生长速度慢等因素也不利于对菌种进行遗传改造[2]。随着对链霉菌(60%抗生素药物的产生菌)基因簇表达调控机制的深入研究,研究者意识到天然产物的生物合成受到严谨的调控。其不仅被多种全局调控因子控制,而且层次性地受到多种途径特异因子的激活和抑制[2]。由此可见,在原始菌株中优化基因簇的表达受到诸多因素制约。因此,利用遗传背景清晰、遗传操作容易且不受内源调控网络影响的模式生物(如白色链霉菌或天蓝色链霉菌)作为宿主进行天然产物基因簇的激活和异源表达,成为挖掘新化合物及高效合成目标产物的普适有效的方法[5]。
基因簇的异源克隆及在异源模式菌株中被激活是实现基因簇成功表达的关键,但是存在不少困难。比如,链霉菌基因组的高GC含量和聚酮类化合物(polyketides)基因簇存在重复片段等问题,导致基于PCR的长片段克隆比较困难,并且基于同源重组的Gibson组装效率也较低[6-7];应用传统构建基因组文库方法来筛选目的基因簇耗时又耗力[8-9];RecET重组技术只适用于50 kb以下的基因簇克隆[10]。此外,对基因簇进行编辑或重构是激活基因簇并成功异源表达的必要补充,但也面临着多靶点编辑大型基因簇的技术问题。虽然应用λ噬菌体同源重组系统[11]、Gibson组装、RecET等方法可以对大型基因簇进行操作,但是对多个位点同时编辑较难。随着合成生物学技术的发展,尤其是CRIPSR/Cas技术的兴起,研究者开发了一系列基于CRISPR/Cas的生物合成基因簇克隆和编辑的方法,这大大提高了基因簇激活和优化的效率。
1 CRISPR/Cas技术用于基因簇的直接克隆CRISPR/Cas系统是来源于细菌或古细菌的一种获得性免疫系统,通常该系统主要由CRISPR RNA (crRNA)和Cas蛋白组成,crRNA与靶序列互补,引导Cas核酸内切酶对靶序列进行特异性识别和切割。CRISPR/Cas系统具有设计简单、可编程及易操作等优点[12]。目前已开发了CRISPR/Cas9、CRISPR/Cas12等多种CRISPR系统,被广泛应用于基因编辑、核酸检测及基因调控等领域[13-15]。为了实现目标基因簇的靶向克隆,研究者开发了多种基于CRISPR/Cas的基因簇体外一步克隆法,解决了大型基因簇片段因酶切位点过多或PCR不易扩增导致的难组装问题,其核心策略是应用RNA介导的CRISPR/Cas核酸内切酶可以精确地识别和切割任意目标基因簇的两侧,通过transformation-associated recombination (TAR)酵母转化偶联重组/Gibson等组装方法将切割的基因簇与目标载体连接,从而达到一步定向克隆的目的。
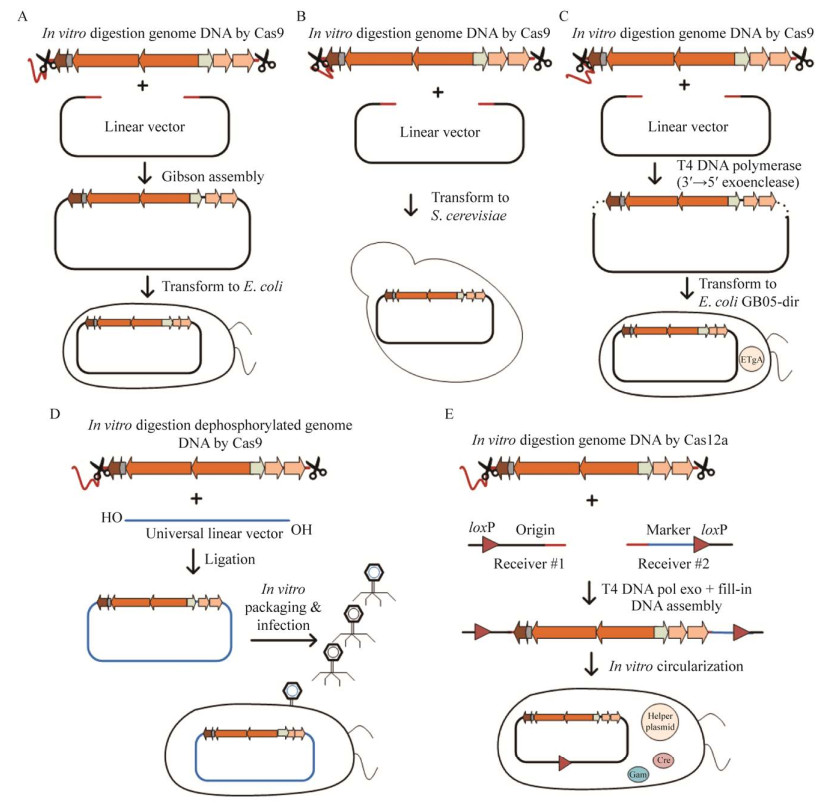
1.1 基于CRISPR/Cas9和Gibson组装的基因簇克隆CRISPR/Cas9系统组成简单,只包括具有核酸酶功能的Cas9蛋白及靶向DNA的tracrRNA和crRNA,二者可以嵌合成一条单链向导RNA (single guide RNA,sgRNA)。CRISPR/Cas9系统识别前间区序列邻近基序(protospacer adjacent motif,PAM)序列NGG,并对其上游序列进行切割[12]。2015年,Jiang等利用CRISPR/Cas9和Gibson组装开发了一种在体外将细菌基因组中特定的基因簇直接克隆的方法(Cas9-assisted targeting of chromosome segments,CATCH)[16] (图 1A)。与构建基因组文库获得基因簇的传统方法相比,该方法快速、简单且效率高。其基本原理是利用CRISPR/Cas核酸内切酶精确地切割目标基因簇片段,然后通过Gibson组装的方法将钓取的基因簇片段与带有基因簇两端同源臂的载体连接,获得含有目标基因簇的重组质粒。研究者利用该方法成功克隆了不同长度(50−150 kb)的大肠杆菌基因组片段,以及枯草芽孢杆菌来源的bacillaene生物合成基因簇和链霉菌来源的金霉素(chlortetracycline)、杰多霉素(jadomycin)生物合成基因簇[16]。研究发现,随着目标基因簇长度增加,克隆效率逐渐降低。比如,获得32 kb金霉素基因簇的阳性率约为90%,而获得78 kb bacillaene基因簇的阳性率仅为12%[16]。利用上述方法,Wang等[17]在工业底盘菌——龟裂链霉菌宿主中,实现了金霉素基因簇的高效异源表达。Li等[18]运用CATCH方法克隆了多杀菌素(spinosad)的生物合成基因簇,并通过基因簇串联倍增的方法实现了多杀菌素在天蓝色链霉菌底盘中的高效异源表达。
|
| 图 1 CRISPR/Cas系统介导的基因簇直接克隆的方法[16, 25] Figure 1 Direct gene cluster cloning methods mediated by CRISPR/Cas[16, 25]. A:基于CRISPR/Cas9和Gibson组装的基因簇克隆技术示意图;B:基于CRISPR/Cas9和TAR的基因簇克隆技术示意图;C:基于CRISPR/Cas9和ExoCET的基因簇克隆技术示意图;D:基于CRISPR/Cas9和λ体外包装系统的基因簇克隆技术示意图;E:基于CRISPR/Cas12a和Cre-loxP的基因簇克隆技术示意图 A: The diagram of gene cluster cloning method mediated by CRISPR/Cas and Gibson assembly; B: The diagram of gene cluster cloning method mediated by CRISPR/Cas and TAR; C: The diagram of gene cluster cloning method mediated by CRISPR/Cas and ExoCET; D: The diagram of gene cluster cloning method mediated by CRISPR/Cas and in vitro λ packaging system; E: The diagram of gene cluster cloning method mediated by CRISPR/Cas12a and Cre-loxP. |
|
|
TAR是一种通过酵母同源重组系统、快速地克隆基因组片段的技术[19],其基本原理是将线性化的TAR克隆载体与酶切后的基因组DNA片段同时转入酵母细胞,利用酵母细胞高效的同源重组系统,使目标基因组DNA片段与TAR载体片段发生重组连接,从而实现对目标基因簇的克隆。然而,传统的TAR是利用限制性内切酶来切割基因组DNA。此方法存在克隆特异性差、效率及准确性低等问题。2015年,Lee等报道了CRISPR/Cas9介导的TAR克隆方法(CRISPR/Cas9-mediated TAR cloning),该方法不仅实现了特定基因组DNA片段的靶向克隆,而且使克隆阳性率从5%提高到32%左右[20] (图 1B)。该方法具有克隆片段尺度大的优点,克隆长度理论上可长达250 kb,但由于需要应用遗传背景复杂的酵母菌细胞,所以克隆过程较为烦琐,用时较长。2017年,Meng等应用该技术成功克隆了pristinamycin I基因簇,并将其转入改造过的宿主细胞,将目标产物的产量提高到原来的2.4倍,成功实现了基因簇高效异源表达[21]。
1.3 基于CRISPR/Cas9和ExoCET的基因簇克隆核酸外切酶与RecET联合重组(Exonuclease combined with RecET recombination,ExoCET)是将T4 DNA聚合酶介导的体外同源重组与RecET介导的体内同源重组相结合开发的基因组大片段直接克隆技术[22] (图 1C)。该技术是由Fu等在RecET克隆技术基础上进一步开发而成[10],其基本原理是利用T4 DNA聚合酶的3′→5′外切酶活性,先在体外将线性化的克隆载体与酶切后的基因组片段退火连接,从而提高其共同进入同一个大肠杆菌细胞的概率,再利用大肠杆菌体内RecET重组系统介导的线线同源重组反应完成目标基因组DNA片段与载体片段重组连接,从而实现对目标基因簇的克隆;将CRISPR/Cas9与ExoCET结合进一步增加了该方法的优势,不仅可以实现基因簇的精确克隆,还能降低假阳性率。Wang等应用CRISPR/Cas9与ExoCET成功克隆了106 kb的盐霉素(salinomycin)生物合成基因簇,克隆正确率为4%[22]。该方法尽管可以实现50 kb以上基因组片段的直接克隆,但其克隆效率有待进一步提高[22]。
1.4 基于CRISPR/Cas9和λ体外包装系统的基因簇克隆λ噬菌体载体能够容纳50 kb以下外源DNA片段的插入,常被应用于构建基因组DNA文库。其基本原理为目标DNA连接至噬菌体载体,随后应用λ体外包装系统包装形成噬菌体颗粒,转导至大肠杆菌细胞,筛选阳性克隆。2019年,Tao等应用CRISPR/Cas9和λ噬菌体体外包装系统建立了一种基因簇的靶向克隆方法[23] (图 1D)。该方法基本过程为:(1) EcoR V酶切获得线性化通用载体;(2) 为了降低非特异序列连接,将提取的基因组DNA去磷酸化处理;(3) Cas9酶切基因组DNA获得目标基因簇;(4) T4 DNA连接酶将目标基因簇片段与通用载体连接;(5) λ体外包装系统将连接产物包装形成噬菌体颗粒,并转导至大肠杆菌细胞中,通过筛选获得阳性克隆。Tao等利用该方法成功克隆了27.4 kb的Tü3010 (stu)基因簇及40.7 kb的sisomicin基因簇,克隆效率在18%−54%之间[23]。通用载体的使用及λ噬菌体颗粒较高的转导能力使得该方法简单、高效。然而因为λ噬菌体载体载量有限,导致该方法仅限于中等长度基因簇的克隆。
1.5 基于CRISPR/Cas12a和Cre-loxP的基因簇克隆Cas12a核酸酶属于Ⅱ类type Ⅴ CRISPR系统。与Cas9相比,Cas12a更简单、更精准、脱靶效应更低。Cas12a识别富含T的PAM序列TTTN,只需要一个crRNA即可实现对目标基因的切割,并且Cas12a切割DNA后形成黏性末端,更便于DNA组装[24]。2021年,Enghiad等建立了一种基于Cas12a和Cre-loxP的快速基因簇克隆方法CAPTURE[25] (图 1E)。该方法基本过程为:(1) 构建2个载体片段,其中一个带有基因簇一端的同源臂及载体起始位点和loxP位点,另外一个片段带有基因簇另一端同源臂及筛选标记和loxP位点;(2) CRISPR/Cas12a酶切获得目标基因簇;(3) T4 DNA聚合酶处理基因簇片段及带有同源臂的载体片段,使得基因簇与载体片段完成组装,从而获得两端带有loxP位点的线性组装产物;(4) 将组装产物转化表达Cre重组酶的大肠杆菌细胞,在体内完成基因簇和载体的线性组装产物的环化。研究者应用该方法克隆了43个不同长度(10−113 kb)的基因簇,结果显示克隆长度为50−99 kb的基因簇效率高达100%,当基因簇长度增至100 kb后克隆效率为95%左右;研究者随后将获得的43个基因簇全部进行异源表达,最终获得高达15种具有新型结构的天然产物[25]。由此可见,CAPTURE方法十分高效,并展现出了高通量挖掘活性天然产物的潜力。
现将几种基于CRISPR/Cas开发的基因簇克隆技术特点、优缺点及成本进行总结,详见表 1。
| Methods | Principle | Maximal size of cloning (kb) | Advantage | Disadvantage | Cost |
| CATCH[16] | In vitro CRISPR/Cas9 digestion and Gibson assembly | 150 | Able to clone BGCs larger than 100 kb. BGCs can be cloned in 1 week | Low cloning efficiency (21.6% for 100 kb genome fragment). Low cloning efficiency for BGCs from high AT-genomes | High cost (Cas9 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation, Gibson Assembly Cloning Kit, E. coli commercial electrocompetent cells et al) |
| CRISPR/Cas9- mediated TAR cloning[20] | In vitro CRISPR/Cas9 digestion and yeast homologous recombination | 55 | Broadly applicable across a wide range of fragment sizes and genome complexities | Must use yeast, technically challenging, time consuming (more than 1 week). Low cloning efficiency (32% for 55 kb genome fragment) | Low cost (Cas9 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation, yeast transformation kit et al) |
| CRISPR/Cas9- ExoCET[22] | In vitro CRISPR/Cas9 digestion and RecET mediated homologous recombination | 106 | Broadly applicable across a wide range of fragment sizes and genome complexities. BGCs can be cloned in 1 week | Low cloning efficiency (4% for 106 kb BGC) | Low cost (Cas9 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation, T4 DNA polymerase et al) |
| In vitro packaging mediated approach[23] | In vitro CRISPR/Cas9 digestion and in vitro λ packaging system | 40.7 | Rapid and convenient, BGCs can be cloned in 1 week. 54% cloning efficiency for 40.7 kb target BGC | Relies on in vitro λ packaging and ligation, technically challenging. Difficult to clone BGCs larger than 50 kb | High cost (Cas9 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation, MaxPlax λ Packaging Extracts kit, E. coli commercial electrocompetent cells, T4 DNA ligase, EcoR V et al) |
| CAPTURE[25] | In vitro CRISPR/Cas12a digestion and in vivo Cre-lox recombination | 113 | Simple and robust, BGCs can be cloned in 3−4 days. About 95% cloning efficiency for 100 kb BGCs | Cas12a recognizes T-rich PAMs, which limits its application in high GC genomic regions | Low cost (Cas12 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation, T4 DNA polymerase, E. coli DNA ligase et al) |
克隆获得基因簇后,对其进行异源表达是获取活性天然产物的有效方法。该方法一方面因为异源宿主可能缺少导致基因簇沉默的严谨的调控网络而有利于基因簇的激活,另一方面异源宿主可能无法识别基因簇自身的调控元件,或缺少天然产物合成的前体、辅因子等因素,导致基因簇无法成功表达或者表达量极低。因此,进一步对基因簇进行编辑重构是基因簇激活和高效表达的必要补充之一。
基因簇重组质粒具有片段较大、限制性酶切位点不单一等问题,使得在基因簇的特定位置上进行片段插入、敲除及替换等精准编辑比较困难,这也是工程化改造基因簇的一个瓶颈。然而,CRISPR/Cas技术本身具有高效、精准、多靶点的靶向编辑特点,完美地促进了其在大片段基因簇精准编辑方面的应用。
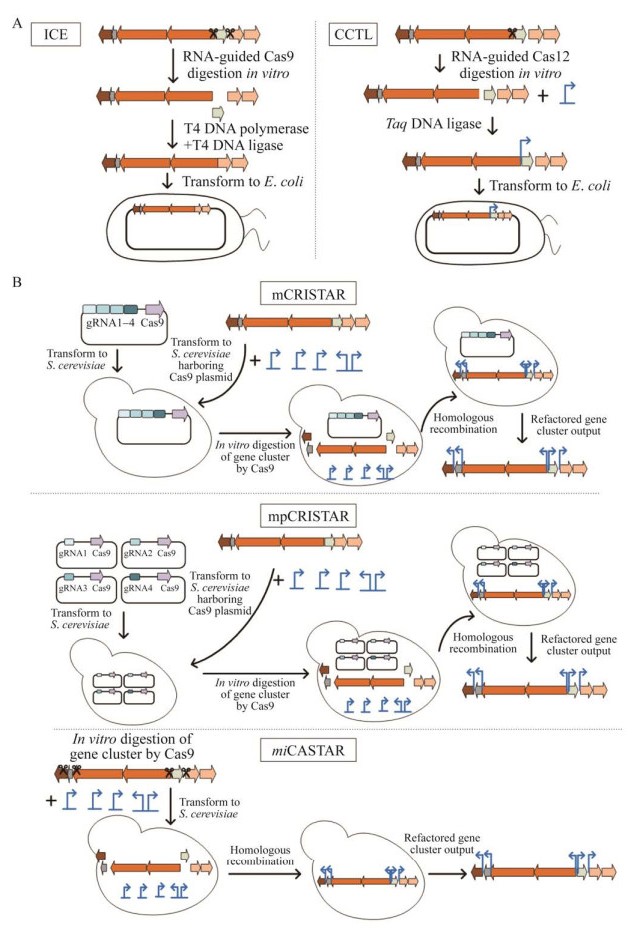
2.1 基于CRISPR/Cas系统的单靶点编辑方法2015年,Liu等构建了一种高效的体外CRISPR/Cas9介导的DNA编辑系统命名为ICE,可用于大片段DNA如生物合成基因簇的编辑[26] (图 2A)。研究者首先对环状pUC18 DNA片段进行Cas9酶切实验,发现酶切后会在切割位点处随机缺失5−14个核苷酸;随后,他们采用T4 DNA聚合酶对产物末端进行补齐处理,再用T4 DNA连接酶进行连接,显著提高了编辑的精确度;他们采用该系统分别删除了RK-682生物合成基因簇的rkD基因和全霉素生物合成基因簇的homE基因[26]。结果证实该方法具有快速、无缝及高效的优点,但目前该方法仅展现了针对基因簇单靶点编辑的能力。
|
| 图 2 基于CRISPR/Cas系统的基因簇编辑技术[26-31] Figure 2 Refactoring gene cluster methods mediated by CRISPR/Cas[26-31]. A:基于CRISPR/Cas系统的基因簇单靶点编辑方法;B:基于CRISPR/Cas系统的基因簇多靶点编辑方法 A: The diagram of single-site refactoring of gene cluster methods mediated by CRISPR/Cas; B: The diagram of multi-site refactoring of gene cluster methods mediated by CRISPR/Cas. |
|
|
此外,Cas12a不仅可用于大型基因簇的克隆,还可应用于基因簇的编辑。2017年,Lei等研究了CRISPR/Cas12a切割靶标DNA的特点,并将其应用于大片段的编辑[27];他们发现在较短间隔(spacer)长度(17−19 nt)的crRNA介导下,CRISPR/Cas12a可以特异切割靶标DNA并形成8个核苷酸的黏性末端。综合CRISPR/Cas12a的酶切和Taq DNA连接酶的连接特性,研究者开发出了针对大片段DNA的编辑系统,命名为CCTL (图 2A)[27]。研究者应用该方法将放线菌紫红素基因簇(act)中正调控基因actII-orf4的启动子替换为组成型强启动子,从而提高了放线菌紫红素的产量[27]。该系统为大片段DNA体外编辑提供了一个可选择的方法,但与ICE法类似,由于连接方式的限制,该方法只是展现了其单靶点编辑能力。
2.2 基于CRISPR/Cas系统的多靶点编辑方法天然产物基因簇的表达往往是由多个基因或操纵子组成的复杂代谢途径,对基因簇重构往往涉及对多个位点的通量化操作,比如将合适的启动子加载到基因簇中每个基因/操纵子的特定位置。因此,开发通量高效的大型基因簇多靶点编辑技术非常有必要,以实现增加片段、删除片段、插入片段和修改特定序列等多种操作。
目前报道的大部分基因簇多靶点编辑方法均基于CRISPR/Cas9的精确切割和酵母同源重组原理而开发,按具体策略一般分为两种。
一种策略是将待编辑基因簇重组质粒、CRISPR/Cas表达质粒,以及带有同源臂的重组片段转化至酵母细胞,在体内进行酶切与同源重组。根据此策略,2016年,Kang等建立了单筛选标记的CRISPR/Cas9和TAR介导的多靶点启动子工程方法,命名为mCRISTAR[28] (图 2B)。该策略需要将靶向多位点的多段crRNA序列、tracrRNA序列及Cas9表达序列一起构建酵母重组表达质粒,筛选阳性克隆;然后将其含有目标基因簇的质粒及带有同源臂的启动子片段共同转入阳性酵母细胞;在酵母体内完成基因簇启动子特定插入位点的Cas9酶切后,与带有相应同源臂的启动子片段进行同源重组,实现多个启动子的替换;该团队利用单标记筛选实现了4个启动子的同时替换,阳性率为21%;随后应用该方法将tetarimycin A (tam)基因簇的8个启动子进行两轮替换,激活了tam基因簇的表达[28]。该方法中CRISPR酵母表达质粒包含了多个重复spacer序列,这使得质粒构建具有一定挑战性,另外,所有的gRNA合并到一个表达质粒上,灵活性较小[28]。曾参与mCRISTAR工作的Kim等在mCRISTAR的基础上进一步开发了mpCRISTAR[29] (图 2B)。该方法基本过程为:构建带有不同营养缺陷型标记和不同gRNA的CRISPR表达质粒,将它们转化至酵母细胞并筛选获得阳性克隆;在此基础上进一步将带有目标基因簇的重组质粒及重组片段转化至阳性酵母细胞,从而实现基因簇中多达4−8个启动子的同时替换[29]。该方法比mCRISTAR效率提高了3倍,4个位点进行替换的阳性率达到了100%。尽管需要构建多个CRISPR表达质粒,但是相较于mCRISTAR方法,不同sgRNA的组合更加灵活。同样地,该方法也需要进行2次酵母转化和阳性克隆筛选,也比较费时费力。
另一种策略是利用CRISPR/Cas9技术将待编辑的基因簇质粒进行体外酶切,并构建带有同源臂的插入元件、筛选标记等片段,将切割后的质粒与DNA片段回收后一起转化至酵母细胞,通过酵母同源重组实现DNA片段的插入、删除或替换。在开发了基因簇一步克隆法(CATCH)后,娄春波等继续开发了组合利用体外CRISPR/Cas9酶切和酵母细胞同源重组系统对大型片段进行编辑的方法,该方法可以实现同时对6个位点进行多靶点编辑[30]。2019年,Kim等也发表了类似方法,命名为miCASTAR[31] (图 2B);他们以tam基因簇为模式基因簇进行了该方法的开发与优化,实现了4个启动子(其中一个启动子带有筛选标记)的同时插入,效率约为25%左右;随后,他们应用该方法对atolypene基因簇进行启动子替换,激活了基因簇的表达,产生了atolypene A和atolypene B两种化合物。
相较于第一种策略,基因簇质粒的体外酶切更简单、灵活、周期更短,而且该策略还适用于重组基因簇的组合文库,有利于针对基因簇进行多个位点的组合优化。但从效率来看,利用第一种策略可实现更多位点的同时编辑,编辑效率优于体外策略。
现将几种基于CRISPR/Cas开发的基因簇编辑技术特点、优缺点及成本进行总结,详见表 2。
| Methods | Principles | Number of simultaneous editing sites | Advantages | Disadvantages | Costs |
| ICE[26] | In vitro CRISPR/Cas9 digestion, T4 DNA polymerase+T4 DNA ligase assembly | 1 | Simple, short time period | Single-site editing | High cost (Cas9 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation, T4 DNA polymerase, T4 DNA ligase et al) |
| CCTL[27] | In vitro CRISPR/Cas12a digestion, Taq DNA ligase assembly | 1 | Simple, short time period. High efficiency (70% for single-site editing) | Single-site editing | High cost (Cas9 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation, Taq DNA ligase et al) |
| mCRISTAR[28] | In vivo CRISPR/Cas9 digestion and yeast homologous recombination | 4 | Multi-sites editing (21% efficiency for simultaneous replacement of 4 promoter sites) | Must use yeast, technically challenging. Times consuming |
Low cost (construction of CRISPR expression plasmid) |
| mpCRISTAR[29] | In vivo CRISPR/Cas9 digestion and yeast homologous recombination | 8 | High efficiency. Multi-sites editing (32% efficiency for replacement of 8 promoter sites) | Must use yeast, technically challenging. Times consuming | Low cost (construction of multiple CRISPR expression plasmids) |
| miCASTAR[31] | In vitro CRISPR/Cas9 digestion and yeast homologous recombination | 8 | Simple, short time period. Multi-sites editing (25% efficiency for replacement of 4 promoter sites) | Must use yeast, technically challenging. Approximately 5% efficiency for replacement of 8 promoter sites | High cost (Cas9 enzyme, T7 High Yield RNA Transcription Kit for sgRNA preparation) |
为了实现海量的微生物基因簇遗传资源向天然产物化合物资源的转变,突破以往各种个性化激活方法在基因簇激活方面的限制,开发具有一定通用性、工程化的基因簇激活策略是实现此目标的一个方向。在合成生物学思想的指导下,以优良模式链霉菌为底盘的异源表达和基因簇多靶点编辑为基础的激活策略,可最大程度地实现系统的可控性及方法的通用性。但因受天然产物基因簇克隆及编辑技术发展不足、优质链霉菌生物元件匮乏等因素的限制,目前这一策略仍然处于初级阶段,尚未充分发挥其工程化上的潜力。比如基因簇克隆方面:基于PCR的克隆方法存在克隆序列长度和模板高GC含量的限制及易发生突变的问题。尽管可以通过Gibson等方法组装多个短片段来生成目的基因簇序列,但往往耗时、费力且昂贵,尤其是想要获得长度大于50 kb的序列时难度和成本更高。另一种常用方法是采用限制性内切酶来消化基因组DNA,通过构建基因组文库获得基因簇片段,然而该方法步骤非常复杂,效率极其低下。在基因簇编辑方面,某些技术如酵母同源重组依赖的启动子工程[32]、DNA assembler[33]等技术已被用于多个基因簇的激活。然而这些技术的应用仍然受到诸多限制,比如酵母同源重组依赖的启动子工程依赖于多个筛选标记的使用,因此编辑位点有限且编辑效率较低。DNA assembler方法则是受到PCR的限制,不适用于较长的基因簇组装。相较于传统的限制性内切酶针对较短(4−8 bp)长度DNA的识别位点,CRISPR/Cas系统使用较长(约20 bp)可编程的sgRNA序列来作为切割靶DNA的识别位点,提高了其靶向特异性和通用性。通过与Gibson、酵母同源重组等组装技术结合有效提高了天然产物生物合成基因簇的克隆及多靶点编辑效率,加快了新型天然产物的开发速度。然而,目前还存在一些根本性的挑战,如现有的基因簇克隆和多靶点编辑技术普遍存在难度大、阳性克隆率低、技术要求高、难以满足通量化需求等问题。随着CRISPR/Cas新型工具的开发及DNA组装技术的发展,未来期待可以实现大片段基因簇克隆和多靶点编辑通量化、高效化,以适应基因簇激活的高通量、自动化操作需求,进而加速新型生物活性分子的大规模挖掘。
| [1] |
Boddy CN. Bioinformatics tools for genome mining of polyketide and non-ribosomal peptides[J]. Journal of Industrial Microbiology and Biotechnology, 2014, 41(2): 443-450. DOI:10.1007/s10295-013-1368-1 |
| [2] |
Liu G, Chater KF, Chandra G, Niu GQ, Tan HR. Molecular regulation of antibiotic biosynthesis in Streptomyces[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1): 112-143. DOI:10.1128/MMBR.00054-12 |
| [3] |
Ochi K, Hosaka T. New strategies for drug discovery: activation of silent or weakly expressed microbial gene clusters[J]. Applied Microbiology and Biotechnology, 2013, 97(1): 87-98. DOI:10.1007/s00253-012-4551-9 |
| [4] |
Rutledge PJ, Challis GL. Discovery of microbial natural products by activation of silent biosynthetic gene clusters[J]. Nature Reviews Microbiology, 2015, 13(8): 509-523. DOI:10.1038/nrmicro3496 |
| [5] |
Kang HS, Kim ES. Recent advances in heterologous expression of natural product biosynthetic gene clusters in Streptomyces hosts[J]. Current Opinion in Biotechnology, 2021, 69: 118-127. DOI:10.1016/j.copbio.2020.12.016 |
| [6] |
Gibson DG, Young L, Chuang RY, Venter JC, Hutchison CA, Smith HO. Enzymatic assembly of DNA molecules up to several hundred kilobases[J]. Nature Methods, 2009, 6(5): 343-345. DOI:10.1038/nmeth.1318 |
| [7] |
Li L, Zhao YW, Ruan LJ, Yang S, Ge M, Jiang WH, Lu YH. A stepwise increase in pristinamycin Ⅱ biosynthesis by Streptomyces pristinaespiralis through combinatorial metabolic engineering[J]. Metabolic Engineering, 2015, 29: 12-25. DOI:10.1016/j.ymben.2015.02.001 |
| [8] |
Yu Y, Bai LQ, Minagawa K, Jian XH, Li L, Li JL, Chen SY, Cao EH, Mahmud T, Floss HG, et al. Gene cluster responsible for validamycin biosynthesis in Streptomyces hygroscopicus subsp. jinggangensis 5008[J]. Applied and Environmental Microbiology, 2005, 71(9): 5066-5076. DOI:10.1128/AEM.71.9.5066-5076.2005 |
| [9] |
Deng Q, Zhou L, Luo MZ, Deng ZX, Zhao CM. Heterologous expression of avermectins biosynthetic gene cluster by construction of a bacterial artificial chromosome library of the producers[J]. Synthetic and Systems Biotechnology, 2017, 2(1): 59-64. DOI:10.1016/j.synbio.2017.03.001 |
| [10] |
Fu J, Bian XY, Hu S, Wang HL, Huang F, Seibert PM, Plaza A, Xia LQ, Müller R, Stewart AF, et al. Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J]. Nature Biotechnology, 2012, 30(5): 440-446. DOI:10.1038/nbt.2183 |
| [11] |
Yamanaka K, Reynolds KA, Kersten RD, Ryan KS, Gonzalez DJ, Nizet V, Dorrestein PC, Moore BS. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A[J]. PNAS, 2014, 111(5): 1957-1962. DOI:10.1073/pnas.1319584111 |
| [12] |
Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [13] |
Jiang WY, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [14] |
Zetsche B, Heidenreich M, Mohanraju P, Fedorova I, Kneppers J, DeGennaro EM, Winblad N, Choudhury SR, Abudayyeh OO, Gootenberg JS, et al. Multiplex gene editing by CRISPR–Cpf1 using a single crRNA array[J]. Nature Biotechnology, 2017, 35(1): 31-34. DOI:10.1038/nbt.3737 |
| [15] |
Du P, Lou CB, Zhao XJ, Wang QH, Ji XY, Wei WJ. CRISPR-based genetic switches and other complex circuits: research and application[J]. Life: Basel, Switzerland, 2021, 11(11): 1255. |
| [16] |
Jiang WJ, Zhao XJ, Gabrieli T, Lou CB, Ebenstein Y, Zhu TF. Cas9-assisted targeting of chromosome segments CATCH enables one-step targeted cloning of large gene clusters[J]. Nature Communications, 2015, 6: 8101. DOI:10.1038/ncomms9101 |
| [17] |
Wang XF, Yin SL, Bai J, Liu Y, Fan KQ, Wang HZ, Yuan F, Zhao BH, Li ZL, Wang WS. Heterologous production of chlortetracycline in an industrial grade Streptomyces rimosus host[J]. Applied Microbiology and Biotechnology, 2019, 103(16): 6645-6655. DOI:10.1007/s00253-019-09970-1 |
| [18] |
Li H, Pan YY, Liu G. Multiplying the heterologous production of spinosad through tandem amplification of its biosynthetic gene cluster in Streptomyces coelicolor[J]. Microbial Biotechnology, 2022, 15(5): 1550-1560. DOI:10.1111/1751-7915.13965 |
| [19] |
Kouprina N, Larionov V. TAR cloning: insights into gene function, long-range haplotypes and genome structure and evolution[J]. Nature Reviews Genetics, 2006, 7(10): 805-812. DOI:10.1038/nrg1943 |
| [20] |
Lee NCO, Larionov V, Kouprina N. Highly efficient CRISPR/Cas9-mediated TAR cloning of genes and chromosomal loci from complex genomes in yeast[J]. Nucleic Acids Research, 2015, 43(8): e55. DOI:10.1093/nar/gkv112 |
| [21] |
Meng JL, Feng RR, Zheng GS, Ge M, Mast Y, Wohlleben W, Gao JF, Jiang WH, Lu YH. Improvement of pristinamycin I (PI) production in Streptomyces pristinaespiralis by metabolic engineering approaches[J]. Synthetic and Systems Biotechnology, 2017, 2(2): 130-136. DOI:10.1016/j.synbio.2017.06.001 |
| [22] |
Wang HL, Li Z, Jia RN, Yin J, Li AY, Xia LQ, Yin YL, Müller R, Fu J, Stewart AF, et al. ExoCET: exonuclease in vitro assembly combined with RecET recombination for highly efficient direct DNA cloning from complex genomes[J]. Nucleic Acids Research, 2017, 46(5): e28. |
| [23] |
Tao WX, Chen L, Zhao CH, Wu J, Yan DZ, Deng ZX, Sun YH. In vitro packaging mediated one-step targeted cloning of natural product pathway[J]. ACS Synthetic Biology, 2019, 8(9): 1991-1997. DOI:10.1021/acssynbio.9b00248 |
| [24] |
Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, Van Der Oost J, Regev A, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [25] |
Enghiad B, Huang CS, Guo F, Jiang GD, Wang B, Tabatabaei SK, Martin TA, Zhao HM. Cas12a-assisted precise targeted cloning using in vivo Cre-lox recombination[J]. Nature Communications, 2021, 12: 1171. DOI:10.1038/s41467-021-21275-4 |
| [26] |
Liu YK, Tao WX, Wen SS, Li ZY, Yang AN, Deng ZX, Sun YH. In vitro CRISPR/Cas9 system for efficient targeted DNA editing[J]. mBio, 2015, 6(6): e01714-e01715. |
| [27] |
Lei C, Li SY, Liu JK, Zheng X, Zhao GP, Wang J. The CCTL (Cpf1-assisted Cutting and Taq DNA ligase-assisted ligation) method for efficient editing of large DNA constructs in vitro[J]. Nucleic Acids Research, 2017, 45(9): e74. |
| [28] |
Kang HS, Charlop-Powers Z, Brady SF. Multiplexed CRISPR/Cas9- and TAR-mediated promoter engineering of natural product biosynthetic gene clusters in yeast[J]. ACS Synthetic Biology, 2016, 5(9): 1002-1010. DOI:10.1021/acssynbio.6b00080 |
| [29] |
Kim H, Ji CH, Je HW, Kim JP, Kang HS. mpCRISTAR: multiple plasmid approach for CRISPR/Cas9 and TAR-mediated multiplexed refactoring of natural product biosynthetic gene clusters[J]. ACS Synthetic Biology, 2020, 9(1): 175-180. DOI:10.1021/acssynbio.9b00382 |
| [30] |
Lou CB, Zhao XJ. Molecular cloning method based on CRISPR/Cas9 and homologous recombination of Saccharomyces cerevisiae cell endogenous genes: CN105624146B[P]. 2019-02-15 (in Chinese) 娄春波, 赵学金. 基于CRISPR/Cas9和酿酒酵母细胞内源的同源重组的分子克隆方法: CN105624146B[P]. 2019-02-15. |
| [31] |
Kim SH, Lu WL, Ahmadi MK, Montiel D, Ternei MA, Brady SF. Atolypenes, tricyclic bacterial sesterterpenes discovered using a multiplexed in vitro Cas9-TAR gene cluster refactoring approach[J]. ACS Synthetic Biology, 2019, 8(1): 109-118. DOI:10.1021/acssynbio.8b00361 |
| [32] |
Montiel D, Kang HS, Chang FY, Charlop-Powers Z, Brady SF. Yeast homologous recombination-based promoter engineering for the activation of silent natural product biosynthetic gene clusters[J]. PNAS, 2015, 112(29): 8953-8958. DOI:10.1073/pnas.1507606112 |
| [33] |
Shao ZY, Zhao H, Zhao HM. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways[J]. Nucleic Acids Research, 2009, 37(2): e16. DOI:10.1093/nar/gkn991 |
 2022, Vol. 49
2022, Vol. 49