扩展功能
文章信息
- 张亦弛, 乔雪姣, 余珂
- ZHANG Yichi, QIAO Xuejiao, YU Ke
- 厌氧氨氧化细菌及其群落内细菌互作关系研究进展
- Characteristics of anammox bacteria and the interaction with other bacteria in anammox community: a review
- 微生物学通报, 2022, 49(2): 737-755
- Microbiology China, 2022, 49(2): 737-755
- DOI: 10.13344/j.microbiol.china.210732
-
文章历史
- 收稿日期: 2021-08-11
- 接受日期: 2021-09-24
- 网络首发日期: 2021-11-15
氮的生物地球化学循环是自然界最重要的元素循环过程之一,厌氧氨氧化的发现和研究是人们认识氮循环过程的重要一环。厌氧氨氧化细菌由于特殊的生命活动形式而具有独特的生态位。不同生境中存在的厌氧氨氧化细菌种类和特性有所差别,但都具有类似的细胞结构。厌氧氨氧化细菌细胞中最独特的细胞器是厌氧氨氧化体,其中氮代谢、碳代谢和ATP产生相耦合,共同构成了厌氧氨氧化细菌基本的生命活动过程。
目前人们仍未能实现厌氧氨氧化细菌的纯培养,但成功实现了对厌氧氨氧化细菌群落的富集,借助宏组学手段研究厌氧氨氧化细菌及其群落内细菌之间的互作关系是近年来厌氧氨氧化细菌的研究趋势。群落内的厌氧氨氧化细菌和其他细菌一起,通过物质代谢和信息交流相互作用、相互影响,共同维持厌氧氨氧化群落的稳定。本文详细阐述了厌氧氨氧化细菌的特性和厌氧氨氧化群落内细菌之间的物质代谢联系和信息交流方式,以期为深入研究厌氧氨氧化群落内细菌间的互作关系提供参考。
1 厌氧氨氧化细菌的种类早在20世纪中期,人们便预测在自然界中存在某种自养过程,以铵作为电子受体,使用硝酸盐或亚硝酸盐作为氧化剂,以氮气作为其能量代谢的最终产物[1]。人们在缺氧的海洋盆地中发现了铵损失的情况[2],之后也在反硝化生物反应器中发现了类似的现象[3],人们对此进行研究后最终将这种现象归结于微生物的生命活动,并把这一过程命名为厌氧氨氧化(anaerobic ammonium oxidation,anammox),实现该过程的细菌叫作厌氧氨氧化细菌(anaerobic ammonium oxidation bacteria,AnAOB)[4]。
厌氧氨氧化是指在厌氧条件下以亚硝酸盐[5]或一氧化氮[6]作为电子受体将铵氧化生成氮气和硝酸盐的过程。因其具有效率高、能耗低、环境相对友好的特点[7-9],尤其在处理低碳氮比污水中表现出色而备受关注。
厌氧氨氧化细菌分布广泛,既存在于海洋[10]、河流[11]、湖泊[12]等常规自然生境和热泉、热液口[13]等极端自然生境中,也存在于人工湿地[14]、农田[15]、污水处理厂[16]等人工生境中。厌氧氨氧化细菌是大气中氮气的主要生产来源,根据目前的估计,释放到大气中的约30%–70%氮气是由这些细菌产生的[8, 17-18]。
厌氧氨氧化细菌属于浮霉菌门(Planctomycetes)浮霉菌纲(Planctomycetia)厌氧氨氧化细菌目(Brocadiaceae)[4, 19],目前已发现的厌氧氨氧化细菌包括7个属[20],分别是Candidatus Brocadia[4]、Candidatus Kuenenia[21]、Candidatus Scalindua[22]、Candidatus Anammoxoglobus[23]、Candidatus Jettenia[24]、Candidatus Anammoximicrobium[25]和Candidatus Brasilis[26-27]。不同属的厌氧氨氧化细菌之间也有较大差异,Ca. Scalindua大部分为嗜盐菌[22];Ca. Anammoxoglobus的Ca. Anammoxoglobus propionicus能氧化甲酸和丙酸[23];Ca. Jettenia对亚硝酸盐有较高的耐受性[24];Ca. Brocadia的Ca. Brocadia fulgida有发光性能[28],显示出厌氧氨氧化细菌的物种多样性。
2 厌氧氨氧化细菌的特征 2.1 个体形态和细胞结构厌氧氨氧化细菌单一细胞呈不规则球形、椭球形、卵形、短杆形等,大小为(0.7−1.1) μm× (1.1−1.3) μm[29],是革兰氏阴性菌,细胞外无荚膜结构,细胞内含有丰富的细胞色素c,其富集培养物呈红棕色[30]。
厌氧氨氧化细菌细胞主要分为三部分[31]。最外层有细胞壁,成分为蛋白质[30];细胞从外至内共有三层膜,分别是细胞外膜、细胞质膜和厌氧氨氧化体膜,细胞外膜内部包裹着含细胞质的空间,细胞质膜所包裹着的细胞质中含有大量遗传物质,厌氧氨氧化体膜围成厌氧氨氧化体[32]。厌氧氨氧化体是厌氧氨氧化细菌特有的细胞器[33],作为厌氧氨氧化的场所,其在细胞分裂过程中被垂直继承给子细胞;厌氧氨氧化体膜是高度弯曲且封闭的,这种结构有利于在狭小空间内尽可能增大表面积,为酶提供附着位点,大量的羟胺氧化还原酶(hydroxylamine oxidoreductase,HAO)[34-35]、联氨合酶(hydrazine synthase,HZS)[36]、ATP合酶(ATP synthase,ATPase)[37]在其中发挥作用;厌氧氨氧化体膜中含有特殊的阶梯烷膜脂,这是一种类似梯形的致密结构,能够有效防止厌氧氨氧化中间代谢物的扩散损失。除厌氧氨氧化体膜外,阶梯烷膜脂也存在于厌氧氨氧化细菌其他的膜结构中,阶梯烷膜脂与传统的非阶梯烷膜脂相结合,使得厌氧氨氧化细菌的膜结构具有较低的渗透性[38]。
2.2 理化特性厌氧氨氧化细菌为自养细菌,生长速率缓慢,通常倍增时间为7−14 d[39],最适pH值在6.0−9.0之间[35]。
厌氧氨氧化细菌严格厌氧。有研究显示,当空气中氧饱和度高于0.5%时厌氧氨氧化细菌活性即会受到抑制[40],当氧饱和度维持1%时,抑制作用是可逆的,恢复厌氧条件后抑制即可被解除;当氧饱和度达18%时,会对厌氧氨氧化细菌活性造成不可逆的抑制作用[41]。
亚硝酸根离子和铵根离子是厌氧氨氧化的基质成分。有研究认为,厌氧氨氧化细菌活性受铵根离子浓度的影响较小,在不高于1 000 mg/L的情况下不会影响厌氧氨氧化过程[42],但过高浓度的铵根离子会产生游离态的氨[43],从而抑制厌氧氨氧化作用。与铵根离子浓度相比,厌氧氨氧化细菌对亚硝酸根离子浓度更为敏感[44],有研究认为,Ca. Brocadia适合生存于较高亚硝氮浓度的环境,而Ca. Kuenenia适合生存于较低亚硝氮浓度的环境[45],亚硝氮对厌氧氨氧化细菌活性的影响会因厌氧氨氧化细菌种类不同而有所差异。
在一定温度范围内,适当提高温度有利于提高厌氧氨氧化细菌活性。有研究显示,在35−40 ℃范围内厌氧氨氧化细菌活性最高,温度高于40 ℃会导致厌氧氨氧化细胞裂解从而永久失活[46]。另有研究发现,在6−43 ℃条件下均能进行厌氧氨氧化反应,当温度高于40 ℃或者低于15 ℃时,厌氧氨氧化速率迅速下降[47-48]。
金属离子主要通过影响厌氧氨氧化电子供受体及颗粒污泥的形成从而影响厌氧氨氧化细菌活性。有研究表明,Fe2+可以作为厌氧氨氧化反应的电子供体[49],Fe3+、FeOOH可以作为电子受体[21],Fe2+还能促进厌氧氨氧化颗粒污泥的形成[50]。另有研究显示,在向厌氧氨氧化反应器中投加Ca2+、Mg2+后,与对照组相比,体系中均形成了结构密实、轮廓清晰的颗粒污泥;当体系中厌氧氨氧化活性分别达到最高水平时,Mg2+最佳投加浓度为1.45 mg/L,Ca2+最佳投加浓度为1.20 mg/L,而且投加Mg2+比投加Ca2+有着更高的厌氧氨氧化效率[42]。还有研究表明,适当浓度的Cu2+和Ni2+对厌氧氨氧化活性也有促进作用。相关研究显示,添加浓度为1.00 mg/L的Cu2+使体系内厌氧氨氧化活性最大增强了41.0%,添加1.74 mg/L的Ni2+使厌氧氨氧化活性增强了63.5%[51]。然而过量的金属离子对厌氧氨氧化细菌活性具有抑制作用[52]。
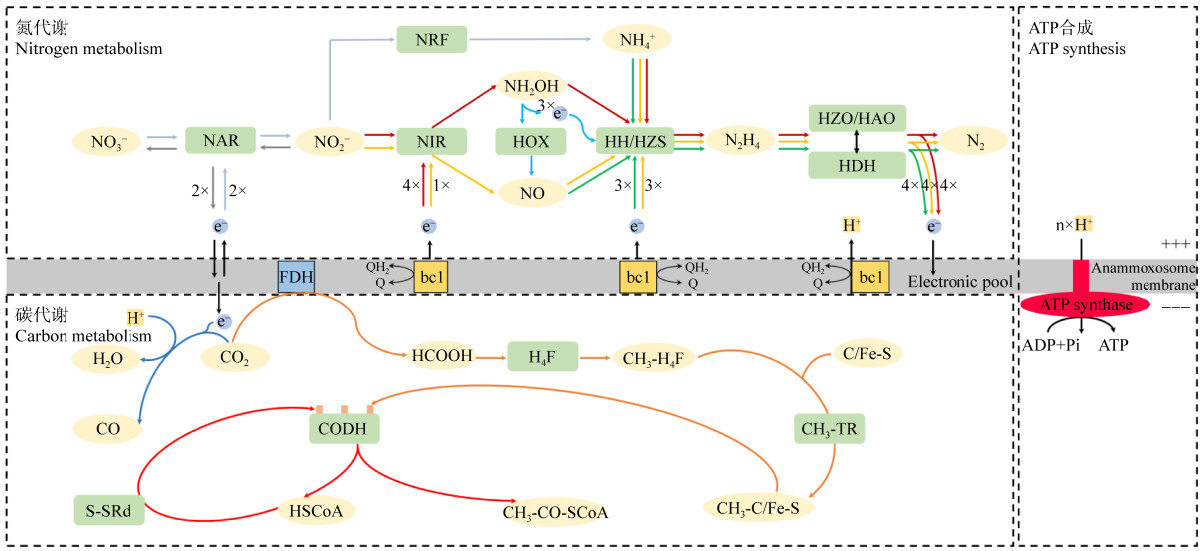
3 厌氧氨氧化细菌的代谢 3.1 氮代谢厌氧氨氧化细菌具有的厌氧氨氧化代谢过程是通过中间代谢产物分步进行的。目前已发现的厌氧氨氧化反应有3种实现途径,途径I和途径II均以亚硝酸盐为最初电子受体,途径III以一氧化氮为最初电子受体,分别以羟胺+联氨(途径I)、一氧化氮+联氨(途径II)、联氨(途径III)作为中间代谢产物进行厌氧氨氧化反应。
起初Van De Graaf等[53]在研究羟胺对厌氧氨氧化的作用时发现存在联氨的瞬时积累,后续的研究也发现人为添加羟胺或联氨能够增强厌氧氨氧化活性[28],Van De Graaf等基于传统微生物学研究提出了以羟胺和联氨为中间代谢产物的三步模型[53] (途径I):(1) 1分子亚硝酸盐得4分子e–被还原为1分子羟胺;(2) 1分子羟胺和1分子铵反应生成1分子联氨;(3) 1分子联氨失去4分子e–生成1分子氮气。在此模型中,亚硝酸盐转化为羟胺并与铵反应生成氮气的过程具有闭合的氧化还原平衡,但该模型未指出每一步催化作用的酶。之后随着分子生物学和细胞生物学的发展,Jetten等[54]在研究Ca. Brocadia anammoxidans时发现了厌氧氨氧化体的存在,由此提出假设:存在于厌氧氨氧化体膜外表面的亚硝酸还原酶(nitrite reductase,NIR)能够将细胞质内的亚硝酸盐转化为羟胺,在跨膜的联氨水解酶(hydrazine hydrolyase,HH)作用下,厌氧氨氧化体膜外侧的羟胺与存在于厌氧氨氧化体膜内侧的铵反应生成联氨,存在于厌氧氨氧化体内部的联氨经附着在厌氧氨氧化体膜内表面的联氨氧化酶(hydrazine oxidase,HZO)催化生成氮气,同时释放出的4个电子通过电子传递链转移到附着在厌氧氨氧化体外膜上的亚硝酸还原酶上,以此不断补充还原亚硝氮所消耗的电子。这一设想初步定位了三步模型中氮代谢相关酶的存在。
随着基因组学技术的发展,Strous等[21]在研究Ca. Kuenenia stuttgartiensis基因组时发现其缺失编码由亚硝酸盐到羟胺的还原酶基因,但存在由亚硝酸盐到一氧化氮的还原酶基因,因此提出了以一氧化氮和联氨为中间代谢产物的新三步模型[21, 35, 55] (途径II):(1) 亚硝酸还原酶(NIR)的1分子e–催化1分子亚硝酸盐生成1分子一氧化氮;(2) 联氨水解酶(HH)的3分子e−催化1分子一氧化氮和1分子铵反应生成1分子联氨;(3) 在羟胺氧化还原酶(HAO)作用下1分子联氨失去4分子e–生成1分子氮气。在此模型中,联氨氧化过程提供的4分子电子经辅酶Q和细胞色素bc1复合物传递后,3分子电子给联氨水解酶,1分子电子给亚硝酸还原酶,实现电子流的循环补充和利用。该模型认为,参与氮代谢的酶位于厌氧氨氧化体内,传递电子的辅酶Q和细胞色素bc1复合物位于厌氧氨氧化体膜上,该观点在后续相关研究中被普遍接受。
Kartal等[17]以新三步模型(途径II)为基础,提出在Ca. Kuenenia stuttgartiensis中存在于厌氧氨氧化体内的1分子羟胺能够经羟胺氧化酶(hydroxylamine oxidase,HOX)氧化为1分子一氧化氮,该过程释放出的3分子电子可供HZS利用;之后这部分一氧化氮与由亚硝酸盐氧化而来的一氧化氮一同被联氨合酶转化为联氨,继而经联氨脱氢酶(hydrazine dehydrogenase,HDH)转化为氮气;并且明确指出厌氧氨氧化体膜上含有ATP合酶用以产生能量。
Hu等[6]报道了Ca. Kuenenia stuttgartiensis中存在直接以一氧化氮为电子受体的厌氧氨氧化作用途径(途径III):(1) 1分子一氧化氮和1分子铵反应生成1分子联氨;(2) 1分子联氨失去4分子e–生成1分子氮气。该过程中1分子联氨氧化所提供的4分子电子,其中3分子用于一氧化氮的还原,剩余电子用于碳的固定。
目前已发现的3种厌氧氨氧化途径如表 1所示。
| 途径 Processes |
代谢过程 Metabolic processes |
碳固定电子来源 Carbon fixation electron source |
研究对象 Research anammox bacteria |
参考文献 References |
| Process I | NO2–+4e–→NH2OH NH2OH+NH4+→N2H4 N2H4→N2+4e– |
NO2–→NO3–+2e– | Ca. Brocadia anammoxidans Ca. Brocadia sinica |
[53-54] |
| Process II | NO2−+e−→NO NO+NH4++3e–→N2H4 N2H4→N2+4e– |
NO2–→NO3–+2e– | Ca. Kuenenia stuttgartiensis | [21, 35, 55] |
| Process III | NO+NH4++3e–→N2H4 N2H4→N2+4e– |
N2H4→N2+4e– | Ca. Kuenenia stuttgartiensis | [6] |
| 注:上述反应式仅体现厌氧氨氧化过程中氮元素(包括NO2–、NH4+、NH2OH、NO、N2H4、N2)转化及电子转移过程。未体现其他物质(如H2O、H+等)的参与 Note: The above equations only reflect the transformation of nitrogen elements (including NO2–, NH4+, NH2OH, NO, N2H4, N2) and electron transfer process about anammox processes. No other substances (such as H2O, H+, etc) are involved. |
||||
值得注意的是,厌氧氨氧化代谢过程(途径I和途径II)第一步涉及的亚硝酸还原酶(NIR)在不同厌氧氨氧化菌属中存在差异。已知的NIR包括2种:含血红素的cd1亚硝酸盐还原酶(cd1-NIR,由nirS基因编码)[56]和含铜的亚硝酸盐还原酶(Cu-NIR,由nirK基因编码)[57]。目前,nirS基因只在Ca. Scalindua[58]和Ca. Kuenenia[55, 59]中发现,并且相较而言,Ca. Kuenenia中编码血红蛋白c和d的基因在转录和蛋白水平上表达量较小[21],这可能解释了厌氧氨氧化细菌生长缓慢的原因。相反地,Ca. Jettenia[60-61]中的亚硝酸还原酶由nirK基因编码。然而在一些Ca. Brocadia (如B. fulgida和B. sinica)中均未发现nirS和nirK基因的存在[62-64]。另外,跟亚硝酸在Ca. Kuenenia stuttgartiensis中被还原为一氧化氮[55]不同,亚硝酸在Ca. Brocadia sinica中被还原为羟胺[62]。有研究推测,在Ca. Brocadia中或许存在一种尚未被发现的类HAO酶能够实现将亚硝氮还原为羟胺[65]。
厌氧氨氧化细菌除了具有独特的厌氧氨氧化代谢过程外,也具有其他的氮代谢途径。有研究[66]利用15N标记的硝酸盐作为底物对经物理纯化得到的高纯度Ca. Kuenenia stuttgartiensis进行培养,发现厌氧氨氧化细菌能够借助外源有机碳作为电子供体,将15NO3–还原为15NO2–继而还原为15NH4+,最终检测到了15N15N的氮气分子,证实厌氧氨氧化细菌具有将硝酸盐异化为铵的能力,并且该过程能够与厌氧氨氧化过程相耦合。最近的研究显示,厌氧氨氧化细菌同样可以利用自身细胞内的有机碳作为电子供体为硝酸盐异化为铵的过程提供电子[67]。该过程中,硝酸盐能够被硝酸盐还原酶(nitrate reductase,NAR)转化为亚硝酸盐,随后在另一类亚硝酸盐还原酶(nitrite reductase,NRF)作用下被还原为铵。通过硝酸盐异化为铵过程,厌氧氨氧化细菌就能在不借助其他微生物的情况下独立利用硝酸盐为其厌氧氨氧化反应提供底物,这是厌氧氨氧化细菌的一种独特的生存策略[66-68]。由此可见,厌氧氨氧化细菌具有氮代谢多样性,这种多样性能够提高厌氧氨氧化细菌在不同自然生态系统中的生存能力。
3.2 碳代谢一般来说,厌氧氨氧化细菌碳代谢过程即无机碳被还原为有机碳的过程[69]。基于碳的同位素示踪手段,人们将厌氧氨氧化细菌的碳固定途径确定为乙酰辅酶A途径[70-72],在后续的研究中也在Ca. Kuenenia基因组中发现了完整的与乙酰辅酶A途径有关基因的存在[21, 73-74],而其他所有已知的碳固定途径基因仍然缺失或不完整。乙酰辅酶A固碳途径主要为:(1) 1分子二氧化碳在甲酸脱氢酶(formic acid dehydrogenase,FDH)作用下生成1分子甲酸(HCOOH),并在四氢叶酸酶(tetrahydrofolate enzyme)作用下生成1分子甲基四氢叶酸(CH3-H4F),之后在甲基转移酶(methyltransferase,CH3-TR)作用下,与类咕啉/铁硫蛋白(C/Fe-S)结合生成甲基类咕啉/铁硫蛋白(CH3-C/Fe-S),最终-CH3结合到CO脱氢酶(CO dehydrogenase,CODH)的甲基结合位点上;(2) 另外1分子二氧化碳的2分子e–被还原为1分子一氧化碳,随后一氧化碳结合到CODH的羰基结合位点上;(3) 辅酶A (HSCoA)在二硫化物还原酶(disulfide reductase,S-SRd)作用下将-SCoA转移到CODH上的-SCoA位点。至此,-CH3、-CO和-SCoA在CODH作用下生成乙酰辅酶A (CH3-CO-SCoA),完成由无机碳到有机碳的固定[75]。在此过程中碳固定所利用的电子来源于厌氧氨氧化细菌体内的氮代谢过程。
除二氧化碳、碳酸氢根、碳酸根等无机碳外,厌氧氨氧化细菌也可以代谢一些小分子有机碳,显示出了厌氧氨氧化细菌的有机营养特性。有研究发现,厌氧氨氧化细菌内存在甲酸脱氢酶[76],可以将甲酸转化为二氧化碳,从而进行乙酰辅酶A途径的碳固定作用。另外有研究报道,Ca. Kuenenia、Ca. Jettenia、Ca. Brocadia等厌氧氨氧化细菌都具有ADP型乙酰辅酶A合成酶(ADP-forming acetyl-CoA synthetase,ADP-ACS)[73, 77-78],能够将乙酸催化生成乙酰辅酶A;乙酸盐(C/N为0.3)的加入也会刺激厌氧氨氧化细菌内乙酰辅酶A合成酶基因的表达[77],从而进一步提高厌氧氨氧化菌代谢乙酸的能力。同样地,有研究发现在Ca. Kuenenia、Ca. Brocadia和Ca. Jettenia中也有丙酸代谢相关的酶存在[79],表明厌氧氨氧化细菌也有代谢丙酸的能力。宏基因组分析表明,Ca. Kuenenia、Ca. Brocadia可以通过三羧酸循环途径降解乙酸和丙酸[74, 80]。厌氧氨氧化细菌利用有机碳的特性能够在一定程度上增强其环境适应能力。
3.3 氮代谢-碳代谢耦合和能量的产生正如前文所述,目前已报道的厌氧氨氧化细菌氮代谢途径中,除直接以一氧化氮为电子受体[6]的厌氧氨氧化作用途径外,一般认为亚硝酸盐与铵反应生成联氨进而生成氮气的过程具有闭合的氧化还原平衡,因此,任何与无机碳有关的碳固定过程必须从其他反应中得到电子。Van De Graaf等[53]提出,用于碳固定过程的电子是厌氧氨氧化细菌通过体内的硝酸氧化还原酶将亚硝酸盐在厌氧条件下氧化为硝酸盐的过程中提供的。Strous等[21]认为进入乙酰辅酶A固碳途径的4个高能电子是由联氨脱氢时提供的,并通过联氨脱氢酶传递给铁氧还原蛋白,最后转移到CO脱氢酶中用于二氧化碳的固定,而亚硝酸盐氧化为硝酸盐所产生的电子通过电子传递链分别传递给了辅酶Q和细胞色素bc1复合物,最终传递给亚硝酸还原酶和联氨水解酶用于亚硝酸的还原和联氨的产生。Hu等[6]根据以一氧化氮为电子受体的厌氧氨氧化模型提出,1分子联氨氧化所提供的4分子电子,其中3分子用于一氧化氮的还原,剩余电子用于碳的固定。尽管人们对于碳固定过程所用到电子的来源意见不一,但公认碳固定过程的电子是由氮代谢过程提供的,氮代谢和碳代谢之间有着密切的耦合过程。
厌氧氨氧化细菌生长所需的ATP由存在于厌氧氨氧化体膜上的ATP合酶提供[76],伴随电子传递,厌氧氨氧化体内有大量质子被排至厌氧氨氧化体膜外侧,在膜两侧形成质子动力(proton-motive force,PMF)[35],从而实现了ATP的合成。
厌氧氨氧化细菌体内氮代谢-碳代谢耦合和能量的产生过程如图 1所示。
|
| 图 1 厌氧氨氧化细菌体内氮代谢-碳代谢耦合和能量的产生过程 Figure 1 Nitrogen metabolism-carbon metabolism coupling and energy generation process in anaerobic ammonium oxidation bacteria. 不同颜色线条代表不同路径。氮代谢主要包含3种厌氧氨氧化过程、亚硝酸盐氧化为硝酸盐过程、异化硝酸盐还原成铵过程, NIR: 亚硝酸还原酶; HH: 联氨水解酶; HZS: 联氨合酶; HZO: 联氨氧化酶; HAO: 羟胺氧化还原酶; HDH: 联氨脱氢酶; NAR: 硝酸盐还原酶; NRF: 亚硝酸还原酶; HOX: 羟胺氧化酶。碳代谢主要包含乙酰辅酶A途径所涉及的二氧化碳和辅酶A的转化, FDH: 甲酸脱氢酶; H4F: 四氢叶酸酶系; CH3-TR: 甲基转移酶; C/Fe-S: 类咕啉/铁硫蛋白; CODH: CO脱氢酶; S-SRd: 二硫化物还原酶 Different colored lines represent different paths.Nitrogen metabolism mainly includes three kinds of anaerobic ammonium oxidation process, nitrite oxidation to nitrate process, dissimilated nitrate reduction to ammonium process.NIR: Nitrite reductase; HH: Hydrazine hydrolyase; HZS: Hydrazine synthase; HZO: Hydrazine oxidase; HAO: Hydroxylamine oxidoreductase; HDH: Hydrazine dehydrogenase; NAR: Nitrate reductase; NRF: Nitrite reductase; HOX: Hydroxylamine oxidase.Carbon metabolism mainly involves the conversion of CO2 and CoA involved in acetyl-CoA pathway.FDH: Formic acid dehydrogenase; H4F: Tetrahydrofolate enzyme; CH3-TR: Methyltransferase; C/Fe-S: Corrinoid/ferrithionein; CODH: CO dehydrogenase; S-SRd: Disulfide reductase. |
|
|
目前,厌氧氨氧化细菌仍未实现分离纯培养,在厌氧氨氧化细菌富集群落中,多种厌氧氨氧化细菌共存[81],并且除厌氧氨氧化细菌所属的浮霉菌门(Planctomycetes)外,变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、拟杆菌门(Bacteroidetes)等菌群也占有一定丰度,通常占30%−70%[78]。厌氧氨氧化细菌不仅自身具有代谢多样性,与群落内的其他菌群也存在代谢关联性[65, 82],这些细菌之间存在氮代谢和碳代谢的协同和竞争作用。
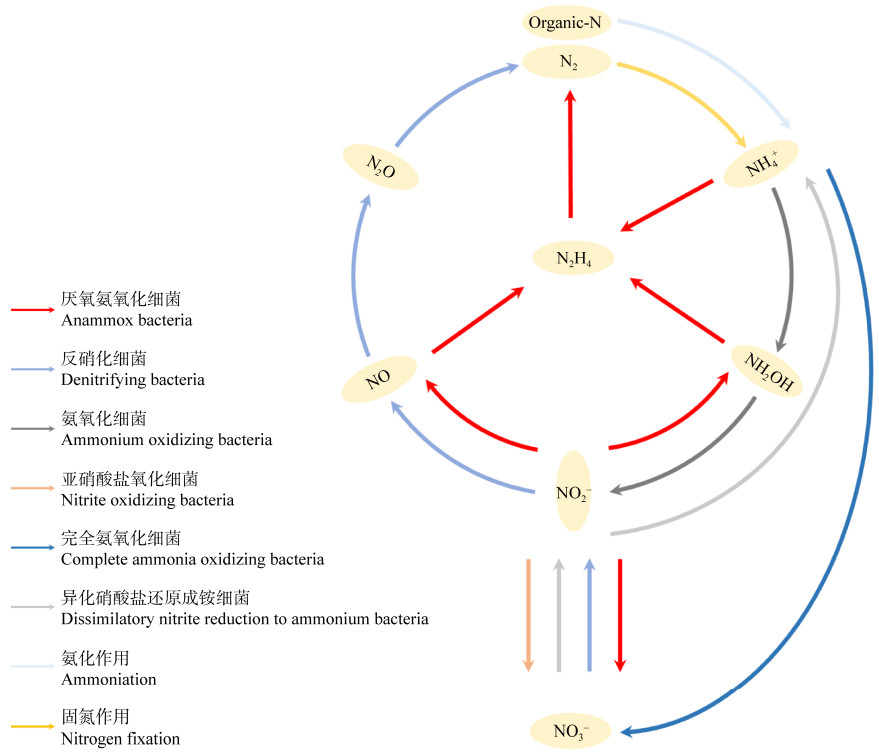
厌氧氨氧化群落内细菌间存在着氮代谢相关性,主要通过“产物-底物”的方式产生协同作用,通过“底物-底物”的方式产生竞争作用[83]。在群落内,硝化细菌中的氨氧化细菌(ammonium oxidizing bacteria,AOB)对氨氮的不完全氧化作用能够将氨氮氧化成亚硝氮,从而为厌氧氨氧化细菌提供电子受体;同时,厌氧氨氧化细菌对亚硝酸盐的利用则去除了对氨氧化细菌活性具有潜在抑制作用的产物[84],体现了氨氧化细菌和厌氧氨氧化细菌的协同作用。硝化细菌中的亚硝酸盐氧化细菌(nitrite oxidizing bacteria,NOB)和厌氧氨氧化细菌都以亚硝酸盐作为反应的底物,二者之间存在底物竞争[85]。完全氨氧化细菌(complete ammonia oxidizing bacteria,Comammox)能够将氨氮一步转化为硝氮,同样跟厌氧氨氧化细菌存在底物竞争作用[86]。异化性硝酸盐还原过程(dissimilatory nitrate reduction)包括异养反硝化作用(heterotrophic denitrification)和异化硝酸盐还原成铵作用(dissimilatory nitrate reduction toammonium,DNRA)两类,对于反硝化作用来说,反硝化细菌(denitrification bacteria)以硝酸盐作为电子受体,将硝酸盐逐步还原为氮气,在反硝化过程中产生的中间产物亚硝酸盐,一方面可以被厌氧氨氧化细菌所利用,另一方面也能够继续被反硝化细菌所还原[87]。对于DNRA过程来说,dissimilatory nitrate reduction to ammonium (DNRA)细菌产生的铵和亚硝酸盐作为厌氧氨氧化作用的底物被厌氧氨氧化细菌所利用,同时由于DNRA细菌和厌氧氨氧化细菌均以亚硝酸盐作为底物,DNRA细菌大量增殖时会造成与厌氧氨氧化细菌的过度竞争[88]。另外,反硝化细菌和DNRA细菌都能够将硝酸盐还原为亚硝酸盐,并利用亚硝酸盐完成后续反应[89]。可见,反硝化细菌、DNRA细菌和厌氧氨氧化细菌之间也同样存在氮代谢的协同和竞争作用。有研究报道,在厌氧氨氧化群落体系中呼吸硝酸还原酶基因narGH在绝大多数异养微生物体内被编码和表达[90],这表明体系中共存的异养细菌大部分可以借助该基因将硝酸盐还原为亚硝酸盐;有研究通过宏基因组和宏转录组手段分析厌氧氨氧化细菌和其他伴生菌的基因表达水平,推测绿菌门可高效分解胞外有机物同时可以将硝酸盐还原为亚硝酸盐[65],这均可为厌氧氨氧化细菌的厌氧氨氧化过程提供底物。了解不同菌群间的氮代谢相关性有助于剖析不同细菌的氮代谢潜力,为提高厌氧氨氧化体系脱氮性能提供参考。厌氧氨氧化群落内细菌之间氮相关底物协同利用与竞争关系如图 2所示。
|
| 图 2 厌氧氨氧化群落内细菌之间氮代谢关系图 Figure 2 Relationship of nitrogen metabolism among bacteria in anammox community. |
|
|
除氮代谢相关性外,群落内细菌之间的碳代谢相关性也同样存在,主要是通过底物的交叉供应实现的。有研究表明,Ca. Kuenenia缺乏丙氨酸、甲硫氨酸合成酶,而Nitrosomonas和Nitrospira等硝化细菌及Thauera和Comamonas等反硝化细菌则有这些氨基酸的合成酶[73],因此厌氧氨氧化细菌可以从硝化菌和反硝化菌处获取相应氨基酸,用以维持自身的正常生命活动。相反地,有研究发现Ca. Brocadia的AMX1物种是群落内精氨酸、丝氨酸和蛋氨酸的提供者,与之共存的绿弯菌门的CFX2物种体内缺乏这些氨基酸的合成途径,其所需的相应氨基酸是由厌氧氨氧化菌为它们提供的[78]。这预示着在厌氧氨氧化群落内可能存在氨基酸互补利用机制,这种互补机制可以加强厌氧氨氧化群落内细菌间的合作关系,提高群落稳定性,同时也是降低各细菌能量消耗的一种有效策略[91-92]。除氨基酸外,厌氧氨氧化细菌和群落内其他细菌之间也存在其他碳代谢联系。有研究发现,厌氧氨氧化细菌缺乏钼辅因子和叶酸生物合成途径,钼辅因子是甲酸脱氢酶的重要辅助因子[93],叶酸是组成厌氧二氧化碳固定酶的重要亚基[94],二者都和厌氧氨氧化细菌内二氧化碳的乙酰辅酶A途径固定过程息息相关。厌氧氨氧化细菌所需的钼辅因子和叶酸均可由装甲菌门(Armatimonadetes)和变形菌门提供[95],协助厌氧氨氧化细菌完成碳固定过程。
群落内细菌之间存在交叉供食的途径。厌氧氨氧化细菌在生长过程中容易分泌大量的胞外聚合物(extracellular polymeric substance,EPS),EPS附着于细菌细胞表面,对厌氧氨氧化体系的生物絮凝性、吸附性和稳定性有显著影响[96]。EPS成分比较复杂,主要含多糖、蛋白质、腐殖酸、核酸和脂质等[97],其中多糖和蛋白占主要部分。在活性污泥的生物絮凝中,多糖和蛋白质作为主要的组成物质起到了重要作用[98]。有研究认为,蛋白质对于颗粒的粘连起到重要作用,蛋白质结构松散且含有疏水性氨基酸,能够充分暴露内部的疏水集团,从而促进生物絮凝[99];还有研究认为,EPS中的多糖在活性污泥的生物絮凝中起主要作用,多糖中含有羧基、羟基等带负电荷的官能团具有架桥作用,可促进颗粒污泥的形成[100]。除此之外,多糖和蛋白质作为重要的营养物质存在于EPS中,由于EPS中营养物质丰富,因此EPS可以作为厌氧氨氧化群落内细菌之间交叉供食的媒介[65],比如运输氨基酸、维生素等公共营养物质供整个群落内细菌使用,这种群落内细菌间物质代谢联系[74]有利于维持厌氧氨氧化体系微生物生态的稳定。
4.2 群体感应群体感应(quorum sensing,QS)是细菌中普遍存在的一种通讯机制,是细菌通过产生细胞外信号分子来完成的,这种信号分子称为自诱导物(autoinducers,AI)[101]。当微环境中的信号分子达到阈值时,相关信号就会被群落中的细胞接收,并由此开始调节特定基因的表达,从而调节整个群落的各种行为,这种调节对整体菌群而言通常是趋利的[102-103]。
厌氧氨氧化群落内存在不止一种群体感应机制[104]。厌氧氨氧化细菌属于革兰氏阴性菌,一般以N乙酰化高丝氨酸内酯(N-acyl-homoserine lactone,AHL)类化合物作为种内交流的信号分子[105],以扩散性信号因子(diffusible signal factor,DSF,顺-2-不饱和脂肪酸化合物)作为种内和种间交流的信号分子[106]。此外,环二鸟苷酸(bis-(3-5′)-cyclic dimeric guanosine monophosphate,c-di-GMP)[107]是细菌中普遍存在的第二信使分子,能与群体感应系统分泌的AI相互作用形成复杂的信号传递网络,也在厌氧氨氧化群落中发挥接收胞外信号、调节生理功能的作用[108]。厌氧氨氧化群落内常见的信号分子类型及功能如表 2所示。
| 信号分子 Signaling molecules |
作用群体 Affected groups |
作用层面 Affected aspects |
参考文献 References |
|
| AHL | C4-HSL | Intraspecific | Growth rate | [109] |
| Gene expression | ||||
| C6-HSL | Intraspecific | Growth rate | [105, 110] | |
| Granular sludge activity | ||||
| Aggregation | ||||
| C8-HSL | Intraspecific | Granular sludge formation | [105, 110] | |
| C12-HSL | Intraspecific | Sludge adhesion growth | [111] | |
| Intraspecific | Heterotrophic bacteria growth | [110] | ||
| 3OC6-HSL | Intraspecific | Growth rate | [105] | |
| Granular sludge formation | ||||
| Aggregation | ||||
| 3OC8-HSL | Intraspecific | Granular sludge formation | [105] | |
| Aggregation | ||||
| 3OC10-HSL | Intraspecific | Granular sludge formation | [111] | |
| 3OC12-HSL | Intraspecific | Aggregation | [111] | |
| DSF | Intra- and interspecific | EPS production | [112] | |
| Signal grading regulation | ||||
| c-di-GMP | Intra- and interspecific | EPS production | [107, 113] | |
| Defend against adverse circumstances | ||||
不同的信号分子有不同的来源。AHL是由hdtS基因编码的,有研究显示,hdtS基因在Ca. Jettenia caeni中被证实为jqsI-1和jqsI-2,产物分别是OC6-HSL、C6-HSL、OC8-HSL和C8-HSL[105]。由AHL参与的群体感应主要通过LuxI/LuxR类机制实现,自诱导合成酶LuxI负责产生特定的AHL,并转移至细胞外,AHL可通过自由扩散进入细菌细胞内。当AHL达到临界浓度时,AHL与细胞内LuxR样受体蛋白结合,形成LuxR-HSL自诱导剂复合物共同激活靶基因的转录,从而调节特定基因的表达。DSF参与的群体感应有三类[114],分别由RpfF/RpfC/ RpfG蛋白、RpfF/RpfR蛋白和顺式-2-癸烯酸介导。目前除顺式-2-癸烯酸有待研究外,研究发现,在厌氧氨氧化细菌Ca. Brocadia sinica基因组中存在合成RpfF/RpfC/RpfG蛋白的基因[115],某些与之共存的变形菌门细菌基因组中存在合成RpfF/RpfR的基因,这意味着在厌氧氨氧化细菌群落内能够通过这2种方式合成DSF,从而对菌群进行调控。c-di-GMP是细菌群落中普遍存在的第2种信使,一般由鸟苷酸环化酶合成,广泛存在于细菌群落中进行群落调控和信息传递[108]。
不同的信号分子对群落调节起到不同的作用。人们对厌氧氨氧化相关工艺中AHL的种类及含量进行了检测,发现在不同厌氧氨氧化群落中的AHL有所不同,功能也有所差异。有研究显示,C6-HSL可以明显提高厌氧菌的活性和生长速率[116-117],C12-HSL信号分子对厌氧氨氧化污泥的附着生长能力有促进作用[111],还能刺激群落内其他异养菌生长[110]。人为添加3-碳位置取代基为羰基的AHL,对污泥附着生长的促进作用优于3-碳位置无取代基的AHL[118],长期添加一定浓度C10-HSL能够有效提升厌氧氨氧化体系整体脱氮率,并促使体系内产生丰富的EPS[119]。也有研究报道,当向厌氧氨氧化体系中添加AHL抑制剂后,会导致厌氧氨氧化整体脱氮率下降,EPS的合成也受到影响[120]。另外,有研究表明,DSF具有调控EPS的产生和生物膜形成等功能,还可以通过调节c-di-GMP进一步调控细菌代谢,能够调节厌氧氨氧化菌群落中的公共物品交叉供给[115],在细胞物质利用中发挥了关键作用。有研究显示,在c-di-GMP的调节下,面对低温、有氧和低pH值等不利的环境压力时,细胞内较高水平的c-di-GMP会降低细菌运动能力并增加细胞外聚合物的合成量,从而使厌氧氨氧化细菌更易于形成颗粒以抵御不利的环境[113]。
当然,不同类型的信号分子可以协同发挥作用。有研究发现,厌氧氨氧化群落中存在的AHL与c-di-GMP能够协同调控细菌胞外多糖的合成[121]。还有研究发现,DSF含有的RpfC/RpfG、RpfR能够将c-di-GMP降解生成转录抑制性配体Clp,而Clp作为DSF的信号调节剂,能够调节群落细菌的氨基酸协同生产与利用、基因表达和转录以及脂质合成[115]。
此外,有研究发现在厌氧氨氧化群落中除了发生细菌通信外,也发现了通信干扰,这可能是由于相关信号分子的降解而发生的[122]。群体感应与厌氧氨氧化群落内微生物的生命活动息息相关,是调控微生物群落的重要方式。
5 总结和展望厌氧氨氧化细菌在自然界中分布广泛、种类多样,独特的代谢特征决定了其在水处理中的巨大应用潜力。然而由于厌氧氨氧化细菌生长速率缓慢、生活条件苛刻、活性易被抑制等原因,导致厌氧氨氧化反应器启动周期长、运行不稳定,脱氮效率难以保障。
从厌氧氨氧化细菌特性入手开展研究,是探寻厌氧氨氧化体系稳定化模式的重要途径。由于目前厌氧氨氧化细菌仍难以在纯菌株水平上开展特性研究,宏组学技术的应用为厌氧氨氧化细菌的深入研究提供了手段。现有研究显示,DNRA细菌的大量增殖会导致厌氧氨氧化反应器脱氮性能下降,影响厌氧氨氧化细菌活性和整个厌氧氨氧化群落的稳定,因此,控制厌氧氨氧化体系中DNRA细菌的大量增殖可能是维持群落稳定的重要一环[88]。采用宏组学联用技术解析厌氧氨氧化群落核心微生物的互作关系,可以进一步理解厌氧氨氧化细菌及其主要共生菌之间的相互作用机制,进而为调控厌氧氨氧化细菌群落、维持厌氧氨氧化体系的稳定提供指导。
本文对厌氧氨氧化细菌种类、特征、代谢及与群落内其他共生菌之间的互作关系进行了较为系统地梳理,认为以下几方面值得后续关注:
(1) 探寻新的技术手段以实现厌氧氨氧化细菌的纯培养仍是厌氧氨氧化细菌研究的重点内容。使用特定选择性培养基,控制厌氧、温度、pH等条件,增加或减少特定生长因子等,是分离培养厌氧氨氧化细菌的一种策略。此外,有些微生物生命活动所必需的分子或电子在群落内不同微生物之间可以进行直接交换,这种微生物的种间依赖关系称为共养。对于共养微生物,可以用组合的方式进行细胞分选,从而建立稳定的共培养体系来实现目标微生物的分离。目前对厌氧氨氧化细菌及其群落内的共生细菌研究较少,厌氧氨氧化细菌是否也存在与之共养的细菌,还需要开展进一步的研究,从而为之后的分离培养提供方向。
(2) 厌氧氨氧化群落内细菌之间存在物质代谢的协同作用,群体感应是调控菌群代谢的重要途径。厌氧氨氧化群落内是否存在尚未发现的信号分子和群体感应类型,不同信号分子是否存在未知的协同作用仍然需要进一步研究。后续研究可以将宏基因组、宏转录组、代谢组结合起来,多角度分析细菌之间物质协同利用和信号分子调控的原理和机制,从而加深对细菌之间物质代谢协同作用和群体感应调控作用的理解。
(3) 厌氧氨氧化群落中的细菌间物质代谢联系和信息交流方式对群落稳定性的影响及对厌氧氨氧化体系氮代谢性能的作用值得进一步研究,例如可以通过比较不同厌氧氨氧化体系对外部冲击的耐受程度和遭受冲击而崩溃后体系的恢复能力这两方面差异,来深入探究影响厌氧氨氧化群落稳定性的因素,进而为构建稳定高效的厌氧氨氧化脱氮体系提供新的思路和方法,这对厌氧氨氧化脱氮能够实现广泛应用具有重要的现实意义。
| [1] |
Broda E. Two kinds of lithotrophs missing in nature[J]. Zeitschrift Für Allgemeine Mikrobiologie, 1977, 17(6): 491-493. DOI:10.1002/jobm.19770170611 |
| [2] |
Hamm RE, Thompson TG. Dissolved nitrogen in the sea water of the northeast Pacific with notes on the total carbon dioxide and the dissolved oxygen[J]. Journal of Marine Research, 1941, 11-27. |
| [3] |
van de Graaf AA, De Bruijn P, Robertson LA, Jetten MSM, Kuenen JG. Autotrophic growth of anaerobic ammonium-oxidizing micro-organisms in a fluidized bed reactor[J]. Microbiology, 1996, 142(8): 2187-2196. DOI:10.1099/13500872-142-8-2187 |
| [4] |
Strous M, Fuerst JA, Kramer EHM, Logemann S, Muyzer G, Van De Pas-Schoonen KT, Webb R, Kuenen JG, Jetten MSM. Missing lithotroph identified as new planctomycete[J]. Nature, 1999, 400(6743): 446-449. DOI:10.1038/22749 |
| [5] |
Strous M, Kuenen JG, Jetten MSM. Key physiology of anaerobic ammonium oxidation[J]. Applied and Environmental Microbiology, 1999, 65(7): 3248-3250. DOI:10.1128/AEM.65.7.3248-3250.1999 |
| [6] |
Hu ZY, Wessels HJCT, Van Alen T, Jetten MSM, Kartal B. Nitric oxide-dependent anaerobic ammonium oxidation[J]. Nature Communications, 2019, 10: 1244. DOI:10.1038/s41467-019-09268-w |
| [7] |
Jetten MSM, Logemann S, Muyzer G, Robertson LA, De Vries S, Van Loosdrecht MCM, Kuenen JG. Novel principles in the microbial conversion of nitrogen compounds[J]. Antonie Van Leeuwenhoek, 1997, 71: 75-93. DOI:10.1023/A:1000150219937 |
| [8] |
Kartal B, Van Niftrik L, Keltjens JT, Op Den Camp HJM, Jetten MSM. Anammox- growth physiology, cell biology, and metabolism[J]. Advances in Microbial Physiology, 2012, 60: 211-262. |
| [9] |
Strous M, Van Gerven E, Zheng P, Kuenen JG, Jetten MSM. Ammonium removal from concentrated waste streams with the anaerobic ammonium oxidation (anammox) process in different reactor configurations[J]. Water Research, 1997, 31(8): 1955-1962. DOI:10.1016/S0043-1354(97)00055-9 |
| [10] |
Thamdrup B, Dalsgaard T. Production of N2 through anaerobic ammonium oxidation coupled to nitrate reduction in marine sediments[J]. Applied and Environmental Microbiology, 2002, 68(3): 1312-1318. DOI:10.1128/AEM.68.3.1312-1318.2002 |
| [11] |
Zhang Y, Ruan XH, Op Den Camp HJM, Smits TJM, Jetten MSM, Schmid MC. Diversity and abundance of aerobic and anaerobic ammonium-oxidizing bacteria in freshwater sediments of the Xinyi River (China)[J]. Environmental Microbiology, 2007, 9(9): 2375-2382. DOI:10.1111/j.1462-2920.2007.01357.x |
| [12] |
Schubert CJ, Durisch-Kaiser E, Wehrli B, Thamdrup B, Lam P, Kuypers MMM. Anaerobic ammonium oxidation in a tropical freshwater system (Lake Tanganyika)[J]. Environmental Microbiology, 2006, 8(10): 1857-1863. DOI:10.1111/j.1462-2920.2006.01074.x |
| [13] |
Jaeschke A, Op Den Camp HJM, Harhangi H, Klimiuk A, Hopmans EC, Jetten MSM, Schouten S, Sinninghe Damsté JS. 16S rRNA gene and lipid biomarker evidence for anaerobic ammonium-oxidizing bacteria (anammox) in California and Nevada hot springs[J]. FEMS Microbiology Ecology, 2009, 67: 343-350. DOI:10.1111/j.1574-6941.2008.00640.x |
| [14] |
Zhu GB, Wang SY, Feng XJ, Fan GN, Jetten MSM, Yin CQ. Anammox bacterial abundance, biodiversity and activity in a constructed wetland[J]. Environmental Science & Technology, 2011, 45(23): 9951-9958. |
| [15] |
Ding BJ, Zhang H, Luo WQ, Sun SY, Cheng F, Li ZK. Nitrogen loss through denitrification, anammox and Feammox in a paddy soil[J]. Science of the Total Environment, 2021, 773: 145601. DOI:10.1016/j.scitotenv.2021.145601 |
| [16] |
Wang SY, Peng YZ, Ma B, Wang SY, Zhu GB. Anaerobic ammonium oxidation in traditional municipal wastewater treatment plants with low-strength ammonium loading: widespread but overlooked[J]. Water Research, 2015, 84: 66-75. DOI:10.1016/j.watres.2015.07.005 |
| [17] |
Kartal B, Keltjens JT. Anammox biochemistry: a tale of heme c proteins[J]. Trends in Biochemical Sciences, 2016, 41(12): 998-1011. DOI:10.1016/j.tibs.2016.08.015 |
| [18] |
Arrigo KR. Marine microorganisms and global nutrient cycles[J]. Nature, 2005, 437(7057): 349-355. DOI:10.1038/nature04159 |
| [19] |
Fuerst JA, Sagulenko E. Beyond the bacterium: Planctomycetes challenge our concepts of microbial structure and function[J]. Nature Reviews Microbiology, 2011, 9(6): 403-413. DOI:10.1038/nrmicro2578 |
| [20] |
Jia FX, Peng YZ, Li JW, Li XY, Yao H. Metagenomic prediction analysis of microbial aggregation in anammox-dominated community[J]. Water Environment Research, 2021. |
| [21] |
Strous M, Pelletier E, Mangenot S, Rattei T, Lehner A, Taylor MW, Horn M, Daims H, Bartol-Mavel D, Wincker P, et al. Deciphering the evolution and metabolism of an anammox bacterium from a community genome[J]. Nature, 2006, 440(7085): 790-794. DOI:10.1038/nature04647 |
| [22] |
Schmid M, Walsh K, Webb R, Rijpstra WI, Van De Pas-Schoonen K, Verbruggen MJ, Hill T, Moffett B, Fuerst J, Schouten S, et al. Candidatus "Scalindua brodae", sp. nov., Candidatus "Scalindua wagneri" sp. nov., two new species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2003, 26(4): 529-538. DOI:10.1078/072320203770865837 |
| [23] |
Kartal B, Rattray J, Van Niftrik LA, Van De Vossenberg J, Schmid MC, Webb RI, Schouten S, Fuerst JA, Damsté JS, Jetten MSM, et al. Candidatus "Anammoxoglobus propionicus" a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2007, 30(1): 39-49. DOI:10.1016/j.syapm.2006.03.004 |
| [24] |
Quan ZX, Rhee SK, Zuo JE, Yang Y, Bae JW, Park JR, Lee ST, Park YH. Diversity of ammonium-oxidizing bacteria in a granular sludge anaerobic ammonium-oxidizing (anammox) reactor[J]. Environmental Microbiology, 2008, 10(11): 3130-3139. DOI:10.1111/j.1462-2920.2008.01642.x |
| [25] |
Khramenkov SV, Kozlov MN, Kevbrina MV, Dorofeev AG, Kazakova EA, Grachev VA, Kuznetsov BB, Polyakov DY, Nikolaev YA. A novel bacterium carrying out anaerobic ammonium oxidation in a reactor for biological treatment of the filtrate of wastewater fermented sludge[J]. Microbiology, 2013, 82(5): 628-636. DOI:10.1134/S002626171305007X |
| [26] |
Viancelli A, Kunz A, Esteves PA, Nishiyama T, Fujii T, Vanotti M, Antonio RV. Analysis of bacterial community from a sludge reactor with anammox activity[J]. I Simpósio Internacional sobre Gerenciamento de Resíduos de Animais Tratamento de Dejetos de Animais, 2009, March: 11-13
|
| [27] |
Viancelli A, Kunz A, Esteves PA, Bauermann FV, Furukawa K, Fujii T, Antônio RV, Vanotti M. Bacterial biodiversity from an anaerobic up flow bioreactor with ANAMMOX activity inoculated with swine sludge[J]. Brazilian Archives of Biology and Technology, 2011, 54(5): 1035-1041. DOI:10.1590/S1516-89132011000500022 |
| [28] |
Kartal B, van Niftrik L, Rattray J, van de Vossenberg JLCM, Schmid MC, Sinninghe Damsté J, Jetten MSM, Strous M. Candidatus 'Brocadia fulgida': an autofluorescent anaerobic ammonium oxidizing bacterium[J]. FEMS Microbiology Ecology, 2008, 63(1): 46-55. DOI:10.1111/j.1574-6941.2007.00408.x |
| [29] |
van Niftrik L, Geerts WJC, van Donselaar EG, Humbel BM, Webb RI, Fuerst JA, Verkleij AJ, Jetten MSM, Strous M. Linking ultrastructure and function in four genera of anaerobic ammonium-oxidizing bacteria: cell plan, glycogen storage, and localization of cytochrome c proteins[J]. Journal of Bacteriology, 2008, 190(2): 708-717. DOI:10.1128/JB.01449-07 |
| [30] |
Niftrik L. Cell biology of unique anammox bacteria that contain an energy conserving prokaryotic organelle[J]. Antonie Van Leeuwenhoek, 2013, 104(4): 489-497. DOI:10.1007/s10482-013-9990-5 |
| [31] |
Oshiki M, Shimokawa M, Fujii N, Satoh H, Okabe S. Physiological characteristics of the anaerobic ammonium-oxidizing bacterium 'Candidatus Brocadia sinica'[J]. Microbiology, 2011, 157(6): 1706-1713. DOI:10.1099/mic.0.048595-0 |
| [32] |
Kuenen JG. Anammox and beyond[J]. Environmental Microbiology, 2020, 22(2): 525-536. DOI:10.1111/1462-2920.14904 |
| [33] |
van Niftrik L, Geerts WJC, van Donselaar EG, Humbel BM, Yakushevska A, Verkleij AJ, Jetten MSM, Strous M. Combined structural and chemical analysis of the anammoxosome: a membrane-bounded intracytoplasmic compartment in anammox bacteria[J]. Journal of Structural Biology, 2008, 161(3): 401-410. DOI:10.1016/j.jsb.2007.05.005 |
| [34] |
Lindsay MR, Webb RI, Strous M, Jetten MS, Butler MK, Forde RJ, Fuerst JA. Cell compartmentalisation in planctomycetes: novel types of structural organisation for the bacterial cell[J]. Archives of Microbiology, 2001, 175(6): 413-429. DOI:10.1007/s002030100280 |
| [35] |
Jetten MSM, Niftrik LV, Strous M, Kartal B, Keltjens JT, Op Den Camp HJM. Biochemistry and molecular biology of anammox bacteria[J]. Critical Reviews in Biochemistry and Molecular Biology, 2009, 44(2-3): 65-84. DOI:10.1080/10409230902722783 |
| [36] |
Karlsson R, Karlsson A, Bäckman O, Johansson BR, Hulth S. Identification of key proteins involved in the anammox reaction[J]. FEMS Microbiology Letters, 2009, 297(1): 87-94. DOI:10.1111/j.1574-6968.2009.01677.x |
| [37] |
van Niftrik L, van Helden M, Kirchen S, van Donselaar EG, Harhangi HR, Webb RI, Fuerst JA, den Camp HJMO, Jetten MSM, Strous M. Intracellular localization of membrane-bound ATPases in the compartmentalized anammox bacterium 'Candidatus Kuenenia stuttgartiensis'[J]. Molecular Microbiology, 2010, 77(3): 701-715. DOI:10.1111/j.1365-2958.2010.07242.x |
| [38] |
Kuypers MMM, Sliekers AO, Lavik G, Schmid M, Jørgensen BB, Kuenen JG, Sinninghe Damsté JS, Strous M, Jetten MSM. Anaerobic ammonium oxidation by anammox bacteria in the Black Sea[J]. Nature, 2003, 422(6932): 608-611. DOI:10.1038/nature01472 |
| [39] |
Wu GX, Zhang TQ, Gu MQ, Chen Z, Yin QD. Review of characteristics of anammox bacteria and strategies for anammox start-up for sustainable wastewater resource management[J]. Water Science and Technology, 2020, 82(9): 1742-1757. DOI:10.2166/wst.2020.443 |
| [40] |
Strous M, Van Gerven E, Kuenen JG, Jetten M. Effects of aerobic and microaerobic conditions on anaerobic ammonium-oxidizing (anammox) sludge[J]. Applied and Environmental Microbiology, 1997, 63(6): 2446-2448. DOI:10.1128/aem.63.6.2446-2448.1997 |
| [41] |
Egli K, Fanger U, Alvarez PJJ, Siegrist H, Van Der Meer JR, Zehnder AJB. Enrichment and characterization of an anammox bacterium from a rotating biological contactor treating ammonium-rich leachate[J]. Archives of Microbiology, 2001, 175(3): 198-207. DOI:10.1007/s002030100255 |
| [42] |
Su Y, Yu PF, Fu JX, Zhou MJ, Zhao HQ, Wang GQ. Research on the influences of metallic ions concentrations on the granulation of ANAMMOX sludge[J]. Industrial Water Treatment, 2016, 36(3): 74-77, 108. (in Chinese) 苏杨, 于鹏飞, 傅金祥, 周明俊, 赵鹤谦, 王国强. 金属离子浓度对ANAMMOX污泥颗粒化的影响研究[J]. 工业水处理, 2016, 36(3): 74-77, 108. |
| [43] |
Dapena-Mora A, Fernández I, Campos JL, Mosquera-Corral A, Méndez R, Jetten MSM. Evaluation of activity and inhibition effects on Anammox process by batch tests based on the nitrogen gas production[J]. Enzyme and Microbial Technology, 2007, 40(4): 859-865. DOI:10.1016/j.enzmictec.2006.06.018 |
| [44] |
Shen MY, Wu LN, Li Z, Peng YZ, Zhang SJ, Yang AM. Research and application progress of anaerobic ammonium oxidation in wastewater treatment[J]. China Water & Wastewater, 2019, 35(6): 16-21. (in Chinese) 沈明玉, 吴莉娜, 李志, 彭永臻, 张树军, 杨岸明. 厌氧氨氧化在废水处理中的研究及应用进展[J]. 中国给水排水, 2019, 35(6): 16-21. |
| [45] |
Oshiki M, Satoh H, Okabe S. Ecology and physiology of anaerobic ammonium oxidizing bacteria[J]. Environmental Microbiology, 2016, 18(9): 2784-2796. DOI:10.1111/1462-2920.13134 |
| [46] |
Dosta J, Fernández I, Vázquez-Padín JR, Mosquera-Corral A, Campos JL, Mata-Álvarez J, Méndez R. Short- and long-term effects of temperature on the Anammox process[J]. Journal of Hazardous Materials, 2008, 154(1-3): 688-693. DOI:10.1016/j.jhazmat.2007.10.082 |
| [47] |
Zhu GB, Peng YZ, Li BK, Guo JH, Yang Q, Wang SY. Biological Removal of Nitrogen from Wastewater[M]. Reviews of Environmental Contamination and Toxicology. New York, NY: Springer New York, 2008: 159-195
|
| [48] |
Huo TR. Metabolic responses of anammox consortia to low temperature[D]. Beijing: Master's Thesis of Peking University, 2020 (in Chinese) 霍唐燃. 厌氧氨氧化菌群对低温的代谢响应机制研究[D]. 北京: 北京大学硕士学位论文, 2020 |
| [49] |
Dong ZY, Hu BL, Han JH. Research progress in the uptake and utilization of iron by the anaerobic ammonium-oxidizing bacterium Candidatus Kuenenia stuttgartiensis[J]. Microbiology China, 2021, 48(5): 1780-1787. (in Chinese) 董子阳, 胡宝兰, 韩佳慧. 厌氧氨氧化细菌Candidatus Kuenenia stuttgartiensis铁的吸收利用研究进展[J]. 微生物学通报, 2021, 48(5): 1780-1787. |
| [50] |
Chen CZ, Ereaihan, Liu HG, Wang JK, Lan MJ, Liu SB, Du KQ, Li JF. Effect of iron on anammox process and nitrogen removal performance: a review[J]. Environmental Science & Technology, 2021, 44(5): 14-24. (in Chinese) 陈翠忠, 额热艾汗, 刘洪光, 王健康, 蓝明菊, 刘生宝, 杜可清, 李俊峰. 铁对厌氧氨氧化过程及脱氮性能的影响[J]. 环境科学与技术, 2021, 44(5): 14-24. |
| [51] |
Chen H, Yu JJ, Jia XY, Jin RC. Enhancement of anammox performance by Cu(II), Ni(II) and Fe(III) supplementation[J]. Chemosphere, 2014, 117: 610-616. DOI:10.1016/j.chemosphere.2014.09.047 |
| [52] |
Wang Y, Ji XM, Jin RC. How anammox responds to the emerging contaminants: status and mechanisms[J]. Journal of Environmental Management, 2021, 293: 112906. DOI:10.1016/j.jenvman.2021.112906 |
| [53] |
van de Graaf AA, de Bruijn P, Robertson LA, Jetten MSM, Kuenen JG. Metabolic pathway of anaerobic ammonium oxidation on the basis of 15N studies in a fluidized bed reactor[J]. Microbiology, 1997, 143(7): 2415-2421. DOI:10.1099/00221287-143-7-2415 |
| [54] |
Jetten MSM, Wagner M, Fuerst J, Van Loosdrecht M, Kuenen G, Strous M. Microbiology and application of the anaerobic ammonium oxidation ('anammox') process[J]. Current Opinion in Biotechnology, 2001, 12(3): 283-288. DOI:10.1016/S0958-1669(00)00211-1 |
| [55] |
Kartal B, Maalcke WJ, De Almeida NM, Cirpus I, Gloerich J, Geerts W, Op Den Camp HJM, Harhangi HR, Janssen-Megens EM, Francoijs KJ, et al. Molecular mechanism of anaerobic ammonium oxidation[J]. Nature, 2011, 479(7371): 127-130. DOI:10.1038/nature10453 |
| [56] |
Allen JWA, Barker PD, Daltrop O, Stevens JM, Tomlinson EJ, Sinha N, Sambongi Y, Ferguson SJ. Why isn't 'standard' heme good enough for c-type and d1-type cytochromes?[J]. Dalton Transactions, 2005(21): 3410-3418. DOI:10.1039/b508139b |
| [57] |
Helen D, Kim H, Tytgat B, Anne W. Highly diverse nirK genes comprise two major clades that harbour ammonium-producing denitrifiers[J]. BMC Genomics, 2016, 17: 155. DOI:10.1186/s12864-016-2465-0 |
| [58] |
van de Vossenberg J, Woebken D, Maalcke WJ, Wessels HJCT, Dutilh BE, Kartal B, Janssen-Megens EM, Roeselers G, Yan J, Speth D, et al. The metagenome of the marine anammox bacterium 'Candidatus Scalindua profunda' illustrates the versatility of this globally important nitrogen cycle bacterium[J]. Environmental Microbiology, 2013, 15(5): 1275-1289. DOI:10.1111/j.1462-2920.2012.02774.x |
| [59] |
de Almeida NM, Wessels HJCT, de Graaf RM, Ferousi C, Jetten MSM, Keltjens JT, Kartal B. Membrane-bound electron transport systems of an anammox bacterium: a complexome analysis[J]. Biochimica et Biophysica Acta (BBA)-Bioenergetics, 2016, 1857(10): 1694-1704. DOI:10.1016/j.bbabio.2016.07.006 |
| [60] |
Hira D, Toh H, Migita CT, Okubo H, Nishiyama T, Hattori M, Furukawa K, Fujii T. Anammox organism KSU-1 expresses a NirK-type copper-containing nitrite reductase instead of a NirS-type with cytochrome cd1[J]. FEBS Letters, 2012, 586(11): 1658-1663. DOI:10.1016/j.febslet.2012.04.041 |
| [61] |
Hu ZY, Speth DR, Francoijs KJ, Quan ZX, Jetten MSM. Metagenome analysis of a complex community reveals the metabolic blueprint of anammox bacterium "Candidatus Jettenia asiatica"[J]. Frontiers in Microbiology, 2012, 3: 366. |
| [62] |
Oshiki M, Ali M, Shinyako-Hata K, Satoh H, Okabe S. Hydroxylamine-dependent anaerobic ammonium oxidation (anammox) by "Candidatus Brocadia sinica"[J]. Environmental Microbiology, 2016, 18(9): 3133-3143. DOI:10.1111/1462-2920.13355 |
| [63] |
Gori F, Tringe SG, Kartal B, Marchiori E, Jetten MSM. The metagenomic basis of anammox metabolism in Candidatus 'Brocadia fulgida'[J]. Biochemical Society Transactions, 2011, 39(6): 1799-1804. DOI:10.1042/BST20110707 |
| [64] |
Ali M, Haroon MF, Narita Y, Zhang L, Rangel Shaw D, Okabe S, Saikaly PE. Draft genome sequence of the anaerobic ammonium-oxidizing bacterium "Candidatus Brocadia sp. 40"[J]. Genome Announcements, 2016, 4(6). |
| [65] |
Lawson CE, Wu S, Bhattacharjee AS, Hamilton JJ, McMahon KD, Goel R, Noguera DR. Metabolic network analysis reveals microbial community interactions in anammox granules[J]. Nature Communications, 2017, 8: 15416. DOI:10.1038/ncomms15416 |
| [66] |
Kartal B, Kuypers MMM, Lavik G, Schalk J, Op Den Camp HJMO, Jetten MSM, Strous M. Anammox bacteria disguised as denitrifiers: nitrate reduction to dinitrogen gas via nitrite and ammonium[J]. Environmental Microbiology, 2007, 9(3): 635-642. DOI:10.1111/j.1462-2920.2006.01183.x |
| [67] |
Wang C, Qiao S, Bi Z, Zhou JT. Nitrate removal by anammox biomass with intracellular carbon source as electron donors via DNRA pathway[J]. Environmental Research, 2021, 200: 111390. DOI:10.1016/j.envres.2021.111390 |
| [68] |
Castro-Barros CM, Jia MS, Van Loosdrecht MCM, Volcke EIP, Winkler MKH. Evaluating the potential for dissimilatory nitrate reduction by anammox bacteria for municipal wastewater treatment[J]. Bioresource Technology, 2017, 233: 363-372. DOI:10.1016/j.biortech.2017.02.063 |
| [69] |
Liu YY, Wang S, Li SZ, Deng Y. Advances in molecular ecology on microbial functional genes of carbon cycle[J]. Microbiology China, 2017, 44(7): 1676-1689. (in Chinese) 刘洋荧, 王尚, 厉舒祯, 邓晔. 基于功能基因的微生物碳循环分子生态学研究进展[J]. 微生物学通报, 2017, 44(7): 1676-1689. |
| [70] |
Schouten S, Strous M, Kuypers MMM, Rijpstra WIC, Baas M, Schubert CJ, Jetten MSM, Sinninghe Damsté JS. Stable carbon isotopic fractionations associated with inorganic carbon fixation by anaerobic ammonium-oxidizing bacteria[J]. Applied and Environmental Microbiology, 2004, 70(6): 3785-3788. DOI:10.1128/AEM.70.6.3785-3788.2004 |
| [71] |
Song Y, Lee JS, Shin J, Lee GM, Jin S, Kang S, Lee JK, Kim DR, Lee EY, Kim SC, et al. Functional cooperation of the glycine synthase-reductase and Wood-Ljungdahl pathways for autotrophic growth of Clostridium drakei[J]. PNAS, 2020, 117(13): 7516-7523. DOI:10.1073/pnas.1912289117 |
| [72] |
Liu L, Ming YZ, Lv AP, Jiao JY, Li WJ. Recent advance on the anaerobic ammonium oxidation bacteria[J]. Acta Microbiologica Sinica, 2021, 61(4): 969-986. (in Chinese) 刘兰, 明语真, 吕爱萍, 焦建宇, 李文均. 厌氧氨氧化细菌的研究进展[J]. 微生物学报, 2021, 61(4): 969-986. |
| [73] |
Zhang TQ, Wei N, Wu GX. Autotrophic nitrogen removal and potential microbial interactions in anammox systems with different ammonia and organic carbon concentrations[J]. Journal of Water Process Engineering, 2020, 37: 101493. DOI:10.1016/j.jwpe.2020.101493 |
| [74] |
Zhang TQ, Yin QD, Shi YH, Wu GX. Microbial physiology and interactions in anammox systems with the intermittent addition of organic carbons[J]. Bioresource Technology, 2021, 319: 124226. DOI:10.1016/j.biortech.2020.124226 |
| [75] |
Zhang L, Zheng P. Metabolism of anaerobic ammonium oxidation (anammox) bacteria[J]. Bulletin of Science and Technology, 2010, 26(6): 931-937. (in Chinese) 张蕾, 郑平. 厌氧氨氧化菌的代谢[J]. 科技通报, 2010, 26(6): 931-937. DOI:10.3969/j.issn.1001-7119.2010.06.026 |
| [76] |
Kartal B, De Almeida NM, Maalcke WJ, Op Den Camp HJM, Jetten MSM, Keltjens JT. How to make a living from anaerobic ammonium oxidation[J]. FEMS Microbiology Reviews, 2013, 37(3): 428-461. DOI:10.1111/1574-6976.12014 |
| [77] |
Feng Y, Zhao YP, Jiang B, Zhao HZ, Wang QL, Liu ST. Discrepant gene functional potential and cross-feedings of anammox bacteria Ca. Jettenia caeni and Ca. Brocadia sinica in response to acetate[J]. Water Research, 2019, 165: 114974. DOI:10.1016/j.watres.2019.114974 |
| [78] |
Wu Y. Study of community structure and functional composition of anammox cross-feeding bacterial and their response under nitrite shock[D]. Beijing: Doctoral Dissertation of Peking University, 2020 (in Chinese) 吴扬. 厌氧氨氧化细菌互养微生物群落与功能组成及其对亚硝氮冲击的相应机制研究[D]. 北京: 北京大学博士学位论文, 2020 |
| [79] |
Tao Y, Huang XL, Gao DW, Wang XL, Chen CH, Liang H, Van Loosdrecht MCM. NanoSIMS reveals unusual enrichment of acetate and propionate by an anammox consortium dominated by Jettenia asiatica[J]. Water Research, 2019, 159: 223-232. DOI:10.1016/j.watres.2019.05.006 |
| [80] |
Zhang LY, Qiao XJ, Yu K. Influence of propionate on performance and community structure of an anammox reactor[J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2021, 57(3): 545-555. (in Chinese) 张立羽, 乔雪姣, 余珂. 丙酸盐对厌氧氨氧化除氮性能及群落结构的影响[J]. 北京大学学报: 自然科学版, 2021, 57(3): 545-555. |
| [81] |
Wu Y, Wang YX, De Costa YG, Tong ZD, Cheng JJ, Zhou LJ, Zhuang WQ, Yu K. The co-existence of anammox genera in an expanded granular sludge bed reactor with biomass carriers for nitrogen removal[J]. Applied Microbiology and Biotechnology, 2019, 103(3): 1231-1242. DOI:10.1007/s00253-018-9494-3 |
| [82] |
Speth DR, Zandt MHI, Guerrero-Cruz S, Dutilh BE, Jetten MSM. Genome-based microbial ecology of anammox granules in a full-scale wastewater treatment system[J]. Nature Communications, 2016, 7: 11172. DOI:10.1038/ncomms11172 |
| [83] |
Kuypers MMM, Marchant HK, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [84] |
Lü XT, Jiang Y, Meng CL, Zhang SJ, Gu PC, Yang AM, Wang JW, Han XY. Inhibitory of FNA on the activity of AOB and NOB under aerobic and anoxic conditions[J]. Microbiology China, 2019, 46(8): 1927-1935. (in Chinese) 吕心涛, 蒋勇, 孟春霖, 张树军, 谷鹏超, 杨岸明, 王佳伟, 韩晓宇. 好氧和缺氧条件下游离亚硝酸对氨氧化菌和亚硝酸盐氧化菌的选择性抑制[J]. 微生物学通报, 2019, 46(8): 1927-1935. |
| [85] |
Zhang BY, Yu K. Application of microbial gene databases in the annotation of nitrogen cycle functional genes[J]. Microbiology China, 2020, 47(9): 3021-3038. (in Chinese) 张博雅, 余珂. 微生物基因数据库在氮循环功能基因注释中的应用[J]. 微生物学通报, 2020, 47(9): 3021-3038. |
| [86] |
Fowler SJ, Palomo A, Dechesne A, Mines PD, Smets BF. Comammox Nitrospira are abundant ammonia oxidizers in diverse groundwater-fed rapid sand filter communities[J]. Environmental Microbiology, 2018, 20(3): 1002-1015. DOI:10.1111/1462-2920.14033 |
| [87] |
Shibata H, Tatara M, Ueno Y. Enhanced nitrogen removal by a combination of anammox and heterotrophic denitrification[J]. Water and Environment Journal, 2021, 35(2): 740-747. DOI:10.1111/wej.12666 |
| [88] |
Keren R, Lawrence JE, Zhuang WQ, Jenkins D, Banfield JF, Alvarez-Cohen L, Zhou LJ, Yu K. Increased replication of dissimilatory nitrate-reducing bacteria leads to decreased anammox bioreactor performance[J]. Microbiome, 2020, 8(1): 7. DOI:10.1186/s40168-020-0786-3 |
| [89] |
Jia MS, Winkler MKH, Volcke EIP. Elucidating the competition between heterotrophic denitrification and DNRA using the resource-ratio theory[J]. Environmental Science & Technology, 2020, 54(21): 13953-13962. |
| [90] |
Wang YL, Niu QG, Zhang X, Liu L, Wang YB, Chen YQ, Negi M, Figeys D, Li YY, Zhang T. Exploring the effects of operational mode and microbial interactions on bacterial community assembly in a one-stage partial-nitritation anammox reactor using integrated multi-omics[J]. Microbiome, 2019, 7(1): 122. DOI:10.1186/s40168-019-0730-6 |
| [91] |
Engler K, Zaramela LS. The social network of microorganisms—how auxotrophies shape complex communities[J]. Nature Reviews Microbiology, 2018, 16(6): 383-390. DOI:10.1038/s41579-018-0004-5 |
| [92] |
Liao C, Wang T, Maslov S, Xavier JB. Modeling microbial cross-feeding at intermediate scale portrays community dynamics and species coexistence[J]. PLoS Computational Biology, 2020, 16(8): e1008135. DOI:10.1371/journal.pcbi.1008135 |
| [93] |
Cotton CA, Edlich-Muth C, Bar-Even A. Reinforcing carbon fixation: CO2 reduction replacing and supporting carboxylation[J]. Current Opinion in Biotechnology, 2018, 49: 49-56. DOI:10.1016/j.copbio.2017.07.014 |
| [94] |
Wang YJ, Hu XM, Jiang BH, Song ZH, Ma YG. Symbiotic relationship analysis of predominant bacteria in a lab-scale anammox UASB bioreactor[J]. Environmental Science and Pollution Research, 2016, 23(8): 7615-7626. DOI:10.1007/s11356-015-6016-z |
| [95] |
Zhao YP, Liu SF, Jiang B, Feng Y, Zhu TT, Tao HC, Tang X, Liu ST. Genome-centered metagenomics analysis reveals the symbiotic organisms possessing ability to cross-feed with anammox bacteria in anammox consortia[J]. Environmental Science & Technology, 2018, 52(19): 11285-11296. |
| [96] |
Chen TT, Zheng P, Shen LD. Growth and metabolism characteristics of anaerobic ammonium-oxidizing bacteria aggregates[J]. Applied Microbiology and Biotechnology, 2013, 97(12): 5575-5583. DOI:10.1007/s00253-012-4346-z |
| [97] |
Li JY, Du QP, Peng HQ, Zhang YL, Bi YQ, Shi Y, Xu YB, Liu T. Optimization of biochemical oxygen demand to total nitrogen ratio for treating landfill leachate in a single-stage partial nitrification- denitrification system[J]. Journal of Cleaner Production, 2020, 266: 121809. DOI:10.1016/j.jclepro.2020.121809 |
| [98] |
Adav SS, Lee DJ, Tay JH. Extracellular polymeric substances and structural stability of aerobic granule[J]. Water Research, 2008, 42(6/7): 1644-1650. |
| [99] |
Laspidou CS, Rittmann BE. A unified theory for extracellular polymeric substances, soluble microbial products, and active and inert biomass[J]. Water Research, 2002, 36(11): 2711-2720. DOI:10.1016/S0043-1354(01)00413-4 |
| [100] |
Nouha K, Kumar RS, Balasubramanian S, Tyagi RD. Critical review of EPS production, synthesis and composition for sludge flocculation[J]. Journal of Environmental Sciences, 2018, 66: 225-245. DOI:10.1016/j.jes.2017.05.020 |
| [101] |
Swift S, Throup JP, Williams P, Salmond GPC, Stewart GSAB. Quorum sensing: a population-density component in the determination of bacterial phenotype[J]. Trends in Biochemical Sciences, 1996, 21(6): 214-219. DOI:10.1016/S0968-0004(96)80018-1 |
| [102] |
Waters CM, Bassler BL. Quorum sensing: cell-to-cell communication in bacteria[J]. Annual Review of Cell and Developmental Biology, 2005, 21: 319-346. DOI:10.1146/annurev.cellbio.21.012704.131001 |
| [103] |
Goo E, An JH, Kang Y, Hwang I. Control of bacterial metabolism by quorum sensing[J]. Trends in Microbiology, 2015, 23(9): 567-576. DOI:10.1016/j.tim.2015.05.007 |
| [104] |
Ding S, Zheng P, Zhang M, Lu HF. Quorum sensing in anaerobic ammonium oxidation bacteria[J]. Acta Ecologica Sinica, 2012, 32(8): 2581-2587. (in Chinese) 丁爽, 郑平, 张萌, 陆慧锋. 厌氧氨氧化菌群体感应系统研究[J]. 生态学报, 2012, 32(8): 2581-2587. |
| [105] |
Tang X, Guo YZ, Zhu TT, Tao HC, Liu ST. Identification of quorum sensing signal AHLs synthases in Candidatus Jettenia caeni and their roles in anammox activity[J]. Chemosphere, 2019, 225: 608-617. DOI:10.1016/j.chemosphere.2019.02.192 |
| [106] |
Deng YY, Wu JE, Tao F, Zhang LH. Listening to a new language: DSF-based quorum sensing in Gram-negative bacteria[J]. Chemical Reviews, 2011, 111(1): 160-173. DOI:10.1021/cr100354f |
| [107] |
Hengge R. Principles of c-di-GMP signalling in bacteria[J]. Nature Reviews Microbiology, 2009, 7(4): 263-273. DOI:10.1038/nrmicro2109 |
| [108] |
Ma YW, Sundar S, Park H, Chandran K. The effect of inorganic carbon on microbial interactions in a biofilm nitritation-anammox process[J]. Water Research, 2015, 70: 246-254. DOI:10.1016/j.watres.2014.12.006 |
| [109] |
Liu YM, Guo JB, Lian J, Chen Z, Li YJ, Xing YJ, Wang T. Effects of extracellular polymeric substances (EPS) and N-acyl-L-homoserine lactones (AHLs) on the activity of anammox biomass[J]. International Biodeterioration & Biodegradation, 2018, 129: 141-147. |
| [110] |
Tang X, Liu ST, Zhang ZT, Zhuang GQ. Identification of the release and effects of AHLs in anammox culture for bacteria communication[J]. Chemical Engineering Journal, 2015, 273: 184-191. DOI:10.1016/j.cej.2015.03.045 |
| [111] |
Chen SH, Li AJ, Wang YX, Wu LJ. Identification and influence of quorum sensing on anaerobic ammonium oxidation process[J]. Environmental Science, 2017, 38(3): 1137-1143. (in Chinese) 陈舒涵, 李安婕, 王越兴, 吴玲娟. 厌氧氨氧化污泥群体感应信号分子检测及影响研究[J]. 环境科学, 2017, 38(3): 1137-1143. |
| [112] |
Ryan RP, Dow JM. Communication with a growing family: diffusible signal factor (DSF) signaling in bacteria[J]. Trends in Microbiology, 2011, 19(3): 145-152. DOI:10.1016/j.tim.2010.12.003 |
| [113] |
Guo YZ, Liu ST, Tang X, Yang FL. Role of c-di-GMP in anammox aggregation and systematic analysis of its turnover protein in Candidatus Jettenia caeni[J]. Water Research, 2017, 113: 181-190. DOI:10.1016/j.watres.2017.02.018 |
| [114] |
Zhou L, Zhang LH, Cámara M, He YW. The DSF family of quorum sensing signals: diversity, biosynthesis, and turnover[J]. Trends in Microbiology, 2017, 25(4): 293-303. DOI:10.1016/j.tim.2016.11.013 |
| [115] |
Guo YZ, Zhao YP, Tang X, Na TX, Pan JJ, Zhao HZ, Liu ST. Deciphering bacterial social traits via diffusible signal factor (DSF) -mediated public goods in an anammox community[J]. Water Research, 2021, 191: 116802. DOI:10.1016/j.watres.2020.116802 |
| [116] |
Ma HJ, Wang XZ, Zhang Y, Hu HD, Ren HQ, Geng JJ, Ding LL. The diversity, distribution and function of N-acyl-homoserine lactone (AHL) in industrial anaerobic granular sludge[J]. Bioresource Technology, 2018, 247: 116-124. DOI:10.1016/j.biortech.2017.09.043 |
| [117] |
Liu LJ, Ji M, Wang F, Tian ZK, Wang TY, Wang SY, Wang SY, Yan Z. Insight into the short-term effect of fulvic acid on nitrogen removal performance and N-acylated-L-homoserine lactones (AHLs) release in the anammox system[J]. Science of the Total Environment, 2020, 704: 135285. DOI:10.1016/j.scitotenv.2019.135285 |
| [118] |
Li AJ, Hou BL, Li MX. Cell adhesion, ammonia removal and granulation of autotrophic nitrifying sludge facilitated by N-acyl-homoserine lactones[J]. Bioresource Technology, 2015, 196: 550-558. DOI:10.1016/j.biortech.2015.08.022 |
| [119] |
Ma HJ, Ma SJ, Luo WQ, Ding LL, Wang JF, Ren HQ. Long-term exogenous addition of synthetic acyl homoserine lactone enhanced the anaerobic granulation process[J]. Science of the Total Environment, 2019, 696: 133809. DOI:10.1016/j.scitotenv.2019.133809 |
| [120] |
Zhao R, Zhang HM, Zou X, Yang FL. Effects of inhibiting acylated homoserine lactones (AHLs) on anammox activity and stability of granules[J]. Current Microbiology, 2016, 73(1): 108-114. DOI:10.1007/s00284-016-1031-y |
| [121] |
Wang C, Liu ST, Xu XC, Zhao CQ, Yang FL, Wang D. Potential coupling effects of ammonia-oxidizing and anaerobic ammonium-oxidizing bacteria on completely autotrophic nitrogen removal over nitrite biofilm formation induced by the second messenger cyclic diguanylate[J]. Applied Microbiology and Biotechnology, 2017, 101(9): 3821-3828. DOI:10.1007/s00253-016-7981-y |
| [122] |
Tang X, Guo YZ, Jiang B, Liu ST. Metagenomic approaches to understanding bacterial communication during the anammox reactor start-up[J]. Water Research, 2018, 136: 95-103. DOI:10.1016/j.watres.2018.02.054 |
 2022, Vol. 49
2022, Vol. 49




