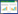扩展功能
文章信息
- 杨梦莹, 谢晨杰, 李时勇, 张红岩, 王一兵, 申乃坤, 姜明国
- YANG Mengying, XIE Chenjie, LI Shiyong, ZHANG Hongyan, WANG Yibing, SHEN Naikun, JIANG Mingguo
- 一株海洋来源铜绿假单胞菌Gxun-7角蛋白酶基因的克隆、表达及重组酶酶学性质
- Cloning and expression of keratinase gene from a marine-derived Pseudomonas aeruginosa Gxun-7 and enzymatic properties of the recombinant enzyme
- 微生物学通报, 2022, 49(11): 4629-4643
- Microbiology China, 2022, 49(11): 4629-4643
- DOI: 10.13344/j.microbiol.china.220270
-
文章历史
- 收稿日期: 2022-03-17
- 接受日期: 2022-05-06
- 网络首发日期: 2022-05-13
角蛋白是一类广泛存在于自然界不溶或微溶于水的硬性蛋白,分子间以大量的二硫键、氢键和疏水相互作用交联形成稳定的α-螺旋和β-折叠结构,难以被常见的蛋白酶(木瓜蛋白酶、胰蛋白酶和胃蛋白酶)水解[1]。羽毛作为家禽加工过程中的副产品之一,其主要成分是角蛋白,如不加以有效利用,不仅会造成资源浪费,还会严重污染环境。因此,将羽毛废弃物转化为可利用的蛋白质资源至关重要。
角蛋白酶可以特异性降解羽毛角蛋白,根据活性中心不同主要分为丝氨酸蛋白酶和金属蛋白酶,在动物饲料[2]、生物肥料[3]、医学[4]、洗涤[5]、制革[6]及环境治理[7]等方面具有重要的应用潜力。利用角蛋白酶对废弃羽毛进行降解,不但条件温和、能耗低、绿色环保,而且降解过程中不会破坏氨基酸,成为目前发展最快、最有前途的羽毛降解方式。角蛋白酶在细菌、真菌和放线菌中均有分布,目前研究较多的角蛋白酶主要来源于细菌,如枯草芽孢杆菌B-3来源的角蛋白酶,酶活力为14.8 U/mL,该酶在50 ℃处理10 min后剩余酶活在20%左右[8];地衣芽孢杆菌CP-16来源的角蛋白酶,在75 ℃处理5 min后相对酶活力仅为30.65%[9];地衣芽孢杆菌HK-1来源的角蛋白酶最适温度为60 ℃,但酶活力会被NaCl、EDTA等试剂强烈抑制[10]。虽然细菌来源的角蛋白酶具有易提取、易纯化和后期处理工艺相对简单等优势,但仍然存在酶活力低、稳定性差、反应条件适用范围窄等系列问题,直接影响角蛋白酶的产业化应用[11-13]。因此亟须探索稳定性好、酶活力高的角蛋白酶,以满足角蛋白酶降解废弃羽毛的产业化需求。
本实验室前期从广西壮族自治区北部湾海鸭养殖场淤泥分离出一株可高效降解羽毛的铜绿假单胞菌Gxun-7,该菌株48 h内可将2.5% (质量体积分数)整根羽毛几乎完全降解,而且分泌的粗酶液具有良好的温度、化学试剂及盐的耐受性,因此推测该酶具有工业应用价值[14]。然而铜绿假单胞菌为条件致病菌,对人类健康及环境污染存在潜在风险,而且目前国内未见关于对铜绿假单胞菌(Pseudomonas aeruginosa)角蛋白酶基因异源表达的研究。因此,本研究从该菌株中克隆出角蛋白酶基因,异源表达于大肠杆菌,利用镍柱对重组角蛋白酶进行纯化并探究其酶学特性,以期为将来的大规模应用及酶分子结构改造研究提供基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒铜绿假单胞菌Gxun-7由本实验室从广西壮族自治区北部湾海鸭养殖场淤泥中筛选获得;质粒pET-22b(+)由本实验室保存;大肠杆菌E. coli Top10和E. coli RosettagamiB (DE3)感受态细胞购自生工生物工程(上海)股份有限公司。
1.1.2 主要试剂和仪器2×SuperStar Plus PCR Mix、快速质粒小提试剂盒、细菌基因组DNA提取试剂盒和DNA纯化回收试剂盒,天根生物科技(北京)有限公司;限制性内切酶Nco I、BamH I及T4 DNA连接酶,北京全式金生物技术有限公司。镍柱,宝日医生物技术(北京)有限公司。
1.2 方法 1.2.1 角蛋白酶基因的克隆根据菌株全基因组测序结果、NCBI已发布的铜绿假单胞菌角蛋白酶基因序列及表达载体pET-22b(+)的多克隆位点,利用Vector NTI软件设计特异性引物kp2-F (5′-CCATGGCCGACCTGATCGACGTGTCCAAACT-3′,下划线为Nco Ⅰ酶切位点)和kp2-R (5′-GGATCCAACGCGCTCGGGCAGGTCACGCCGACG-3′,下划线为BamH Ⅰ酶切位点)。以P. aeruginosa Gxun-7基因组为模板进行PCR扩增。PCR反应体系:2×SuperStar Plus PCR Mix 25 μL,DNA 2 μL,kp2-F/R引物(10 μmol/L)各1 μL,ddH2O 21 μL。PCR反应条件:95 ℃ 4 min;95 ℃ 1 min,55 ℃ 1 min,72 ℃ 2 min,30个循环;72 ℃ 10 min。PCR产物经过琼脂糖凝胶电泳检测后,交由生工生物工程(上海)股份有限公司进行测序,并与GenBank上已发布的序列进行比对。
1.2.2 重组表达载体的构建将扩增获得的角蛋白酶基因产物用DNA纯化试剂盒纯化后,以Nco Ⅰ和BamH Ⅰ进行双酶切处理,并连接到经相同双酶切的pET-22b(+)载体上。将连接产物转化至E. coli Top10感受态细胞,挑取转化子,提取质粒用BamH Ⅰ和Nco Ⅰ进行双酶切验证,将验证正确的质粒交由生工生物工程(上海)股份有限公司进行测序。
1.2.3 重组蛋白的诱导表达将测序正确的重组质粒pET22b-kp2转入E. coli RosettagamiB (DE3)感受态细胞,获得重组菌株。将重组菌株及对照菌株在LB平板上划线分离单菌落,并接种于20 mL含有100 mg/mL氨苄的LB液体培养基中,37 ℃、200 r/min培养过夜制备种子液,将种子液以5% (体积分数)的接种量转接到50 mL含有相同抗生素的LB液体培养基中,37 ℃、200 r/min培养3−4 h,当OD600约为0.8时加入终浓度为0.5 mmol/L的IPTG,然后调整培养温度至30 ℃,诱导4 h,4 ℃、8 000 r/min离心10 min后取菌体进行SDS-PAGE电泳检测。
1.2.4 诱导表达条件的优化以1.2.3所述为标准的诱导流程,采取单一变量控制法对IPTG浓度、OD600、诱导温度、诱导时间进行逐一优化,以提高表达量。其中,IPTG浓度分别为0.1、0.3、0.5、0.7、1.0、1.5、2.0 mmol/L;OD600分别为0.2、0.4、0.6、0.8、1.0、1.2;诱导温度分别为20、25、30、37 ℃;诱导时间分别为2、4、6、8、10、12 h。
1.2.5 重组蛋白的纯化在最佳诱导表达条件下,4 ℃、8 000 r/min离心10 min收集菌体,将菌体用预冷的细胞破壁缓冲液重悬,在冰浴条件下超声破碎(工作时间3 s,间歇时间5 s,功率200 W),超声约20 min至菌液清亮,再次离心并收集上清液。利用镍柱分离纯化上清液中的重组角蛋白酶,将纯化后的重组角蛋白酶进行SDS-PAGE电泳检测。使用改良型Bradford法测定蛋白的浓度[15]。
1.2.6 角蛋白酶活力测定采用福林酚法对角蛋白酶活力进行测定,具体方法参照文献[14]。
角蛋白酶活力单位定义:在50 ℃,每分钟生成1 μg酪氨酸所需的酶量定义为1个酶活单位(U)。
1.2.7 重组角蛋白酶的酶学性质(1) 最适温度和温度稳定性
在pH 7.5的条件下,分别于30、40、50、60、70、80和90 ℃测定酶活力。并将酶液放置在上述温度条件下保温1 h,测定剩余酶活,以探究其温度稳定性。以测得的最高酶活为100%,计算不同温度条件下的相对酶活力。
(2) 最适pH和pH稳定性
在最适温度条件下,分别于pH值为6.5、7.0、7.5、8.0、8.5的Tris-HCl缓冲液和pH值为8.5、9.0、9.5的Gly-NaOH缓冲液测定酶活力。将酶液保存在上述不同pH的缓冲液中1 h,测定其剩余酶活。将测得的最高酶活定义为100%,计算不同pH条件下的相对酶活力。
(3) 金属离子对酶活力的影响
在最适条件下,向反应体系中添加不同的金属离子(MnCl2、MgCl2、CaCl2、KCl、BaCl2、SiCl2、CoCl2、AlCl3、CuCl2),使各金属离子终浓度分别为0.002 5、0.025和0.25 mol/L,然后测定酶活力。以未添加金属离子测定的酶活为100%,计算不同金属离子条件下的相对酶活力。
(4) 化学试剂对酶活力的影响
在最适条件下,在反应体系中加入终浓度分别为2.5、5.0 mmol/L的EDTA、PMSF、SDS及终浓度为2.5%、5.0% (体积分数)的异丙醇、巯基乙醇、DTT、DMSO,测定酶活力。以不添加化学试剂测定的酶活为100%,计算不同化学试剂条件下的相对酶活力。
(5) 底物特异性
在最适条件下,配制2% (质量体积分数)的不同底物溶液(头发、羽毛、羽毛粉、角蛋白、酪蛋白和牛血清白蛋白)测定酶活力。以最高酶活为100%,计算不同底物下的相对酶活力。
(6) NaCl浓度对酶活力的影响
在最适条件下,向反应体系中分别加入终浓度为2.5%、5.0%、7.5%、10.0%和12.5% (质量体积分数)的NaCl,于4 ℃保存1 h,测定其剩余酶活力。将不添加NaCl测得的酶活定义为100%,计算不同NaCl浓度下的相对酶活力。
(7) 酶动力学常数
在最适条件下,以酪蛋白为底物进行酶动力学检测,底物浓度分别配制成0.5%、1.0%、1.5%、2.0%、2.5%、3.0%、3.5%和4.0% (质量体积分数),测定酶活力。以不同浓度酪蛋白的倒数为横坐标,以不同浓度酪蛋白条件下重组角蛋白酶反应速率的倒数为纵坐标,基于米氏方程进行双倒数作图,计算出酶的Vmax和Km值。
1.3 统计分析采用Excel 2016和SPSS 21对实验数据进行统计分析,以GraphPad Prism 8制图。
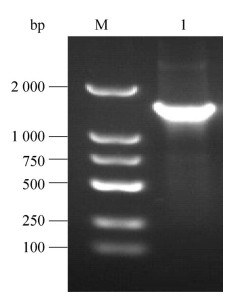
2 结果与分析 2.1 角蛋白酶基因kp2的克隆如图 1所示,以铜绿假单胞菌Gxun-7基因组DNA为模板,以kp2-F、kp2-R为引物,扩增出一条1 428 bp的DNA片段,与预期大小一致。将kp2序列提交到NCBI,获得的GenBank登录号为OM992359。序列比对分析发现,kp2与NCBI公布的铜绿假单胞菌KS-1来源的角蛋白酶基因KP2[16]的相似性为99.93%,属于M4蛋白家族,编码475个氨基酸。利用SMART (http://smart.embl-heidelberg.de/)分析蛋白质的结构域,结果表明该角蛋白酶由FTP、PepSY、Peptidase_M4和Peptidase_M4_C这4个保守的蛋白结构域组成,无信号肽序列。前两者为角蛋白酶的前导肽区域,包含169个氨基酸,后两者为成熟肽区域,包含306个氨基酸(图 2)。
|
| 图 1 角蛋白酶基因的扩增 Figure 1 Keratinase gene amplification. M: DL2000 DNA Marker;1:kp2 基因 PCR 产物 M: DL2000 DNA Marker; 1: PCR product of kp2 gene. |
|
|
|
| 图 2 角蛋白酶的结构域分析 Figure 2 Domain analysis of keratinase. |
|
|
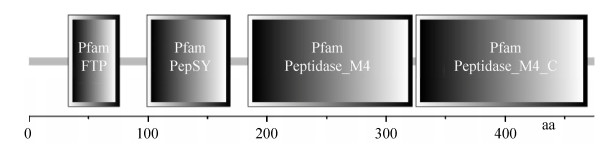
将kp2基因连接到表达载体pET-22b(+)上获得重组质粒pET22b-kp2,并用Nco Ⅰ和BamH Ⅰ进行双酶切验证,结果如图 3所示。重组质粒经双酶切后得到2个条带,一条在1 500 bp附近,与kp2基因大小一致;另一条大于5 000 bp,与线性的pET22b(+)质粒大小一致,同时测序结果也正确,由此表明重组质粒构建成功。
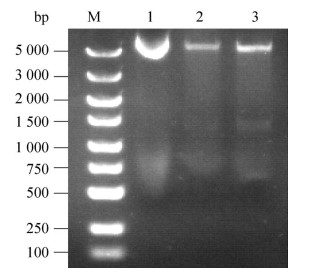
|
| 图 3 重组质粒的双酶切鉴定 Figure 3 Double digestion identification of recombinant plasmid. M: DL5000 DNA Marker;1:pET22b-kp2 重组质粒;2–3:重组 质粒 Nco I/BamH I 双酶切 M: DL5000 DNA Marker; 1: pET22b-kp2 recombinant plasmid; 2–3: Double digestion by Nco I/BamH I of recombinant plasmid. |
|
|
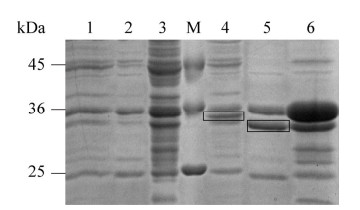
将构建成功的重组质粒pET22b-kp2转化到E. coli RosettagamiB (DE3)中,获得重组菌株。经IPTG诱导后,重组菌具有角蛋白酶活力(27.92 U/mL),而对照菌未检测到酶活力。同时,进行SDS-PAGE电泳分析,结果见图 4,相较于诱导后的对照菌,诱导后的重组菌在33 kDa附近出现了非常明显的蛋白条带,表明重组角蛋白酶成功实现了在大肠杆菌中的异源表达。
|
| 图 4 重组角蛋白酶的诱导表达 Figure 4 Induced expression of recombinant keratinase. M:蛋白Marker;1−3:E. coli RosettagamiB (DE3)/pET22b菌液、破胞后上清、破胞后沉淀;4−6:E. coli RosettagamiB (DE3)/pET22b-kp2菌液、破胞后上清、破胞后沉淀 M: Protein Marker; 1−3: E. coli RosettagamiB (DE3)/pET22b bacterial liquid, broken supernatant, broken precipitation; 4−6: E. coli RosettagamiB (DE3)/pET22b-kp2 bacterial liquid, broken supernatant, broken precipitation. |
|
|
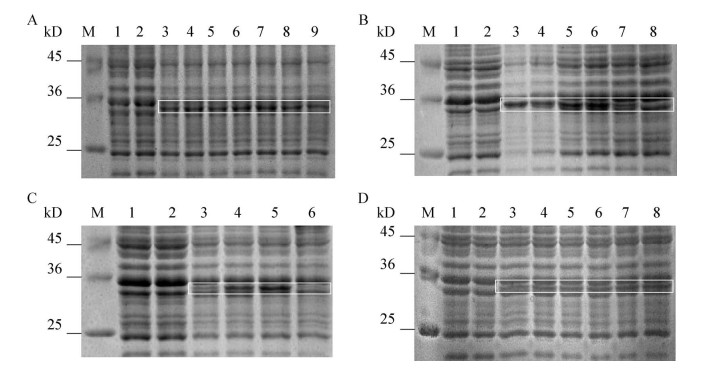
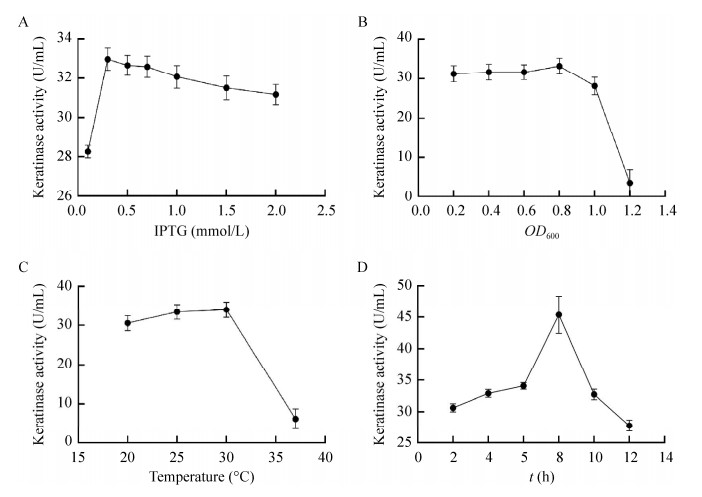
为提高重组角蛋白酶在大肠杆菌中的表达量及酶活力,对IPTG浓度、OD600、诱导温度和诱导时间进行优化。结果发现,这4个因素对角蛋白酶表达量及酶活力的影响较大,而且角蛋白酶的表达量与酶活力呈正相关(图 5、图 6)。随着IPTG浓度的增加,重组角蛋白酶活力不断升高,当IPTG浓度为0.3 mmol/L时酶活力最高,为32.99 U/mL;当IPTG浓度超过0.3 mmol/L时酶活力基本稳定,维持在31.00 U/mL (图 6A)。OD600在0.2−0.8范围内,重组角蛋白酶活力变化不明显,但当OD600为0.8时重组角蛋白酶活力为33.26 U/mL,当OD600增加至1.2时重组角蛋白酶活力降低为3.52 U/mL (图 6B)。随着诱导温度的增加,重组角蛋白酶活力呈现先升高后降低的趋势,诱导温度为30 ℃时最适合菌株的生长,此时酶活力为34.09 U/mL (图 6C)。在30 ℃诱导8 h时酶活力最高,为45.38 U/mL,诱导时间的继续增加会影响蛋白的表达,导致酶活力降低(图 6D)。因此,重组角蛋白酶表达的最佳诱导条件为:IPTG浓度为0.3 mmol/L,OD600为0.8,诱导温度为30 ℃,诱导时间为8 h,酶活力达45.38 U/mL。
|
| 图 5 不同诱导表达条件对重组角蛋白酶表达量的影响 Figure 5 Effects of different induced expression conditions on the expression yield of recombinant keratinase. M:蛋白Marker;1:E. coli RosettagamiB (DE3)/pET22b未诱导;2:E. coli RosettagamiB (DE3)/pET22b-kp2未诱导。A:IPTG浓度对蛋白表达的影响;3−9:E. coli RosettagamiB (DE3)/pET22b-kp2诱导(IPTG浓度为0.1、0.3、0.5、0.7、1.0、1.5和2.0 mmol/L)。B:OD600对蛋白表达的影响;3−8:E. coli RosettagamiB (DE3)/pET22b-kp2诱导(OD600为0.2、0.4、0.6、0.8、1.0和1.2)。C:诱导温度对蛋白表达的影响;3−6:E. coli RosettagamiB (DE3)/pET22b-kp2诱导(诱导温度为20、25、30和37 °C)。D:诱导时间对蛋白表达的影响;3−8:E. coli RosettagamiB (DE3)/pET22b-kp2诱导(诱导时间为2、4、6、8、10和12 h) M: Protein Marker; 1: Uninduced E. coli RosettagamiB (DE3)/pET22b; 2: Uninduced E. coli RosettagamiB (DE3)/pET22b-kp2. A: Effect of IPTG concentration on protein expression; 3−9: Induced E. coli RosettagamiB (DE3)/pET22b-kp2 (IPTG concentrations of 0.1, 0.3, 0.5, 0.7, 1.0, 1.5, 2.0 mmol/L). B: Effect of OD600 on protein expression; 3−8: Induced E. coli RosettagamiB (DE3)/pET22b-kp2 (OD600 of 0.2, 0.4, 0.6, 0.8, 1.0, 1.2). C: Effect of induction temperature on protein expression; 3−6: Induced E. coli RosettagamiB (DE3)/pET22b-kp2 (induction temperature of 20, 25, 30, 37 °C). D: Effect of induction time on protein expression; 3−8: Induced E. coli RosettagamiB (DE3)/pET22b-kp2 (induction time of 2, 4, 6, 8, 10, 12 h). |
|
|
|
| 图 6 不同诱导表达条件对重组角蛋白酶活力的影响 Figure 6 Effects of different induced expression conditions on the activity of recombinant keratinase. A:IPTG浓度对酶活力的影响;B:OD600对酶活力的影响;C:诱导温度对酶活力的影响;D:诱导时间对酶活力的影响 A: Effect of IPTG concentration on enzyme activity; B: Effect of OD600 on enzyme activity; C: Effect of induction temperature on enzyme activity; D: Effect of induction time on enzyme activity. |
|
|
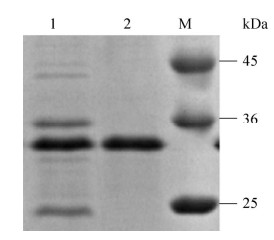
在上述优化得到的最佳条件下诱导重组角蛋白酶的表达,并利用镍柱进行分离纯化。纯化结果见表 1,纯化后重组角蛋白酶的比酶活为427.48 U/mg,回收率为35.14%。重组角蛋白酶的SDS-PAGE检测结果如图 7所示,经镍柱纯化后得到一条单一且清晰的条带,蛋白分子量约为33 kDa。
| 纯化步骤 Purification step |
总酶活 Total activity (U) |
总蛋白 Total protein (mg) |
比酶活 Specific activity (U/mg) |
回收率 Recovery rate (%) |
纯化倍数 Purification fold |
| 破胞上清液 Broken supernatant |
34 127.08 | 636.30 | 53.63 | 100.00 | 1.00 |
| 镍柱 Nickel column |
11 990.75 | 18.05 | 427.48 | 35.14 | 7.97 |
|
| 图 7 重组角蛋白酶的纯化 Figure 7 Purification of recombinant keratinase. M:蛋白Marker;1:重组角蛋白酶粗酶液;2:纯化的重组角蛋白酶 M: Protein marker; 1: Recombinant keratinase crude enzyme; 2: Purified recombinant keratinase. |
|
|
如图 8A所示,重组角蛋白酶的最适反应温度为40 ℃,在30−70 ℃范围内均能保持较高酶活力(> 85%),说明重组角蛋白酶的温度反应范围较宽。将重组角蛋白酶保温1 h后测定其剩余酶活,由图 8B可知,重组角蛋白酶在30−60 ℃具有较好的温度稳定性,相对酶活力超过70%,当温度高于60 ℃时酶活力迅速降低,在90 ℃热处理1 h后剩余酶活仅为2.17%。
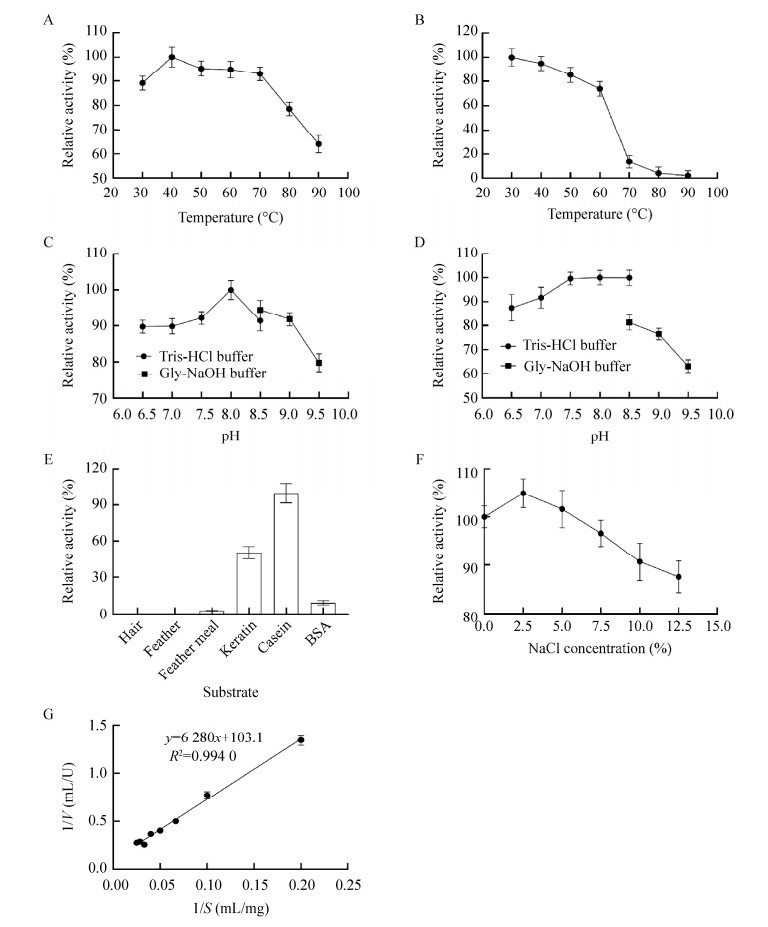
|
| 图 8 重组角蛋白酶的酶学性质 Figure 8 Enzymatic properties of recombinant keratinase. A:最适温度;B:温度稳定性;C:最适pH;D:pH稳定性;E:底物特异性;F:NaCl浓度对酶活力的影响;G:酶动力学常数 A: Optimal temperature; B: Temperature stability; C: Optimal pH; D: pH stability; E: Substrate specificity; F: The effect of NaCl concentration on enzyme activity; G: Enzyme kinetic constant. |
|
|
如图 8C所示,重组角蛋白酶的最适反应pH值为8.0。在pH值为6.5时,相对酶活力为87.95%;pH值为9.5时酶活力降低,但相对酶活力仍超过80%,说明重组角蛋白酶的pH反应范围较宽。pH稳定性实验结果(图 8D)表明,重组角蛋白酶在pH 6.5−8.0处理1 h后剩余酶活力高于85%;在pH超过8.5时酶活损失严重,但相对酶活仍达到60%以上,说明该酶能耐受偏碱性环境。当pH为8.5时,该酶在Tris-HCl缓冲液的稳定性高于Gly-NaOH缓冲液。
2.6.3 底物特异性以不同的底物测定重组角蛋白酶的活性,结果见图 8E。重组角蛋白酶对酪蛋白具有较强的水解能力。对角蛋白的水解能力次之,相对酶活为50.43%,超过酪蛋白酶活力的1/2,符合角蛋白酶的定义[17]。此外,该酶还能水解牛血清蛋白和羽毛粉,表现出的相对酶活性分别为9.23%和2.8%,而以头发和羽毛为底物时未检测到该酶的活性。
2.6.4 NaCl浓度对酶活力的影响NaCl浓度对酶活力的影响如图 8F所示,当NaCl浓度为2.5%时,重组角蛋白酶的活性最高,其相对酶活力达到104.97%;当NaCl浓度超过2.5%时酶活力逐渐降低,在12.5% NaCl的条件下该酶的活性最低,但相对酶活力仍超过85%。
2.6.5 酶动力学常数以不同浓度的酪蛋白作为底物探究重组角蛋白酶的酶动力学常数,结果如图 8G所示,采用Lineweaver-Burk双倒数作图法,得到拟合曲线的线性方程为y=6 280x+103.1 (R2=0.994 0),计算得出米氏常数Km为60.92 mg/mL、Vmax为9.70 U/mL。
2.6.6 金属离子对酶活力的影响不同金属离子对重组角蛋白酶活力的影响结果如表 2所示,当反应体系中Mn2+、Ca2+和Si2+浓度达到0.002 5 mol/L和0.025 mol/L时,对重组角蛋白酶活力有一定的促进作用,其中0.025 mol/L Ca2+促进效果最明显,较空白对照提高了1.58倍;而当Mn2+、Ca2+和Si2+的浓度增至0.25 mol/L时酶活力降低,分别为70.84%、56.24%和77.11%。Mg2+和K+对重组角蛋白酶均存在促进作用,但K+的促进作用略强。Co2+和Cu2+对重组角蛋白酶均存在抑制作用且浓度越高,抑制作用越明显。Al3+在浓度为0.025 mol/L时对重组角蛋白酶的影响很小,超过该浓度则具有抑制作用。
| 金属离子 Metal ions |
浓度 Concentration (mol/L) | ||
| 0.002 5 | 0.025 | 0.25 | |
| Control | 100.00±2.77 | 100.00±2.77 | 100.00±2.77 |
| Mn2+ | 112.32±3.14 | 122.74±3.51 | 70.84±2.32 |
| Mg2+ | 125.48±2.96 | 122.58±2.75 | 101.68±3.06 |
| Ca2+ | 116.11±3.24 | 157.53±6.85 | 56.24±1.96 |
| K+ | 141.06±2.83 | 124.48±2.10 | 122.99±5.68 |
| Si2+ | 144.27±2.09 | 104.37±5.02 | 77.11±2.71 |
| Co2+ | 96.00±4.42 | 61.05±3.17 | 30.23±2.46 |
| Al3+ | 102.95±3.74 | 4.73±1.34 | 5.02±1.71 |
| Cu2+ | 91.26±3.40 | 0.33±0.51 | 0.00±0.30 |
如表 3所示,重组角蛋白酶在不同化学试剂中酶活力差异明显。其中,异丙醇和DMSO对酶活力影响较小,而SDS明显降低酶活力,5.0 mmol/L的SDS使得酶活力几乎完全丧失。PMSF为丝氨酸蛋白酶的抑制剂,在2.5 mmol/L时重组角蛋白酶只能保留15.96%的相对酶活。当PMSF浓度为5.0 mmol/L时剩余酶活为7.03%。EDTA是金属蛋白酶的抑制剂,在2.5 mmol/L和5.0 mmol/L的EDTA作用下,该酶的相对活性分别为5.97%和8.55%。还原剂巯基乙醇和DTT能够提高重组角蛋白酶活力,而且二者均在浓度为5%时表现出的活性最高,分别为511.05%和545.59%。
| 化学试剂 Chemical reagents |
浓度 Concentration (mmol/L) | 化学试剂 Chemical reagents |
浓度 Concentration | ||
| 2.5 | 5 | 2.5% | 5% | ||
| Control | 100.00±2.77 | 100.00±2.77 | Control | 100.00±2.77 | 100.00±2.77 |
| EDTA | 5.97±1.24 | 8.55±0.29 | 异丙醇 Isopropyl alcohol | 111.30±3.11 | 118.60±2.05 |
| SDS | 53.07±2.72 | 5.59±0.33 | DMSO | 110.51±2.40 | 117.02±0.31 |
| PMSF | 15.96±1.75 | 7.03±2.07 | 巯基乙醇 Mercaptoethanol | 503.65±5.34 | 511.05±10.25 |
| DTT | 521.91±4.52 | 545.59±12.32 | |||
角蛋白酶是一类可以降解羽毛等不溶性底物和多种蛋白的蛋白水解酶。已报道的角蛋白酶主要来源于细菌、真菌和放线菌,大部分来自陆地环境,而源自海洋微生物角蛋白酶的报道相对较少[18-20]。随着角蛋白酶多种应用前景的报道,角蛋白酶越来越受到研究者的关注,已经有更多产角蛋白酶的海洋微生物被筛选出来[21]。近年来,国内外一些学者已经克隆了多个角蛋白酶基因,并成功实现了部分基因的异源表达[22-23]。本研究以pET-22b(+)为表达质粒,以E. coli RosettagamiB (DE3)为宿主菌,对海洋来源铜绿假单胞菌Gxun-7的角蛋白酶基因kp2进行异源表达及纯化。最终获得的重组角蛋白酶分子量约为33 kDa,与地衣芽孢杆菌PWD-1 (33 kDa)、假单胞菌MS21 (30 kDa)和枯草芽孢杆菌KD-N2 (30.5 kDa)来源的角蛋白酶分子量相近[24-26]。大肠杆菌遗传背景清晰、表达水平高、操作简单、营养要求低,是重组表达蛋白的首选菌株。pET-22b(+)质粒自带的pel B信号肽也是大肠杆菌高效表达的因素之一。角蛋白酶与其他蛋白酶一样,在达到有催化活性的蛋白之前,首先在前导肽的作用下正确折叠,最后通过自动催化去除前导肽以产生活性成熟酶[27]。本研究结果显示重组角蛋白酶的分子量约为33 kDa,小于理论值,可能由于该酶的结构域中存在前导肽的酶切位点,在蛋白表达过程中前导肽被剪切,导致蛋白分子量变小[21]。之前的研究也证实了角蛋白酶中存在前导肽结构,并且前导肽在蛋白表达过程中会自身剪切掉[28]。Rajput等[29]对前导肽进行改造后发现,地衣芽孢杆菌来源的角蛋白酶热稳定性和底物特异性得到了显著提升。我们也尝试切掉前导肽或缩短前导肽后进行表达,结果发现获得的重组蛋白彻底失去酶活性,证明前导肽在蛋白质翻译后的折叠中起到无可替代的作用。前导肽在角蛋白折叠中所起的作用及机制还需进一步深入研究。
优化诱导表达条件对提高重组蛋白表达量尤为重要。本研究选取IPTG浓度、OD600、诱导温度和诱导时间4个因素进行优化。当IPTG浓度为0.3 mmol/L时重组角蛋白酶活力达到最高,为32.99 U/mL,继续增大IPTG浓度,酶活力不再继续增加,表明高浓度的IPTG对细胞有毒害作用,抑制蛋白的表达[30]。菌浓度OD600对蛋白表达也具有较大影响。当OD600小于0.8时诱导,菌体浓度低,不利于生长,导致蛋白表达量少;当OD600大于0.8时,培养基中的营养物质不能满足大肠杆菌的正常代谢,也导致蛋白的表达量少。因此,重组角蛋白酶表达的最佳OD600为0.8。Fang等[31]利用大肠杆菌表达核酸酶Y. NSN时发现,当OD600为0.8时核酸酶的表达量最高,与本研究结果一致。最佳诱导温度实验结果发现,重组角蛋白酶在30 ℃诱导时酶活力最高,为34.09 U/mL,说明30 ℃条件下大肠杆菌处于最佳生长状态,合成蛋白的速度快,有利于目的蛋白的正确折叠。在37 ℃诱导时酶活力及表达量明显降低,可能原因是高温引起了包涵体的产生,造成可溶性蛋白表达量下降[32]。此外,本研究还发现重组角蛋白酶的最佳诱导时间为8 h,继续延长诱导时间,蛋白质会发生降解,导致酶活力降低。因此确定了重组角蛋白酶表达的最佳诱导条件:IPTG浓度为0.3 mmol/L,OD600为0.8,诱导温度为30 ℃,诱导时间为8 h,重组菌角蛋白酶活力为45.38 U/mL,高于大肠杆菌异源表达的枯草芽孢杆菌B-3 (14.8 U/mL)[8]和蜡样芽孢杆菌(38.2 U/mL)[33]来源的角蛋白酶活力。
重组角蛋白酶的最适反应温度为40 ℃,与嗜麦芽寡养单胞菌DHHJ来源的角蛋白酶一致[34],并且在50−70 ℃时活性都比较高,温度范围较宽。同时该酶在30−50 ℃条件下稳定性较好,而且在60 ℃处理1 h后残余活性仍然超过70%,高于枯草芽孢杆菌KS12来源的角蛋白酶的酶活力(61.4%)[35]。重组角蛋白酶的最适pH值为8.0,与多数角蛋白酶相近,而拟诺卡氏菌TOA-1来源的角蛋白酶在强酸性条件下具有最高的催化活性[36]。该酶具有较好的pH稳定性,在pH 6.5−8.0范围内处理1 h后残余酶活在85%以上,在pH 9.5时残余酶活仍高于60%,优于嗜热古细菌的角蛋白酶,有利于将其开发成商品用酶[37]。金属离子和化学试剂对重组角蛋白酶的影响较大,0.025 mol/L的Mn2+、Mg2+、Ca2+和K+分别将酶活力提高22.74%、22.58%、57.53%和24.48%,这4个金属离子可能是该酶的辅助因子,能够稳定酶的立体结构使酶活力增加。Co2+和Cu2+均对酶活力有抑制作用,可能是金属离子与酶发生反应,破坏了酶的空间结构,这与铜绿假单胞菌C11来源的角蛋白酶相似[38]。EDTA和PMSF抑制了该酶90%的活性,表明该酶属于丝氨酸金属蛋白酶[39]。重组角蛋白酶在表面活性剂SDS存在时酶活力降低,此结果与链霉菌来源的角蛋白酶一致,推测表面活性剂能够与酶形成复合物,破坏酶的盐键、氢键和疏水键[40]。5.0%的巯基乙醇和DTT能将酶活力分别提高至511.05%和545.59%,说明催化过程中可能有巯基基团的参与。该酶具有较好的NaCl耐受性,经12.5%的NaCl处理后残余酶活力仍超过85%,与地衣芽孢杆菌SWJS333来源的角蛋白酶相似,这可能是由于该酶来源于海洋环境,能够在高盐浓度存在下保持其功能构象[41]。在最适底物酪蛋白的作用下,重组角蛋白酶的Km值为60.92 mg/mL,而热带芽孢杆菌[42]和短短芽孢杆菌[43]来源的角蛋白酶水解酪蛋白的Km值分别为15.24和4.64 mg/mL,说明不同来源的角蛋白酶对酪蛋白的降解能力不同,以酪蛋白为底物时测得的Km值也不同。
本研究实现了P. aeruginosa Gxun-7角蛋白酶基因kp2在大肠杆菌中的异源表达。重组角蛋白酶分子量约为33 kDa,最适温度和pH值分别为40 ℃和8.0,在温度30−60 ℃、pH 6.5−8.0范围内保持相对稳定。该酶能被EDTA和PMSF抑制,属于丝氨酸金属蛋白酶,耐盐性较好,对酪蛋白和角蛋白具有较好的降解效果。因此,重组角蛋白酶良好的酶学特性使其在羽毛角蛋白降解工业中具有潜在的应用价值。
| [1] |
Holkar CR, Jain SS, Jadhav AJ, Pinjari DV. Valorization of keratin based waste[J]. Process Safety and Environmental Protection, 2018, 115: 85-98. DOI:10.1016/j.psep.2017.08.045 |
| [2] |
Huang Y, Liu X, Ran Y, Cao Q, Zhang A, Li D. Production of feather oligopeptides by a newly isolated bacterium Pseudomonas otitis H11[J]. Poultry Science, 2019. |
| [3] |
Verma A, Singh H, Anwar S, Chattopadhyay A, Tiwari KK, Kaur S, Dhilon GS. Microbial keratinases: industrial enzymes with waste management potential[J]. Critical Reviews in Biotechnology, 2017, 37(4): 476-491. DOI:10.1080/07388551.2016.1185388 |
| [4] |
Mohorčič M, Torkar A, Friedrich J, Kristl J, Murdan S. An investigation into keratinolytic enzymes to enhance ungual drug delivery[J]. International Journal of Pharmaceutics, 2007, 332(1/2): 196-201. |
| [5] |
Reddy MR, Reddy KS, Chouhan YR, Bee H, Reddy G. Effective feather degradation and keratinase production by Bacillus pumilus GRK for its application as bio-detergent additive[J]. Bioresource Technology, 2017, 243: 254-263. DOI:10.1016/j.biortech.2017.06.067 |
| [6] |
Akhter M, Wal Marzan L, Akter Y, Shimizu K. Microbial bioremediation of feather waste for keratinase production: an outstanding solution for leather dehairing in tanneries[J]. Microbiology Insights, 2020, 13: 1178636120913280. |
| [7] |
Schommer VA, Wenzel BM, Daroit DJ. Anaerobic co-digestion of swine manure and chicken feathers: effects of manure maturation and microbial pretreatment of feathers on methane production[J]. Renewable Energy, 2020, 152: 1284-1291. DOI:10.1016/j.renene.2020.01.154 |
| [8] |
Hou SQ, Wang LH, Lai X, Chen H, Wu Q, Han XY. Isolation, identification of B-3 Bacillus subtilis and cloning, expression of kerC[J]. China Environmental Science, 2012, 32(10): 1845-1852. (in Chinese) 侯晟琦, 王丽华, 赖欣, 陈惠, 吴琦, 韩学易. 角蛋白酶产生菌的分离鉴定及其kerC的克隆表达[J]. 中国环境科学, 2012, 32(10): 1845-1852. DOI:10.3969/j.issn.1000-6923.2012.10.018 |
| [9] |
Fu Y, Zhang TY, Sun YX, Li SY. Enhancing thermostability of keratinase by directed evolution technology[J]. Chinese Journal of Animal Nutrition, 2021, 33(10): 5887-5894. (in Chinese) 傅岩, 张铁鹰, 孙英霞, 李松育. 通过定向进化技术提高角蛋白酶的热稳定性研究[J]. 动物营养学报, 2021, 33(10): 5887-5894. DOI:10.3969/j.issn.1006-267x.2021.10.047 |
| [10] |
Korkmaz H, Hur H, Dincer S. Characterization of alkaline keratinase of Bacillus licheniformis strain HK-1 from poultry waste[J]. Annals of Microbiology, 2004, 54(2): 201-211. |
| [11] |
Arokiyaraj S, Varghese R, Ahmed BA, Duraipandiyan V, Al-Dhabi NA. Optimizing the fermentation conditions and enhanced production of keratinase from Bacillus cereus isolated from halophilic environment[J]. Saudi Journal of Biological Sciences, 2019, 26(2): 378-381. DOI:10.1016/j.sjbs.2018.10.011 |
| [12] |
Bohacz J. Biodegradation of feather waste keratin by a keratinolytic soil fungus of the genus Chrysosporium and statistical optimization of feather mass loss[J]. World Journal of Microbiology & Biotechnology, 2017, 33(1): 13. |
| [13] |
Gousterova A, Braikova D, Goshev I, Christov P, Tishinov K, Vasileva-Tonkova E, Haertlé T, Nedkov P. Degradation of keratin and collagen containing wastes by newly isolated thermoactinomycetes or by alkaline hydrolysis[J]. Letters in Applied Microbiology, 2005, 40(5): 335-340. DOI:10.1111/j.1472-765X.2005.01692.x |
| [14] |
Yang MY, Pang KR, Pan JX, Zhang HY, Wang YB, Jiang MG, Shen NK. Isolation, identification, optimization of enzyme-producing conditions and enzymatic activity of a feather-degradable Pseudomonas aeruginosa strain[J]. Acta Microbiologica Sinica, 2022, 62(3): 968-981. (in Chinese) 杨梦莹, 庞坤容, 潘江欣, 张红岩, 王一兵, 姜明国, 申乃坤. 一株可高效降解羽毛的铜绿假单胞菌的分离、鉴定、产酶条件优化及其酶活研究[J]. 微生物学报, 2022, 62(3): 968-981. DOI:10.13343/j.cnki.wsxb.20210339 |
| [15] |
Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [16] |
Sharma R, Gupta R. Extracellular expression of keratinase Ker P from Pseudomonas aeruginosa in E. coli[J]. Biotechnology Letters, 2010, 32(12): 1863-1868. DOI:10.1007/s10529-010-0361-2 |
| [17] |
Brandelli A, Daroit DJ, Riffel A. Biochemical features of microbial keratinases and their production and applications[J]. Applied Microbiology and Biotechnology, 2010, 85(6): 1735-1750. DOI:10.1007/s00253-009-2398-5 |
| [18] |
Abdel-Fattah AM, El-Gamal MS, Ismail SA, Emran MA, Hashem AM. Biodegradation of feather waste by keratinase produced from newly isolated Bacillus licheniformis ALW1[J]. Journal of Genetic Engineering and Biotechnology, 2018, 16(2): 311-318. DOI:10.1016/j.jgeb.2018.05.005 |
| [19] |
Shapourzadeh A, Rahimi-Verki N, Atyabi SM, Shams-Ghahfarokhi M, Jahanshiri Z, Irani S, Razzaghi-Abyaneh M. Inhibitory effects of cold atmospheric plasma on the growth, ergosterol biosynthesis, and keratinase activity in Trichophyton rubrum[J]. Archives of Biochemistry and Biophysics, 2016, 608: 27-33. DOI:10.1016/j.abb.2016.07.012 |
| [20] |
Nandini A, Madhusudhan DN, Dayanand A. Enhanced production, purification and characterization of alkaline keratinase from Streptomyces minutiscleroticus DNA38[J]. International Letters of Natural Sciences, 2015, 43: 27-37. DOI:10.56431/p-zpi3k0 |
| [21] |
Manivasagan P, Sivakumar K, Gnanam S, Venkatesan J, Kim SK. Production, biochemical characterization and detergents application of keratinase from the marine actinobacterium Actinoalloteichus sp. MA-32[J]. Journal of Surfactants and Detergents, 2014, 17(4): 669-682. DOI:10.1007/s11743-013-1519-4 |
| [22] |
Mwanza EP, Van Der Westhuizen WA, Boucher CE, Charimba G, Hugo C. Heterologous expression and characterisation of a keratinase produced by Chryseobacterium carnipullorum[J]. Protein Expression and Purification, 2021, 186: 105926. DOI:10.1016/j.pep.2021.105926 |
| [23] |
Hu H, Gao J, He J, Yu B, Zheng P, Huang ZQ, Mao XB, Yu J, Han GQ, Chen DW. Codon optimization significantly improves the expression level of a keratinase gene in Pichia pastoris[J]. PLoS One, 2013, 8(3): e58393. DOI:10.1371/journal.pone.0058393 |
| [24] |
Lin X, Lee CG, Casale ES, Shih JC. Purification and characterization of a keratinase from a feather-degrading Bacillus licheniformis strain[J]. Applied and Environmental Microbiology, 1992, 58(10): 3271-3275. DOI:10.1128/aem.58.10.3271-3275.1992 |
| [25] |
Tork S, Aly MM, Nawar L. Biochemical and molecular characterization of a new local keratinase producing Pseudomomanas sp., MS21[J]. Asian Journal of Biotechnology, 2009, 2(1): 1-13. DOI:10.3923/ajbkr.2010.1.13 |
| [26] |
Cai CG, Chen JS, Qi JJ, Yin Y, Zheng XD. Purification and characterization of keratinase from a new Bacillus subtilis strain[J]. Journal of Zhejiang University Science B, 2008, 9(9): 713-720. DOI:10.1631/jzus.B0820128 |
| [27] |
Liu BH. Over expression of Bacillus licheniformis keratinase, its molecular modification for enhanced thermostability and substrate specificity[D]. Wuxi: Doctoral Dissertation of Jiangnan University, 2015 (in Chinese) 刘柏宏. Bacillus licheniformis角蛋白酶的高效表达、热稳定性及底物特异性改造[D]. 无锡: 江南大学博士学位论文, 2015 |
| [28] |
Sharma R, Murty NAR, Gupta R. Molecular characterization of N-terminal pro-sequence of keratinase Ker P from Pseudomonas aeruginosa: identification of region with chaperone activity[J]. Applied Biochemistry and Biotechnology, 2011, 165(3/4): 892-901. |
| [29] |
Rajput R, Tiwary E, Sharma R, Gupta R. Swapping of pro-sequences between keratinases of Bacillus licheniformis and Bacillus pumilus: altered substrate specificity and thermostability[J]. Enzyme and Microbial Technology, 2012, 51(3): 131-138. DOI:10.1016/j.enzmictec.2012.04.010 |
| [30] |
Fang Z, Sha C, Peng Z, Zhang J, Du GC. Protein engineering to enhance keratinolytic protease activity and excretion in Escherichia coli and its scale-up fermentation for high extracellular yield[J]. Enzyme and Microbial Technology, 2019, 121: 37-44. DOI:10.1016/j.enzmictec.2018.11.003 |
| [31] |
Fang XJ, Tang ZX, Li ZH, Zhang ZL, Shi L. Production of a new non-specific nuclease from Yersinia enterocolitica subsp. palearctica: optimization of induction conditions using response surface methodology[J]. Biotechnology, Biotechnological Equipment, 2014, 28(3): 559-566. DOI:10.1080/13102818.2014.915612 |
| [32] |
Zhang ZQ, Liu XC, Niu JZ, Huang Y, Wang B, Jian JC. Prokaryotic expression and optimization of Galectin-4 gene from Nile tilapia (Oreochromis niloticus)[J]. Journal of Guangdong Ocean University, 2020, 40(5): 118-123. (in Chinese) 张志强, 刘鑫潮, 牛金中, 黄瑜, 王蓓, 简纪常. 尼罗罗非鱼Galectin-4基因的原核表达及诱导条件优化[J]. 广东海洋大学学报, 2020, 40(5): 118-123. DOI:10.3969/j.issn.1673-9159.2020.05.015 |
| [33] |
Gurunathan R, Huang B, Ponnusamy VK, Hwang JS, Dahms HU. Novel recombinant keratin degrading subtilisin like serine alkaline protease from Bacillus cereus isolated from marine hydrothermal vent crabs[J]. Scientific Reports, 2021, 11: 12007. DOI:10.1038/s41598-021-90375-4 |
| [34] |
Cao ZJ, Zhang Q, Wei DK, Chen L, Wang J, Zhang XQ, Zhou MH. Characterization of a novel Stenotrophomonas isolate with high keratinase activity and purification of the enzyme[J]. Journal of Industrial Microbiology and Biotechnology, 2009, 36(2): 181-188. DOI:10.1007/s10295-008-0469-8 |
| [35] |
Xu ZL, Yin YJ, Xia X, Liang YX, Zhao SM, Hu YL. Screening, identifing and enzymatic characterizations of a keratinase-producing strain[J]. Journal of Anhui Agricultural University, 2021, 48(4): 620-624. (in Chinese) 徐志龙, 尹雅洁, 夏险, 梁运祥, 赵述淼, 胡远亮. 角蛋白酶生产菌株的筛选鉴定及酶学性质研究[J]. 安徽农业大学学报, 2021, 48(4): 620-624. |
| [36] |
Gupta R, Ramnani P. Microbial keratinases and their prospective applications: an overview[J]. Applied Microbiology and Biotechnology, 2006, 70(1): 21-33. DOI:10.1007/s00253-005-0239-8 |
| [37] |
Parinayawanich S, Sittipol D, Ajingi YS, Rodpan S, Pattanapanyasat K, Jongruja N. Application of recombinant hyperthermostable keratinase for degradation of chicken feather waste[J]. Biocatalysis and Agricultural Biotechnology, 2021, 36: 102146. DOI:10.1016/j.bcab.2021.102146 |
| [38] |
Han MH. Isolation and characterization of a keratinolytic protease from a feather-degrading bacterium Pseudomonas aeruginosa C11[J]. African Journal of Microbiology Research, 2012, 6(9): 2211-2221. |
| [39] |
Zhang RX, Gong JS, Su C, Zhang DD, Tian H, Dou WF, Li H, Shi JS, Xu ZH. Biochemical characterization of a novel surfactant-stable serine keratinase with no collagenase activity from Brevibacillus parabrevis CGMCC 10798[J]. International Journal of Biological Macromolecules, 2016, 93: 843-851. DOI:10.1016/j.ijbiomac.2016.09.063 |
| [40] |
Tatineni R, Doddapaneni KK, Potumarthi RC, Vellanki RN, Kandathil MT, Kolli N, Mangamoori LN. Purification and characterization of an alkaline keratinase from Streptomyces sp.[J]. Bioresource Technology, 2008, 99(6): 1596-1602. DOI:10.1016/j.biortech.2007.04.019 |
| [41] |
Zhong HB, Huan HJ, Lei FF, Zhao QZ, Zhao MM, Cui C. Isolation of producing-proteases marine bacteria and characterization of the proteases in fermented liquid[J]. Food and Fermentation Industries, 2013, 39(8): 108-112. (in Chinese) 钟泓波, 郇惠杰, 雷芬芬, 赵强忠, 赵谋明, 崔春. 产蛋白酶深海细菌的筛选及其蛋白酶酶学性质[J]. 食品与发酵工业, 2013, 39(8): 108-112. |
| [42] |
Shen NK, Yang MY, Xie CJ, Pan JX, Pang KR, Zhang HY, Wang YB, Jiang MG. Isolation and identification of a feather degrading Bacillus tropicus strain Gxun-17 from marine environment and its enzyme characteristics[J]. BMC Biotechnology, 2022, 22(1): 11. |
| [43] |
Xu JH, Yin YJ, Li XY, Liu K, Liang YX, Zhao SM, Hu YL. Construction and characterization of recombinant Pichia pastoris strain with keratinase production[J]. Journal of Huazhong Agricultural University, 2019, 38(3): 71-76. (in Chinese) 许佳惠, 尹雅洁, 李欣悦, 柳凯, 梁运祥, 赵述淼, 胡远亮. 产角蛋白酶毕赤酵母工程菌的构建及酶学性质[J]. 华中农业大学学报, 2019, 38(3): 71-76. |
 2022, Vol. 49
2022, Vol. 49