扩展功能
文章信息
- 余志银, 颜一军, 刘重喜, 黄建萍, 向文胜, 黄胜雄
- YU Zhiyin, YAN Yijun, LIU Chongxi, HUANG Jianping, XIANG Wensheng, HUANG Shengxiong
- 稀有放线菌 Lechevalieria rhizosphaerae NEAU-A2的次级代谢产物研究及遗传操作系统的建立
- Secondary metabolites and genetic system of the rare actinobacteria Lechevalieria rhizosphaerae NEAU-A2
- 微生物学通报, 2021, 48(7): 2318-2328
- Microbiology China, 2021, 48(7): 2318-2328
- DOI: 10.13344/j.microbiol.china.210187
-
文章历史
- 收稿日期: 2021-02-22
- 接受日期: 2021-03-25
- 网络首发日期: 2021-05-07
2. 东北农业大学生命科学学院 黑龙江 哈尔滨 150030
2. College of Life Science, Northeast Agricultural University, Harbin, Heilongjiang 150030, China
稀有放线菌(Rare Actinobacteria)一般指除链霉菌属以外的放线菌。目前己报道的有15个目200多个属,其中研究较多的主要有小单孢菌(Micromonospora)、盐孢菌(Salinispora)、拟无枝酸菌(Amycolatopsis)、马杜拉放线菌(Actinomadura)等[1],然而列舍瓦列氏菌属(Lechevalieria)的研究报道较少。尽管从链霉菌中发现了众多结构新颖的天然产物,包括目前临床上使用的抗生素有2/3来源于链霉菌,但是近年来有越来越多结构新颖的天然产物从稀有放线菌中被发现,来源于链霉菌的新颖天然产物呈现下降趋势[2]。稀有放线菌比链霉菌生长缓慢,特殊的生长条件导致了它们独特的生理特性。因此,对稀有放线菌次级代谢产物的研究也较少,这使得稀有放线菌成为发现新型抗生素的潜在资源[3]。
据统计,从2008年到2018年,对来源于稀有放线菌的次级代谢产物的化学结构和生物活性研究涉及了20个属,共报道了341个代谢产物,占整个微生物来源的代谢产物的16%[3]。张举成等[4]总结发现,自2006年1月至2018年6月在陆地土壤、植物、海洋动物、海洋沉积物等来源的稀有放线菌中分离到515个天然产物,涉及聚酮、蒽醌、联吡啶类、多烯、大环内酯等结构类型,其中部分化合物具有显著的抗肿瘤、抗菌等活性。此外,在过去的20年中,稀有放线菌来源的抗生素占到了已报道抗生素的20%−30%[2]。因此,稀有放线菌已成为发现新颖活性化合物的重要来源。
菌株Lechevalieria rhizosphaerae NEAU-A2由本实验室2017年从河北省廊坊市采集的小麦(Triticum aestivum L.)根际土壤样品中分离获得,通过多相分类学鉴定为一株新的稀有放线菌[5]。近期该菌株的全基因组测序已完成,总共得到78个Scaffold序列,基因组大小约为10.28 Mb。使用antiSMASH[6]软件预测发现基因组中有27个与次级代谢产物生物合成相关的基因簇,其中涉及PKS和NRPS的生物合成基因簇有10个,与萜类生物合成相关的基因簇有6个,表明该菌株具有合成丰富次级代谢产物的潜力。因此,在本研究中,我们首先对菌株NEAU-A2的次级代谢产物进行分析,从中分离鉴定了2个新的吲哚二聚生物碱(1–2)和5个已知化合物(图 1),分别为N-乙酰色胺(3)、4-((2-(1H-Indol-3-Yl)Ethyl)Amino)-4-Oxobutanoic Acid (4)、Brevianamide F (5)、4S, 7R-Germacra-1(10)E, 5E-Diene-11-Ol (6)、1H-吡咯-2-羧酸(7)。同时以PKS-NRPS杂合基因1609为目的基因,采用PCR-Targeting技术[7-8]和接合转移的方法,以期建立稀有放线菌L. rhizosphaerae NEAU-A2的遗传操作体系,为后期通过基因挖掘等手段发现结构新颖的活性天然产物奠定基础。
|
| 图 1 L. rhizosphaerae NEAU-A2中分离得到的化合物1–7的化学结构图 Figure 1 Structure of compounds (1–7) isolated from L. rhizosphaerae NEAU-A2 注:*:新化合物 Note: *: New compounds |
|
|
ISP3固体培养基(g/L):燕麦20.0 g煮沸30 min,用4层纱布过滤取滤液,MgSO4·7H2O 0.2,K2HPO4·3H2O 0.5,KNO3 0.5,琼脂粉20.0,pH 7.0。
种子培养基:TSB培养基30.0 g/L;其成分(g/L):大豆蛋白胨3.0,胰蛋白胨17.0,葡萄糖2.5,NaCl 5.0,K2HPO4·3H2O 2.5,pH 7.3。
发酵培养基(g/L):棉籽饼粉20.0,酵母提取物5.0,葡萄糖10.0,玉米淀粉30.0,MgSO4·7H2O 1.0,麦芽糊精20.0,CaCO3 2.0,pH 7.2。
LB培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,NaCl 10.0,琼脂粉(固体培养基加入) 20.0,pH 7.0。
2×YT培养基(g/L):胰蛋白胨16.0,酵母提取物10.0,NaCl 5.0,pH 7.0。
1.1.2 主要试剂和仪器研究所用酶购于Takara公司或NEB公司,其他试剂为国产分析纯试剂(天津市大茂化学试剂厂、广东环凯微生物科技有限公司)。电穿孔系统、PCR仪,Bio-Rad公司;凝胶成像分析仪,郑州南北仪器设备有限公司;核磁共振仪(TMS作为内标),布鲁克(北京)科技有限公司;Agilent UPLC/Q-Tof液质联用仪,安捷伦科技有限公司;薄层色谱硅胶板和200–300目柱色谱硅胶,青岛海洋化工有限公司;Sephadex LH-20,AIRTECH生物化学试剂公司;高效液相色谱仪,日立(中国)有限公司;YMC-Triat C18型液相色谱柱(250 mm×10 mm),YMC公司。
1.1.3 菌株、质粒和引物| Strains/Plasmids | Genotype/Description | References/Source |
| L. rhizosphaerae NEAU-A2 | This study | |
| ET12567/pUZ8002 | recE, dcm–, dam–, hsdS, Cmr, Tetr, Strr, Kmr | [7-8] |
| BW25113/pIJ790 | λ-RED (gam, bet, exo), cat, araC, rep101ts | [7-8] |
| E. coli DH5α/pIJ773 | aac(3) IV, oriT | [7-8] |
| pSuperCos Ⅰ | Kanr, used for construction of gene knockout cosmoid | Stratagene |
| XL1-Blue | Construction of gene library | Agilent |
| Primers name | Primers sequence (5′→3′) | Purpose |
| 1609-F | GCGTAGACACCGATCACTTCCTGC | Screening cosmid library |
| 1609-R | CCGCGTCACGTCGATCTACCAG | |
| Δ1609-F | CTCCTGAAAACCCCGACGCACCACGGAGGACTACCAGTGATTCCGGGGATCCGTCGACC | Inframe deletion |
| Δ1609-R | GTACAGGTCCTCGATGTTCTTCGCCATCACTGCCTCTCATGTAGCTGGAGCTGCTTC | |
| 1609-check-F | TGAAAACCCCGACGCACCAC | Genotype verification |
| 1609-check-R | TGTTCTTCGCCATCACTGCC |
共选择6种培养基ISP2、ISP3、ISP4、ISP5、ISP6、ISP7[5],分别接种约108个甘油管中保存的孢子,30 ℃培养4–6 d后观察孢子产生情况。
1.2.2 菌株的培养与发酵将ISP3平板上活化好的菌株NEAU-A2接种到20只装有50 mL种子液的250 mL三角瓶中,置于30 ℃、220 r/min摇床上培养36 h。将种子液按10% (体积分数)接种量转接到60只盛有250 mL发酵培养基的1 L三角瓶中,置于30 ℃、220 r/min摇床上培养7 d。
1.2.3 次级代谢产物的提取与分离L. rhizosphaerae NEAU-A2在发酵培养基中培养7 d后共收集到15 L的发酵液,于4 000 r/min转速下离心20 min分离菌体和上清液。上清液用等体积的乙酸乙酯萃取3次,乙酸乙酯萃取相减压浓缩后得10.0 g油状粗提物。将油状粗提物经正相硅胶柱色谱分离,干法上样,石油醚-乙酸乙酯梯度(10:0、10:2、10:10、0:10,体积比)洗脱后,再用乙酸乙酯-甲醇梯度(10:1、10:5、0:10,体积比)洗脱,TLC检测后合并得到4个组分(Frs.A−D)。Frs.A段用半制备高效液相色谱制备得化合物6(36.8 min,10 mg),方法为:0–23.0 min,50%乙腈-水;23.1–38.0 min,70%乙腈-水。Frs.B段(石油醚-乙酸乙酯10:2,体积比)用半制备高效液相色谱制备得化合物7(14.0 min,8 mg),方法为:15%乙腈-水等度洗脱。Frs.D段(乙酸乙酯: 甲醇,10:1,体积比)过凝胶色谱Sephadex LH-20,甲醇洗脱,TLC、HPLC分析后,得到2个组分(Frs.D1和Frs.D2)。Frs.D2段用半制备高效液相色谱制备得到3个组分Frs.D2.1–D2.3,方法为:0–12.0 min,25%乙腈-水;12.1–41.0 min,29%乙腈-水;41.1–45.0 min,100%乙腈-水;45.1–49.0 min,25%乙腈-水,富集13.0 min的流分得Frs.D2.1,富集20.0 min的流分得Frs.D2.2,富集26.0 min的流分得Frs.D2.3。Frs.D2.1段用半制备高效液相色谱制备,方法为:35%甲醇-水等度洗脱,得化合物5(1.0 mg)。Frs.D2.2段用半制备高效液相色谱制备,方法为:22%乙腈-水等度洗脱得化合物3(2.5 mg)和4(1.0 mg)。Frs.D2.3段用半制备高效液相色谱二次纯化得到化合物1(10.0 mg)和2(5.0 mg),方法为:50%甲醇水等度洗脱。
1.2.4 抗生素抗性谱检测在固体ISP3培养基中分别添加不同浓度的卡那霉素(25、50、75、100 µg/mL)、硫酸安普霉素(25、50、75、100 μg/mL)、硫链丝菌素(25、50、75 μg/mL)、红霉素(50、75、100、200 μg/mL)、三甲氧苄氨嘧啶(25、50、75、100 μg/mL),然后将新鲜的L. rhizosphaerae NEAU-A2孢子涂布于平板上,30 ℃培养4–6 d,观察生长情况。
1.2.5 基因组文库构建及筛选根据链霉菌手册的标准操作[9],提取L. rhizosphaerae NEAU-A2的基因组,然后用Mbo I酶切处理后回收30–42 kb大小DNA片段,去磷酸化酶处理后,连接到处理好的pSuperCos I载体上。取适量的包装抽提物MaxPlax Lambda Packaging Extracts包装连接产物,转化到大肠杆菌XL1-Blue菌中,随机挑取2 000个单克隆,保存于–80 ℃。
针对PKS-NRPS杂合基因(1609)设计一对特异性引物,从L. rhizosphaerae NEAU-A2的基因文库中筛选含有1609基因序列的Cosmid。
1.2.6 利用PCR-Targeting技术构建重组质粒首先,根据基因1609的序列设计一对基因中断引物(表 2,Δ1609-F和Δ1609-R)。引物长度为59 bp,其5′端39 bp分别与目的序列同源,而剩余3′端20 bp序列与pIJ773的Apr抗性基因两侧序列相匹配。质粒pIJ773经EcoR Ⅰ和Hind Ⅲ双酶切后,回收1.4 kb片段作为模板,以Δ1609-F和Δ1609-R为引物,用DNA聚合酶进行PCR扩增得到约1.4 kb的aac(3)Ⅳ+oriT片段。与此同时,将含有PKS-NRPS生物合成基因簇的S4H1-cosmid转化大肠杆菌BW25113/pIJ790。取约100 ng的aac(3)Ⅳ+ori T片段,电击转化BW25113/pIJ790/ S4H1-cosmid感受态细胞。在LB+Apr+Kan抗性平板上筛选阳性转化子,从中提取质粒,此为重组含有Apr抗性片段的S4H1-cosmid,将其命名为S4H1-Δ1609-cosmid,转化大肠杆菌ET12567/ pUZ8002感受态细胞。
1.2.7 接合转移将ET12567/pUZ8002/S4H1-Δ1609-cosmid接种于5 mL LB培养基中(Kan、Chl、Apr),37 ℃、220 r/min摇床上培养过夜。次日按1:100转种于5 mL LB培养基中(Kan、Chl、Apr),37 ℃培养大约4 h至OD600为0.4,4 000 r/min离心5 min收集菌体,用无抗生素的液体LB培养基洗涤2次,重悬于500 μL无抗生素的液体LB培养基中待用。
收集L. rhizosphaerae NEAU-A2在固体ISP3培养基上培养大约4 d的新鲜孢子,用无菌水洗下孢子转移至50 mL的离心管中,振荡混匀后,用脱脂棉过滤在干净的离心管中,3 500 r/min离心10 min,去上清后加入500 mL的2×YT培养基轻轻混匀,50 ℃分别热激5、8、10 min,分析热激时间对接合转移的影响。冷却至室温后,30 ℃分别温浴0、0.5、1、2 h,探究孢子预萌发时间对接合转移效率的影响。
分别取500μL的大肠杆菌ET12567/pUZ8002/ S4H1-Δ1609-cosmid菌液和500 μL的孢子液混匀,均匀涂布到含有10 mmol/L MgCl2的ISP3平板上,于30 ℃培养16–18 h。用含25μL Apr (储存浓度50 mg/mL)和30 μL三甲氧苄氨嘧啶(储存浓度50 mg/mL)的1 mL无菌水均匀覆盖平板,表面吸收后于30 ℃培养3–5 d。将长出的阳性菌落在含有Apr抗性(若有细菌可加入三甲氧苄氨嘧啶)的ISP3固体平板上继续培养3–5 d。待长出单菌落后进行单双交换筛选。挑取单菌落同时接种于只含有Apr抗性和同时含有Apr和Kan抗性的平板上,30 ℃培养3–5 d。选择只能在含有Apr抗性的平板上生长而不能在同时含有Apr和Kan抗性的平板上生长的菌落(双交换突变株),划线培养获得纯种单菌落。用对应的验证引物(表 2,1609-check-F/1609-check-R)通过PCR鉴定双交换菌株基因组,验证正确后得到对应基因的敲除突变株。
2 结果与分析 2.1 化合物结构鉴定化合物1,黄色粉末,通过高分辨质谱HRESIMS (Found 414.180 2 [M+Na]+,Calcd 414.178 8) 确定其分子式为C23H25N3O3,不饱和度为13。1H-NMR谱显示(表 3)含有1个单峰甲基信号:δH 1.60 (s,3H);低场区9个芳香质子信号,展示了一个特征的吲哚信号:7.54 (1H,d,J=8.0 Hz,H-4′),7.31 (1H,d,J=8.1 Hz,H-7′),6.94 (1H,m,H-5′),7.04 (1H,ddd,J=8.0,7.2,1.0 Hz,H-6′),7.43 (1H,s,H-2′)和一个1,2位取代的苯环信号[δH 7.48 (1H,d,J=7.8 Hz,H-4),7.29 (1H,d,J=8.0 Hz,H-7),7.01 (1H,ddd,J=8.1,7.0,1.1 Hz,H-6),6.94 (1H,m,H-5)]。13C NMR谱数据显示共有23个碳,包含16个芳香碳信号,并呈现了成对信号,推测可能是吲哚二聚衍生物。仔细分析13C NMR和HSQC谱,23个碳信号分别为9个sp2杂化的次甲基碳(δC 111.8、112.2、119.0、119.6、119.7、119.8、121.8、122.3、124.4),7个sp2杂化的季碳(δC 108.8、114.6、129.0、129.4、137.3、137.7、138.2),1个羰基碳(δC 173.3),3个sp3杂化的亚甲基碳(δC 25.3、41.3、65.9),2个sp3杂化的次甲基碳(δC 37.9、76.1),1个甲基碳(δC 22.4)。以上数据与化合物3-(3-(2-Hydroxyethyl)-1H-Indol-2-Yl)-3-(1H-Indol-3- Yl) Propane-1, 2-Diol的核磁数据显示了相似性[10-11]。
| No. | 2 | 1 | |||
| δH | δC | δH | δC | ||
| 2 | 137.4 | 137.3 | |||
| 3 | 110.0 | 108.8 | |||
| 3a | 129.2 | 129.4 | |||
| 4 | 7.48 (d, J=7.8 Hz, 1H) | 118.8 | 7.48 (d, J=7.8 Hz, 1H) | 119.0 | |
| 5 | 6.96 (m, 1H) | 119.5 | 6.94 (m, 1H) | 119.7 | |
| 6 | 7.03 (m, 1H) | 121.8 | 7.01 (ddd, J=8.1, 7.0, 1.1 Hz, 1H) | 121.8 | |
| 7 | 7.32 (overlapped) | 111.8 | 7.29 (d, J=8.0 Hz, 1H) | 111.8 | |
| 7a | 137.4 | 137.7 | |||
| 8 | 3.01 (t, J=7.0 Hz, 2H) | 25.1 | 3.03 (m, 2H) | 25.3 | |
| 9 | 3.38 (m, 2H) | 41.4 | 3.38 (m, 2H) | 41.4 | |
| 10 | 173.4 | 173.3 | |||
| 11 | 1.60 (s, 3H) | 22.3 | 1.68 (s, 3H) | 22.4 | |
| 2′ | 7.23 (s, 1H) | 124.1 | 7.43 (s, 1H) | 124.4 | |
| 3′ | 116.3 | 114.6 | |||
| 3′a | 128.0 | 129.0 | |||
| 4′ | 7.61 (d, J=8.0 Hz, 1H) | 119.4 | 7.54 (d, J=8.0 Hz, 1H) | 119.6 | |
| 5′ | 7.00 (m, 1H) | 119.9 | 6.94 (m, 1H) | 119.8 | |
| 6′ | 7.07 (m, 1H) | 122.5 | 7.04 (ddd, J=8.0, 7.2, 1.0 Hz, 1H) | 122.3 | |
| 7′ | 7.32 (overlapped) | 112.3 | 7.31 (d, J=8.1 Hz, 1H) | 112.2 | |
| 7′a | 137.9 | 138.2 | |||
| 8′ | 4.75 (d, J=5.9 Hz, 1H) | 37.4 | 4.77 (d, J=6.3 Hz, 1H) | 37.9 | |
| 9′ | 4.46 (m, 1H) | 75.5 | 4.39 (td, J=6.5, 4.2 Hz, 1H) | 76.1 | |
| 10′ | 3.50 (dd, J=11.0, 6.7 Hz, 1H) | 66.1 | 3.43 (dd, J=11.1, 6.7 Hz, 1H) | 65.9 | |
| 3.54 (dd, J=11.0, 5.0 Hz, 1H) | 3.59 (dd, J=11.1, 4.2 Hz, 1H) | ||||
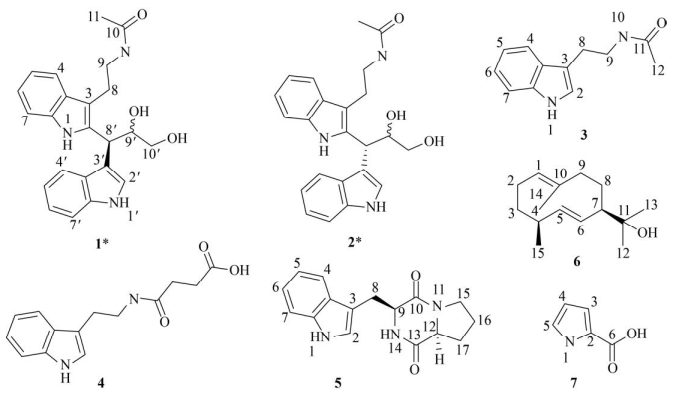
分析二维核磁数据,1H-1H COSY和HSQC谱揭示了4个耦合系统(图 2),2个1、2位取代的芳香环(C4-C5-C6-C7,C-4′-C-5′-C-6′-C-7′),1个1、2位被氧化的丙基链(C-8′-C-9′-C-10′)和一个乙基链(C-8-C-9)。根据HMBC谱中H-8与C-2/C-3/C-3a相关,H-9与C-10/C-3相关,H-11与C-10相关(图 2),可知乙基侧链连接在吲哚环的C-3位,乙酰胺基连接在C-9位。H-8′与C-3′/C-2′/C-3′a、H-2′与C-8′/C-9′存在HMBC远程相关,可以确定1、2位被氧化的丙基侧链通过C-3′位与第2个吲哚环连接。基于HMBC谱中H-8′与C-2/C-3的远程相关,最终确定2个吲哚环通过C-8′位相连。至此,确定了化合物1的平面结构。
|
| 图 2 化合物1的二维相关信号(A)和CD谱(B) Figure 2 Key 2D NMR correlations (A) and CD (B) of compound 1 |
|
|
通过与已报道的化合物3-(3-(2-Hydroxyethyl)- 1H-Indol-2-Yl)-3-(1H-Indol-3-Yl)Propane-1, 2-Diol[10-11]的CD谱比较,发现化合物1在210 nm和228 nm处有相同的Cotton效应(图 2),因此,确定化合物1的C-8′位绝对构型为S。
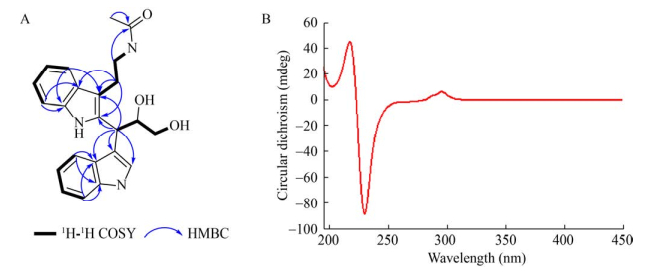
化合物2,黄色粉末,通过高分辨质谱HRESIMS (Found 414.179 5 [M+Na]+,Calcd 414.178 8)确定其分子式为C23H25N3O3,不饱和度为13。化合物2与化合物1具有相同的分子式,1H NMR和13C NMR谱数据也显示了高度的一致性(表 3)。对化合物2的1H-1H COSY、HSQC、HMBC谱数据进行详细分析,发现化合物2与化合物1具有相同的平面结构。此外,化合物2的CD谱在211 nm和228 nm显示了与化合物1相反的Cotton效应(图 3)。因此,化合物2的C-8′位的绝对构型确定为R。
|
| 图 3 化合物2的二维相关信号(A)和CD谱(B) Figure 3 Key 2D NMR correlations (A) and CD (B) of compound 2 |
|
|
化合物3:分子式C12H14N2O;1H NMR (400 MHz,MeOD) δH 7.54 (1H,d,J=7.9 Hz,H-4),7.32 (1H,d,J=8.1 Hz,H-7),7.07 (2H,m,H-2,6),6.99 (1H,t,J=7.4 Hz,H-5),3.46 (2H,t,J=7.3 Hz,H-9),2.93 (2H,t,J=7.3 Hz,H-8),1.91 (3H,s,H-12)。以上数据与文献[12]报道一致,因此被鉴定为N-乙酰色胺(3)。
化合物4:分子式C14H16N2O3;1H NMR (400 MHz,CDCl3) δH 7.56 (1H,d,J=7.8 Hz,H-4),7.32 (1H,d,J=8.1 Hz,H-7),7.07 (1H,t,J=7.2 Hz,H-6),7.07 (1H,Overlapped,H-2),6.99 (1H,t,J=7.3 Hz,H-5),3.47 (2H,t,J=7.3 Hz,H-9),2.93 (2H,dd,J=14.8,7.6 Hz,H-8),2.57 (2H,t,J=6.8 Hz,H-13),2.44 (2H,t,J=6.9 Hz,H-12);13C NMR (150 MHz,MeOD) δC 176.3(C,C-14),174.5 (C,C-11),138.2 (C,C-7a),128.8 (C,C-3a),123.4 (CH,C-2),122.3 (CH,C-6),119.6 (CH,C-5),119.3 (CH,C-4),113.3 (C,C-3),112.2 (CH,C-7),41.5 (CH2,C-9),31.6 (CH2,C-12),30.3 (CH2,C-13),26.3 (CH2,C-8)。以上数据与文献[13]报道一致,因此被鉴定为4-((2-(1H-Indol-3-Yl)Ethyl)Amino)-4-Oxobutanoic Acid (4)。
化合物5:黄色粉末;分子式C16H17N3O2;1H NMR (400 MHz,MeOD) δH 7.58 (1H,d,J=7.9 Hz,H-4),7.33 (1H,d,J=8.1 Hz,H-7),7.10 (2H,Overlapped,H-2,6),7.01 (1H,t,J=7.2 Hz,H-5),4.42 (1H,t,J=4.2 Hz,H-9),4.00 (1H,ddd,J=10.5,6.2,1.1 Hz,H-12),3.47 (1H,dt,J=11.7,8.2 Hz,H-15a),3.27 (2H,m,H-8) 3.26 (1H,m,H-15b),1.99 (1H,m,H-17a),1.69 (1H,m,H-16b) 1.48 (1H,m,H-16a),0.96 (1H,ddd,J=22.8,10.9,8.5 Hz,H-17b);13C NMR (150 MHz,MeOD) δC 170.8 (C,C-13),167.5 (C,C-10),138.0 (C,C-7a),128.7 (C,C-3a),125.6 (CH,C-2),122.6 (CH,C-6),120.0 (CH,C-5),119.8 (CH,C-4),112.3 (CH,C-7),109.6 (C,C-3),60.1 (CH,C-12),57.3 (CH,C-9),46.0 (CH2,C-15),29.2 (CH2,C-17),29.2 (CH2,C-8),22.6 (CH2,C-16)。以上数据与文献[14]报道一致,因此被鉴定为Brevianamide F (5)。
化合物6:分子式C15H26O;1H NMR (400 MHz,CDCl3) δH 5.62 (1H,dd,J=16.0,3.2 Hz,H-5),5.00 (1H,d,J=11.4 Hz,H-1),4.93 (1H,dd,J=16.0,9.9 Hz,H-6),2.39 (2H,m,H-4,2b),2.21 (3H,m,H-7,H-9),1.87 (1H,d,J=13.9 Hz,H-2a),1.67 (1H,m,H-3a),1.51 (3H,s,H-14),1.45 (1H,m,H-3b),1.24 (2H,m,H-8),1.07 (3H,d,J=6.9 Hz,H-15),1.04 (3H,s,H-12),1.12 (3H,s,H-13);13C NMR (150 MHz,CDCl3) δC 143.0 (CH,C-5),131.2 (C,C-10),130.6 (CH,C-1),123.8 (CH,C-6),72.0 (C,C-11),58.9 (CH,C-7),41.4 (CH2,C-9),33.9 (CH,C-4),32.9 (CH2,C-3),26.7 (CH3,C-13),26.5 (CH3,C-12),23.8 (CH2,C-8),22.0 (CH2,C-2),16.8 (CH3,C-14),14.8 (CH3,C-15)。以上数据与文献[15]报道一致,因此鉴定为4S, 7R-Germacra-1(10)E, 5E-Diene-11-Ol (6)。
化合物7:分子式C5H5NO2;1H NMR (400 MHz,MeOD) δH 6.94 (1H,brs,H-5),6.85 (1H,dd,J=3.6,1.2 Hz,H-3),6.18 (1H,m,H-4);13C NMR (100 MHz,MeOD) δC 163.3 (C,C-6),122.9 (CH,C-5),122.7 (C,C-2),115.1 (CH,C-3),109.2 (CH,C-4)。以上数据与文献[16]报道一致,因此鉴定为1H-吡咯-2-羧酸(7)。
2.2 产孢培养基的选择接合转移培养基需要同时满足供体菌和受体菌的生长需求。根据前期的研究选择6种培养基(ISP2、ISP3、ISP4、ISP5、ISP6、ISP7)[5]对L. rhizosphaerae NEAU-A2的产孢情况进行了研究。结果显示,NEAU-A2在ISP5培养基上生长不好,产孢较少;在ISP2、ISP3、ISP4、ISP6、ISP7培养基上都能较好生长,尤其在ISP3培养基上生长较快、产孢较多。因此,选择ISP3作为接合转移的培养基。
2.3 对不同抗生素的敏感试验建立成熟的遗传操作体系是后期对L. rhizosphaerae NEAU-A2中结构新颖且活性较好的次级代谢产物开展生物合成研究的重要基础。在遗传操作中,首先需要选用合适的抗生素抗性基因作为筛选标记,所以本研究对菌株的抗性谱进行了测定。选择实验室常用的多种抗生素进行测试(表 4),结果发现该菌株对硫酸安普霉素(Apr)、卡那霉素(Kan)、硫链丝菌素(Thio)、红霉素(Ery)较敏感,对三甲氧苄氨嘧啶不敏感(Tmp)。因此,选择常用的Apr、Kan作为L. rhizosphaerae NEAU-A2突变株的抗性筛选标记,选择Tmp作为接合转移中大肠杆菌的抑制剂。
| 抗生素种类 Antibiotics |
浓度Concentration (μg/mL) | ||||
| 25 | 50 | 75 | 100 | 200 | |
| 硫酸安普霉Apr | − | − | − | − | nt |
| 卡那霉Kan | − | − | − | − | nt |
| 硫链丝菌素Thio | − | − | − | nt | nt |
| 红霉素Ery | nt | − | − | − | − |
| 三甲氧苄氨嘧啶Tmp | + | + | + | −+ | nt |
| 注:−:不生长;+:生长;−+:抑制生长;nt:未测定 Note : −: No growth; +: Growth; −+: Poor growth; nt: Not tested |
|||||
获得大片段的基因组(≥30 kb)是构建基因组文库的首要条件,基因组提取过程中每一步操作都要温和,以防止基因组发生断裂。在对L. rhizosphaerae NEAU-A2基因组进行去磷酸化实验时(牛小肠碱性磷酸酶CIAP),发现其基因组容易降解,这一特征与常规的链霉菌基因组并不相同,给基因文库的成功构建带来了挑战。我们尝试多次实验,全过程操作在低温环境下均不能避免基因组的降解。仔细分析发现L. rhizosphaerae NEAU-A2的基因组在用Mbo Ⅰ酶切后并未发生降
解,仅在用牛小肠碱性磷酸酶(CIAP)处理后发生降解,因此推测该类去磷酸化酶及其缓冲盐体系导致基因组不稳定。所以,我们重新选择NEB公司的另外一种去磷酸化酶——虾碱性磷酸酶(rSAP)。用rSAP酶处理后,L. rhizosphaerae NEAU-A2的基因组降解现象显著改善,最终成功构建了L. rhizosphaerae NEAU-A2的基因组文库。
2.5 热激条件的选择适宜的热激有助于接合转移的顺利进行,在控制孢子量的前提下观察了3个热激时间:50 ℃分别热激5、8和10 min,结果均有接合子长出,热激8 min和10 min的效率相似且均高于5 min。接下来观察分析了孢子温浴时间对接合转移效率的影响,结果显示,温浴0、0.5、1、2 h后接合效率无显著差异。因此,最终选择50 ℃热激8 min后冷却至室温与供体菌混合。
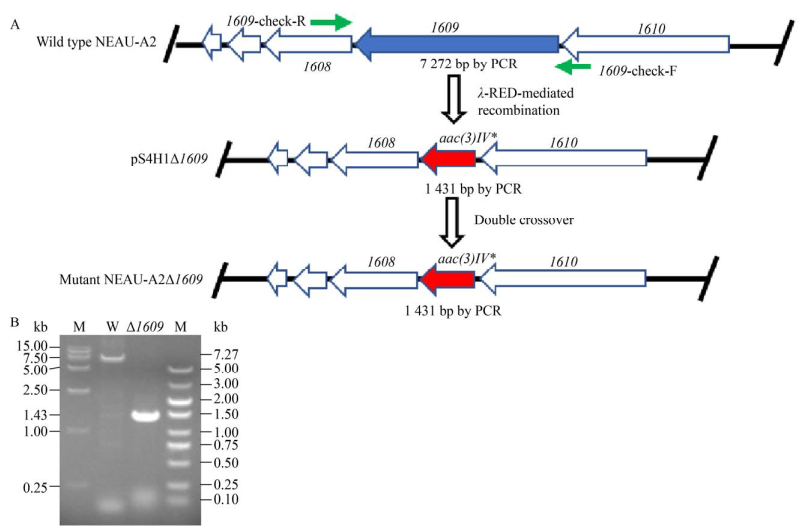
2.6 利用PCR-Targeting技术进行基因的敲除利用PCR-Targeting技术成功获得了敲除PKS-NRPS基因的突变菌株ET12567/pUZ8002/ S4H1-Δ1609-cosmid,使用验证引物(表 2)可以得到1 431 bp的Apr抗性基因片段。以ET12567/ pUZ8002/pCSG2104作为供体菌,以L. rhizosphaerae NEAU-A2菌株的孢子作为受体菌,按照上述优化后的实验条件进行接合转移。培养4–6 d,菌落几乎长满了平板,说明接合转移效率较高。然后挑取3个单菌落在含有Apr抗生素的ISP3平板上培养3–5 d后,再挑取32个单菌落接合子进行抗性筛选,结果表型均为Apr抗性,为双交换重组子。随机挑取其中几个接合子,分别在TSB种子培养基中培养36 h后收集菌体提取基因组,用验证引物1609-check-F和1609-check-R进行PCR验证,以野生型基因作为对照。S4H1-Δ1609-cosmid的双交换突变株有一条1 431 bp的条带,而对应的野生型条带是7 272 bp (图 4),表明在双交换突变株中aac(3)Ⅳ+oriT成功置换了1609基因。
|
| 图 4 PKS-NRPS基因失活突变株构建(A)和双交换突变株电泳检测图(B) Figure 4 Construction of PKS-NRPS inactive mutant (A) and gel electrophoresis analyses of the gene 1609 double exchanged mutant strain (B) 注:“aac(3)IV*”:同时引入抗性基因和oriT;M:DNA Marker;W:野生型 Note: "aac(3)IV*": Resistance gene and oriT were inserted; M: DNA Marker; W: Wild type |
|
|
本文从稀有放线菌L. rhizosphaerae NEAU-A2中分离鉴定了2个新的吲哚二聚体,据文献报道该类化合物显示了广泛的生物活性,如抗乙酰胆碱酯酶活性[10, 17]、抗衣原体活性[11]、抗肿瘤活性[18]等。从生源途径分析,吲哚二聚类化合物是由2分子的色氨酸缩合,再通过一系列的氧化形成[11]。2个新的吲哚生物碱的发现进一步丰富了天然产物中吲哚二聚体结构的多样性,将为广泛的生物活性筛选提供先导化合物。
目前,国内外抗生素产生菌的基因克隆技术大部分仍局限于链霉菌属,关于链霉菌次级代谢的分子生物学研究取得了系列进展,并且出版了相关分子生物学技术的实验手册[19],但有关稀有放线菌的遗传操作报道较少。列舍瓦列氏菌属(Lechevalieria)是一类并不常见的稀有放线菌,截至目前未见对该属遗传操作体系的报道。对L. rhizosphaerae NEAU-A2进行全基因组分析发现,该菌株有着合成丰富的次级代谢产物的潜力,因此,我们对其遗传操作体系进行研究,以期为开展生物合成机制研究奠定基础。
在基因组文库构建时,我们发现该菌株的DNA在用牛小肠碱性磷酸酶(CIAP)处理后容易发生降解,与常规链霉菌DNA的性质并不相同。这对构建基因文库造成了巨大障碍,因为构建基因文库需要一定大小片段的DNA (30–42 kb)。后续我们尝试多次及全过程操作,在低温条件下均不能避免DNA的降解。对这一结果分析探讨后,发现未用去磷酸化酶处理前基因组是稳定的,仅在处理后降解为小片段的DNA。推测是该类去磷酸化酶及其缓冲盐体系导致DNA不稳定。因此我们重新选择虾碱性磷酸酶(rSAP)进行去磷酸化处理,结果发现基因组降解显著减少,最终成功构建了L. rhizosphaerae NEAU-A2的基因文库。该菌株遗传操作体系的成功建立,将为该属中其他菌株遗传操作体系的建立提供参考,也为该菌株中相关次级代谢产物生物合成基因簇的异源表达、诱导激活等遗传学操作奠定基础。
| [1] |
Zhang K. Discovery of juanlimycin-like compounds and genome mining of two rare actinomycetes[D]. Jinan: Master's Thesis of Shandong University, 2019 (in Chinese) 张坤. Juanlimycin类化合物的发现与两株稀有放线菌的基因组挖掘[D]. 济南: 山东大学硕士学位论文, 2019 |
| [2] |
Tiwari K, Gupta RK. Rare actinomycetes: a potential storehouse for novel antibiotics[J]. Critical Reviews in Biotechnology, 2012, 32(2): 108-132. DOI:10.3109/07388551.2011.562482 |
| [3] |
Ding T, Yang LJ, Zhang WD, Shen YH. The secondary metabolites of rare actinomycetes: chemistry and bioactivity[J]. RSC Advances, 2019, 9(38): 21964-21988. DOI:10.1039/C9RA03579F |
| [4] |
Zhang JC, Yang XQ, Zhou H, Yang YB, Ding ZT. New natural products of rare actinomycetes from 2006 to 2018[J]. Chinese Journal of Organic Chemistry, 2019, 39(4): 982-1012. (in Chinese) 张举成, 杨雪琼, 周皓, 杨亚滨, 丁中涛. 2006-2018年稀有放线菌中的新天然产物[J]. 有机化学, 2019, 39(4): 982-1012. |
| [5] |
Zhao JW, Li WC, Shi LL, Wang H, Wang Y, Zhao Y, Xiang WS, Wang XJ. Lechevalieria rhizosphaerae sp. nov., a novel actinomycete isolated from rhizosphere soil of wheat (Triticum aestivum L.) and emended description of the genus Lechevalieria[J]. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(11): 4655-4659. DOI:10.1099/ijsem.0.002351 |
| [6] |
Blin K, Shaw S, Steinke K, Villebro R, Ziemert N, Lee SY, Medema MH, Weber T. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline[J]. Nucleic Acids Research, 2019, 47(W1): W81-W87. DOI:10.1093/nar/gkz310 |
| [7] |
Gust B, Challis GL, Fowler K, Kieser T, Chater KF. PCR-targeted Streptomyces gene replacement identifies a protein domain needed for biosynthesis of the sesquiterpene soil odor geosmin[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(4): 1541-1546. DOI:10.1073/pnas.0337542100 |
| [8] |
Gust B, Chandra G, Jakimowicz D, Tian YQ, Bruton CJ, Chater KF. λ red-mediated genetic manipulation of antibiotic-producing Streptomyces[J]. 2004, 54: 107-128
|
| [9] |
Kieser T, Bibb MJ, Buttner MJ, Chater KF, Hopwood DA. Practical Streptomyces Genetics[M]. Norwich: John Innes Foundation, 2002.
|
| [10] |
Li JL, Huang L, Liu J, Song Y, Gao J, Jung JH, Liu YH, Chen GT. Acetylcholinesterase inhibitory dimeric indole derivatives from the marine actinomycetes Rubrobacter radiotolerans[J]. Fitoterapia, 2015, 102: 203-207. DOI:10.1016/j.fitote.2015.01.014 |
| [11] |
Li J, Chen DD, Huang L, Ni M, Zhao Y, Fan HZ, Bao XF. Antichlamydial dimeric indole derivatives from marine actinomycete Rubrobacter radiotolerans[J]. Planta Medica, 2017, 83(9): 805-811. DOI:10.1055/s-0043-100382 |
| [12] |
Maeda U, Hara N, Fujimoto Y, Srivastava A, Gupra YK, Sahai M. N-fatty acyl tryptamines from Annona reticulata[J]. Phytochemistry, 1993, 34(6): 1633-1635. DOI:10.1016/S0031-9422(00)90860-4 |
| [13] |
Soriano-Agatón F, Lagoutte D, Poupon E, Roblot F, Fournet A, Gantier JC, Hocquemiller R. Extraction, hemisynthesis, and synthesis of canthin-6-one analogues. Evaluation of their antifungal activities[J]. Journal of Natural Products, 2005, 68(11): 1581-1587. DOI:10.1021/np050250z |
| [14] |
Asiri IAM, Badr JM, Youssef DTA. Penicillivinacine, antimigratory diketopiperazine alkaloid from the marine-derived fungus Penicillium vinaceum[J]. Phytochemistry Letters, 2015, 13: 53-58. DOI:10.1016/j.phytol.2015.05.014 |
| [15] |
Ganßer D, Pollak FC, Berger RG. A sesquiterpene alcohol from Streptomyces citreus CBS 109.60[J]. Journal of Natural Products, 1995, 58(11): 1790-1793. DOI:10.1021/np50125a027 |
| [16] |
Guan YQ, Luo Y, Shi LL, Gulinar S, Chen G, Guo XF, Luo DQ. Isolation of secondary metabolites from Streptomyces roseofulvus M63 by OSMAC methods and their anti-tumor activity[J]. Natural Product Research and Development, 2016, 28(12): 1864-1869, 1914. (in Chinese) 关永强, 罗影, 石磊岭, 古丽娜·沙比尔, 陈刚, 郭雄飞, 罗都强. 采用OSMAC法对Streptomyces roseofulvus M63次级代谢产物及其抗肿瘤成分的研究[J]. 天然产物研究与开发, 2016, 28(12): 1864-1869, 1914. |
| [17] |
Queiroz MMF, Queiroz EF, Zeraik ML, Ebrahimi SN, Marcourt L, Cuendet M, Castro-Gamboa I, Hamburger M, Da Silva Bolzani V, Wolfender JL. Chemical composition of the bark of Tetrapterys mucronata and identification of acetylcholinesterase inhibitory constituents[J]. Journal of Natural Products, 2014, 77(3): 650-656. DOI:10.1021/np401003p |
| [18] |
Yang X, Liu JW, Huo ZQ, Yuwen HS, Li Y, Zhang Y. Fluevirines E and F, two new alkaloids from Flueggea virosa[J]. Natural Product Research, 2020, 34(14): 2001-2006. DOI:10.1080/14786419.2019.1569661 |
| [19] |
Huan MH, Chen F, Wu HY, Guan YL, Wang Z, Cheng YT. Construction and initial screening of gene library of the Streptomyces ahygroscopicus[J]. Biotechnology Bulletin, 2009(1): 134-137. (in Chinese) 桓明辉, 陈飞, 吴红艳, 关艳丽, 王志, 程永涛. 不吸水链霉菌基因文库的构建及初步筛选[J]. 生物技术通报, 2009(1): 134-137. |
 2021, Vol. 48
2021, Vol. 48




