
扩展功能
文章信息
- 李亚鹏, 徐轶凡, 陈红歌
- LI Yapeng, XU Yifan, CHEN Hongge
- 纤维二糖磷酸化酶和纤维寡糖磷酸化酶的研究与应用
- Research and application of cellobiose phosphorylase and cellodextrin phosphorylase: a review
- 微生物学通报, 2021, 48(6): 2245-2258
- Microbiology China, 2021, 48(6): 2245-2258
- DOI: 10.13344/j.microbiol.china.210005
-
文章历史
- 收稿日期: 2021-01-04
- 接受日期: 2021-02-09
- 网络首发日期: 2021-03-23
木质纤维素是地球上含量最为丰富的可再生生物质资源,其高效降解和利用对于解决人类面临的环境污染、能源危机和食品短缺等问题具有重大的意义。然而,木质纤维素的结构坚韧性使得其降解环节成为纤维素资源利用的瓶颈,自然界中存在的纤维素降解微生物为人类利用纤维素资源提供了广泛的水解纤维素的酶类资源。这些纤维素降解微生物通常会分泌内切葡聚糖酶(Endo-1, 4-β-D-Glucanase,EG)和纤维二糖水解酶(Cellobiohydrolase,CBH)作用于天然纤维素,产生纤维二糖和纤维寡糖,纤维二糖和纤维寡糖再进一步地被微生物分泌或胞内的β-葡萄糖苷酶(β-Glucosidase,βG)水解,生成葡萄糖被利用[1-2]。对于一些厌氧纤维素降解菌,如Clostridium stercorarium[3]、Ruminococcus albus[4]等,它们除了有βG水解纤维二糖或纤维寡糖,还产生纤维二糖磷酸化酶(Cellobiose Phosphorylase,CBP)和纤维寡糖磷酸化酶(Cellodextrin Phosphorylase,CDP),分别磷酸化裂解纤维二糖和纤维寡糖,磷酸化裂解产物同样被细胞利用。磷酸化酶CBP和CDP与βG一样,是自然界微生物产生的纤维素降解酶系的组分,它们与EG、CBH酶组分协同作用,共同将纤维素基质降解成微生物可利用的碳源和能源。
不同于βG对纤维寡糖底物的水解作用,CBP (EC 2.4.1.20)和CDP (EC 2.4.1.49)是通过磷酸化裂解纤维寡糖实现对底物的降解。其中,CBP作用于纤维二糖,生成1-磷酸-葡萄糖(G-1-P)和葡萄糖,而CDP则作用于聚合度(Degree of Polymerization,DP) ≥3的纤维寡糖,如纤维三糖、纤维四糖、纤维五糖等,生成G-1-P和少1个葡萄糖残基的纤维寡糖。依赖于CBP和CDP的纤维二糖和纤维寡糖的磷酸化裂解(以下简称磷解)过程在酶的来源、酶催化机制、ATP生成、应用领域等多个方面均与依赖βG的纤维二糖和纤维寡糖的水解过程存在显著不同,有关βG的综述文章已有发表[5-6],本文将综述CBP和CDP酶类的研究和应用情况。
1 CBP和CDP的来源CBP于20世纪50年代在Clostridium thermocellum菌中首次被发现[7]。迄今为止,人们先后在多种微生物中发现了CBP,如Clostridium thermocellum[8]、Cellvibrio gilvus[9]、Cellulomonas species[10]、Fomes annosus[11]、Clostridium stercorarium[3]、Thermotoga neapolitana[12]、Cellulomonas uda[13]、Thermotoga maritima[14]、Ruminococcus albus[15]、Ruminococcus flavefaciens[16]和Thermotoga petrophila[17]等。CDP则是于20世纪60年代末同样首先在Clostridium thermocellum中被发现[18],之后陆续在Clostridium stercorarium[3]、Clostridium thermocellum YM4[19]、Saccharophagus degradans[20]、Ruminococcus albus[21]和Clostridium cellulosi[22]等微生物中发现。可以看到无论是CBP还是CDP,它们的来源菌均属于厌氧菌,多分布于生存条件较为严酷的环境中。
CBP和CDP均属于胞内酶,它们的反应产物G-1-P以及葡萄糖直接被细胞利用而不存在跨膜转运的问题。然而,大多数的βG是胞外酶,仅有小部分属于胞内酶。在自然界中一些微生物同时具有βG、CBP和CDP,这些菌种在利用纤维寡糖时大多表现出对磷解途径的偏好。例如,Clostridium thermocellum同时具有胞内的磷酸化酶和βG,但其细胞提取液对纤维二糖的磷解速率比水解速率高20倍以上[23];在Ruminococcus albus中,其胞内通过磷解途径利用纤维二糖的比例也超过90%[24]。这表明微生物在利用纤维寡糖的过程中,磷解途径可能比水解途径更具有代谢优势。
对于微生物来说,纤维寡糖降解过程中由CBP或CDP参与的磷解比βG参与的水解具有的代谢优势可以由纤维寡糖代谢过程中的产能优势来解释。以纤维二糖的降解为例(图 1),水解途径与磷解途径的最终产物均进入糖酵解(Embden- Meyerhof-Parnas Pathway,EMP)途径:葡萄糖直接进入EMP途径,而G-1-P异构为葡萄糖-6-磷酸(G-6-P)后进入EMP途径。1分子葡萄糖经过EMP途径生成丙酮酸时净生成2分子ATP,而1分子G-1-P经G-6-P进入EMP途径生成丙酮酸的过程则由于少消耗1个ATP而净生成3分子ATP。纤维二糖经过水解,产生的2分子葡萄糖进入EMP净生成4分子ATP,而纤维二糖经过磷解,产生的1分子葡萄糖和1分子G-1-P进入EMP则净生成5分子ATP。相较于水解途径,磷解途径能够为来源微生物提供多1分子的ATP,因而更有利于微生物的生长。
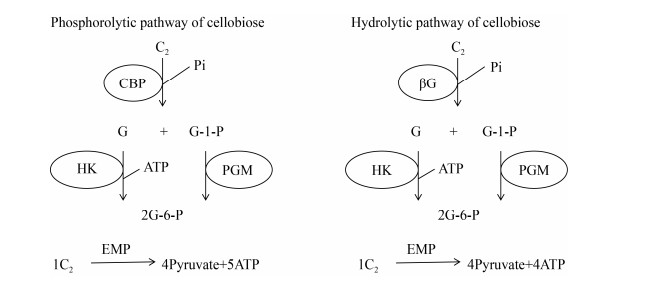
|
| 图 1 纤维二糖的磷解途径相较于水解途径在ATP生成上的优势 Figure 1 The advantage of the phosphorolytic pathway compared to the hydrolytic pathway in ATP production during cellobiose degradation 注:C2:纤维二糖;G:葡萄糖;G-6-P:葡萄糖-6-磷酸;HK:己糖激酶;PGM:磷酸葡萄糖变位酶 Note: C2: Cellobiose; G: Glucose; G-6-P: Glucose-6-phosphate; HK: Hexokinase; PGM: Phosphoglucomutase |
|
|
目前已有来自不同菌种的9种CBP与3种CDP被报道有详细的酶学性质的表征。根据酶的来源菌种的不同,各酶之间的氨基酸序列、蛋白大小、最适温度、最适pH和底物特异性等方面均有一定的差别。根据BRENDA数据库将已有详细报道的CBP和CDP的酶学性质进行汇总,如表 1所示。

|
不同来源的CBP与CDP分子量从91 kD到280 kD不等,其中最大的是来自Cellulomonas gilvus的CBP,分子量为280 kD[25]。不同来源的CBP之间氨基酸序列具有一定的同一性,大约在60%-90%之间,例如:来自Thermotoga neapolitana和Thermotoga petrophila的CBP同一性为89.7%;而来自Cellulomonas uda和Clostridium stercorarium的CBP同一性较低,为61.4%。不同来源的CDP之间氨基酸序列同一性较低,约在40%,例如来自Clostridium thermocellum和Clostridium cellulosi的CDP氨基酸序列之间的同一性为44.3%。
CBP与CDP的最适催化温度大多在60-80 ℃之间,根据来源菌种所处环境的不同,各酶存在一定差异。例如厌氧菌Clostridium thermocellum的最适生长温度为60-65 ℃,其产生的CBP最适催化温度为60 ℃[8];最适生长温度为80 ℃的超嗜热菌Thermotoga maritima所产生的CBP最适催化温度为80 ℃[19];同样来自高温环境的嗜热菌Thermotoga petrophila产生的CBP最适催化温度也达到了80 ℃[17];从地热嗜热微生物菌群中分离出的超嗜热菌Thermotoga neapolitana,其产生的CBP最适催化温度则高达85 ℃[12];而所处环境温度在39-41 ℃之间的瘤胃细菌Ruminococcus albus所产生的CBP最适催化温度则相对较低,为50 ℃[15]。由于CBP与CDP同属于胞内酶,它们所催化的反应均在微生物细胞质中进行,而胞质是一个酸碱度偏向中性、相对温和的反应环境,因此,不同来源菌种的CBP与CDP最适作用pH的差异并不显著,基本都在6.0-8.0之间。
CBP和CDP属于转移酶类,催化的是可逆反应。也就是说,CBP和CDP除了催化纤维寡糖的磷解之外,还可以进行逆向的合成反应,即以葡萄糖或纤维寡糖作为糖基受体,将G-1-P上的D-葡萄糖转移至其上产生聚合度高一级的纤维寡糖并释放出无机磷。以CBP为例,在磷解反应方向,CBP在无机磷的存在下将纤维二糖分解为1分子G-1-P和1分子葡萄糖;在合成反应方向,CBP则利用G-1-P和葡萄糖合成纤维二糖。我们可以看到,在所列出的CBP与CDP催化的双向反应中,大多数酶在磷解方向与合成方向上具有相当的反应速率,例如来自Cellulomonas gilvus的CBP[26]与来自Cellulomonas uda的CBP[13]。另一些酶在2个方向上的反应速率存在显著差异,例如来自Ruminococcus albus的CBP和CDP,前者磷解方向反应速率要高于合成方向[15];后者则相反,更加倾向于合成方向的反应[21]。
除上述酶外,Wu等鉴定了来自Thermosipho africanus TCF52B菌株的THA_1941基因的功能,虽然该蛋白与所有已知GH94家族的CBP酶的同一性均低于12%,但却显示了CBP的活性,而且该酶在合成方向还能利用纤维二糖和聚合度大于2的纤维寡糖作为糖基受体,显示了CDP酶的功能[33];催化效率(kcat/Km)测定表明,纤维四糖和纤维五糖分别是其磷解和合成反应的最优底物,并且其合成方向的反应速率远高于磷解方向;由于该酶在合成方向上更倾向于使用纤维二糖和纤维寡糖而不是葡萄糖作为糖基受体,因此将其最终定义为TaCDP;2019年,Kuhaudomlarp等进一步证实了该酶对β-1, 3糖苷键专一,而非CBP和CDP催化的β-1, 4糖苷键,因此将其重新定义为糖苷磷酸化酶(Glycoside Phosphorylase,GP),并归类为糖苷水解酶161家族(GH161)[34]。
此外,在CBP和CDP催化的合成反应中,除了葡萄糖或纤维寡糖可以被用作糖基受体外,很多酶能够将其他糖类(如木糖、甘露糖、脱氧葡萄糖等)用作糖基受体并具有很高的活性,这显示了CBP与CDP在特殊糖类的合成方面具有潜在的应用价值。
3 CBP和CDP的催化机制及其催化结构的差异已经研究清楚的是,CBP在催化可逆的磷解反应时遵循连续的bi-bi机制,即反应时各底物按照一定的顺序与酶结合,同时反应产物按照一定的顺序释放。但对于不同的CBP,其底物的结合与产物的释放顺序存在一定的差异。在磷解方向上,来源于Cellvibrio gilvus[35]和Cellulomonas uda[13]的CBP首先与纤维二糖结合,再与磷酸基团结合,反应后先释放D-葡萄糖,再释放G-1-P。来源于Clostridium thermocellum的CBP在产物释放顺序与上述2个CBP酶相同,但是底物结合顺序则相反,即酶与磷酸基团的结合在前,而与纤维二糖的结合在后[36]。此外,来源于Thermotoga maritima[14]和Ruminococcus albus[15]的CBP则是以随机的顺序与底物结合。
目前仅有3种不同来源的CBP经过X射线衍射解析出晶体结构,分别为来自Clostridium thermocellum的CBP (CtCBP)[37]、来自Cellvibrio gilvus的CBP (CgCBP)[38]和来自Cellulomonas uda的CBP (CuCBP)[39]。截至目前,仅有来自Clostridium thermocellum的CDP (CtCDP)得到了明确的晶体结构[40]。它们的蛋白质三维结构大致相似,均由2个相同的亚基组成,每个亚基又分为了4个不同的结构——N端β-三明治结构域、螺旋连接体、(α/α)6-桶装结构域和C端的卷曲状结构域;酶的2个亚基通过一系列的疏水相互作用和氢键结合在一起;二聚体界面由(α/α)6-桶状结构域和位于另一亚基中的β-三明治结构域组成,这2个结构域之间的相互作用构成了酶的活性中心[37]。
以CtCBP为例,其单亚基由811个氨基酸组成,是一种模块化蛋白,属于糖苷水解酶94家族,由4个不同的结构域组成(图 2),各个结构域的位置如下:1-279氨基酸残基为N端β-三明治结构域;280-314氨基酸残基为螺旋连接体;321-734氨基酸残基为(α/α)6-桶状结构域;315-320氨基酸残基和735-811氨基酸残基为C端的卷曲状结构域[37]。N端β-三明治结构域由18个反向平行的β链形成相互堆叠的β折叠片组成,之后由形成90°弯曲的2个α-螺旋组成螺旋连接体,N端β-三明治结构域通过这个螺旋连接体与(α/α)6-桶装结构域相连;(α/α)6-桶装结构域由2个同心环形成,而这2个同心环由6个α-螺旋形成,其中包含有几个高度保守的位点,比如His653、Gln699和Thr718,它们的侧链与Gly719的主链N原子在桶状结构域内协调酶与磷酸的结合,并且催化残基Asp483也位于桶状结构域中心附近;C端结构域则采用双层卷曲折叠[37]。在CtCBP中,N端β-三明治结构域和(α/α)6-桶状结构域之间的相互作用决定了底物口袋的大小。一个亚基的2个短螺旋(残基158-169)延伸至另一亚基(α/α)6-桶状结构域形成的二聚体界面中,辅助了活性位点口袋的形成;另一个环(残基488-507)位于活性位点口袋的顶部,对其大小进行了限制,使口袋大到足以结合二糖,但小到不能够容纳聚合度≥3的寡糖,从而使酶可以基于寡糖长度识别不同的底物[37]。
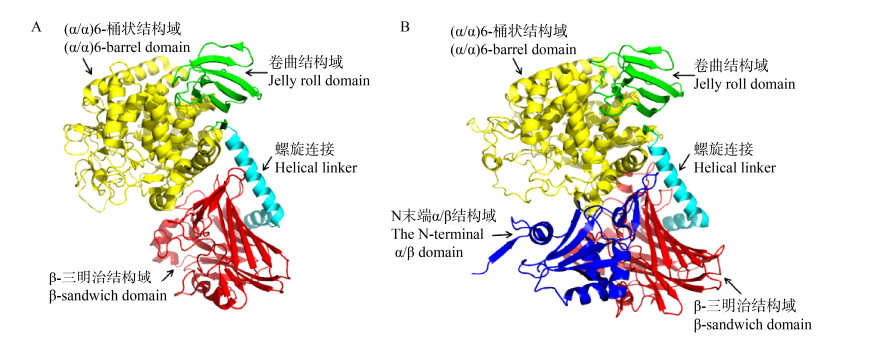
|
| 图 2 CtCBP (A)和CtCDP (B)的单体结构 Figure 2 The monomer structures of CtCBP (A) and CtCDP (B) 注:β-三明治结构域用红色标出;螺旋连接用青色标出;(α/α)6-桶状结构域用黄色标出;卷曲结构域用绿色标出;CtCDP的N末端α/β结构域用蓝色标出 Note: β-sandwich domains are colored red; Helical linkers are colored cyans; (α/α)6-barrel domains are colored yellow; Jelly roll domains are colored green; The N-terminal α/β domain of CtCDP is colored blue |
|
|
CtCBP的整体折叠与其他GH94成员(如CgCBP和CuCBP)相同,同样来自Clostridium thermocellum的CtCDP与这些酶在结构上也具有一定的相似度(图 2)。CtCDP包含一个大的β-三明治结构域,该结构形成了大部分的二聚体界面,通过2个α-螺旋连接至(α/α)6-桶状催化结构域,并以一个小的采用双层卷曲折叠的外围结构域结尾。CtCDP还在N端存在一个独特的由120个氨基酸残基组成的α/β结构,这个α/β结构域与N端β-三明治结构均与相对亚基上的β-三明治结构域存在相互作用,这大大增加了二聚体界面的面积[40]。此外,同样采用X-射线晶体学方法对来自Thermosipho africanus TCF52B的TaCDP (后被命名为GP)底物宽泛性的结构基础进行了研究,发现相较于已解析的CtCDP,其在(α/α)6-桶状催化结构域内紧邻底物结合口袋的位置存在着较长的具有高度柔性的插入序列,这可能会引起底物口袋入口大小发生变化,进而影响其底物特异性[41]。
由图 2可以看到,来源于同一菌株的CtCBP与CtCDP在结构和功能上都非常相似,但二者的底物选择性具有很大差异。与CtCBP相比,CtCDP所催化的底物种类更加宽泛,可以作用于聚合度≥3的纤维寡糖,而CtCBP只能够以纤维二糖为底物。O’Neill等从结构差异方面对这种现象做出了解释:CtCBP的活性位点要比CtCDP更加封闭[40]。这主要是由于3个结构差异造成(图 3):(1) 二者位于底物口袋入口处的催化环(Catalytic Loop)长度不同,CtCDP要比CtCBP长12个氨基酸残基。虽然在包含催化关键位点Asp624的部分上,二者结构相似,但在CtCDP中从Ile628之后采用了与之前完全不同的构象。对于CtCDP单体亚基,该环继续远离活性位点,形成一个从蛋白质表面突出的螺旋结构,之后返回蛋白质核心。在另一个亚基中,在螺旋结构之后的一小段环路是无序的。这有助于CtCDP单个亚基与底物的结合。另一方面,环路除了带有Trp622和Asp624的部分外更多区域是无序的。相比之下,CtCBP催化环则是完全有序的,其C端部分折叠在活性位点口袋之上,限制了底物口袋入口的大小;(2) 紧靠催化环的邻近环(Adjacent Loop)对活性位点口袋也有所冲突,但这种影响对CtCDP相对较小,因为CtCDP中的邻近环比CtCBP中对应的环短5个氨基酸;(3) CtCBP和CtCDP间的第3个显著差异则涉及二聚体界面如何由2个亚基形成。与CtCDP相比,由于CtCBP蛋白质围绕着一个垂直并且几乎等分二聚体的轴进行了整体旋转,因此在CtCBP中2个亚基的相对位置有所不同,这种不同缩小了2个亚基之间的距离,从而把相对亚基上的相反环拉向活性位点;显著缩小了CtCBP的底物口袋,使其没有多余的空间容纳更大的底物[40]。然而在活性位点更加开放的CtCDP中,组成底物口袋的β-三明治结构域内存在2个α-螺旋,这2个螺旋的侧链与底物之间具有相互作用,有助于底物与活性位点的结合。CtCDP独特的N末端α/β结构域也可能存在着一定的作用,其与相对亚基的β-三明治结构域之间的相互作用会使亚基下部聚集在一起,导致上部分开,从而导致二聚体的2个单体之间狭缝体积增加,使活性位点可以与更大的底物结合[40-41]。
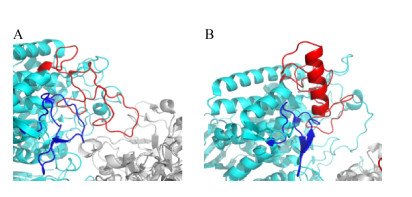
|
| 图 3 CtCBP (A)和CtCDP (B)底物口袋间的差异 Figure 3 Comparison of substrate-binding pocket between CtCBP (A) and CtCDP (B) 注:催化环用红色标出;邻近环用蓝色标出 Note: Catalytic loops are colored red; Adjacent loops are colored blue |
|
|
纤维素作为世界上存量最大的生物质资源,其广泛存在于农林废弃物、城市生活垃圾中,是一种重要的可再生资源。CBP和CDP因其可以磷解纤维素降解的重要中间产物纤维二糖和聚合度≥3的纤维寡糖,是纤维素降解酶系中重要的一员。因此,如何使CBP和CDP在纤维素降解与再利用的过程中发挥最大的作用成为近些年来的新课题。目前,CBP与CDP的应用主要体现在3个方面。
4.1 用于构建直接利用纤维寡糖的工程酵母菌能源的愈加匮乏使得人们开始探索石油等化石燃料的替代能源,以木质纤维素为原料生产纤维乙醇已成为生物能源开发的热点领域。纤维乙醇的生产通常需要组合原料预处理和酶水解等方法使难降解的木质纤维素彻底水解为单糖,再由微生物发酵生产乙醇。由于木质纤维素水解过程产生的包含纤维二糖在内的纤维寡糖中间产物会对纤维素酶产生显著的抑制作用[42],木质纤维素的充分水解成为制约纤维乙醇生产效率的重要瓶颈。构建可以直接利用纤维寡糖的工程酵母菌株进行乙醇发酵,不仅可以避免纤维寡糖对纤维素酶的抑制作用,有助于提高酶解效率,而且也不必向纤维素酶中额外添加βG组分以强化纤维寡糖的水解,因此可以大大降低木质纤维素原料的水解成本。
Sekar等报道了第一个可在胞内表达CBP的大肠杆菌工程菌,其可以磷解并利用纤维二糖生长[43]。接着构建了可以同时水解和磷解纤维二糖的大肠杆菌工程菌,该工程菌株显示了比仅有单一降解途径的大肠杆菌工程菌更高的纤维二糖利用率[44]。说明利用CBP构建直接利用纤维二糖的工程菌株具有可行性和实用性。
酿酒酵母作为生产纤维乙醇的最有竞争力的菌株,具有易生长、易于遗传操作和乙醇产量高等优点[45],但其不能够直接发酵纤维二糖等纤维寡糖。构建可以直接利用纤维寡糖的工程酵母菌株有以下3个策略:(1) 构建可以分泌或表面展示βG的酵母工程菌[46],使酵母菌利用纤维寡糖在胞外降解产生的葡萄糖进行发酵产生乙醇。但这种菌株不能够分泌或展示足够量的酶,仍然需要添加外源性的βG,因此并没有根本解决木质纤维素的水解瓶颈。(2) 将纤维寡糖转运蛋白和βG转入酵母菌中,构建可以摄取纤维寡糖并在胞内进行水解和利用的工程菌株。已有研究表明这种工程菌株相较于第一种方法更加经济,仅需通过提高酵母细胞的浓度便可达到与传统酵母菌株发酵几乎相同甚至更高的乙醇生产率,而不需要额外添加βG[47]。然而葡萄糖作为βG的抑制剂,其在胞内的大量产生限制了酶的活性,导致工程菌株性能低下。(3) 构建胞内磷解纤维寡糖的工程酵母菌。Sadie等构建了过表达乳糖透过酶并胞内表达来源于Clostridium stercorarium的CBP的酵母菌,获得了可以摄取并磷解纤维二糖的工程菌株[48]。同时也有研究将分别来自Cellvibrio gilvus、Saccharophagus degradans和Clostridium thermocellum的CBP与来自粗糙脉孢菌(Neurospora crassa)的纤维寡糖转运蛋白转入酵母菌中,构建了通过磷解途径发酵纤维二糖产生乙醇的酵母工程菌,同时也通过将来源于粗糙脉孢菌的βG转入酵母菌构建了以水解途径利用纤维二糖的工程菌株,该研究结果证实在稳态的厌氧条件下,磷解菌株细胞生物量与乙醇产生量相较于水解菌株至少有28%和32%的增加量;在酵母菌中引入纤维二糖的磷解途径比引入水解途径在ATP生成方面更具优势,特别是在ATP供不应求或是在需要更多ATP的生理条件下,磷解途径赋予的生长优势更显著[49]。因此,以CBP和CDP为核心构建直接利用纤维寡糖的工程酵母菌将会成为今后纤维乙醇生产工业中的一个重要方向。
4.2 酶法纤维素转淀粉体系全球人口不断增加造成粮食需求大幅提升,如何解决日益严重的粮食危机成为科研工作者的重要课题。淀粉作为人类最重要的能源食物,若能以来源广泛的纤维素为原料进行人工合成,即纤维素转淀粉,不仅能够解决人类粮食供给问题,还可以为农业废弃物处理及再利用提供解决方案。You等成功构建了体外酶法纤维素转淀粉体系[50],实现了非粮纤维素到直链淀粉的转化。
酶法纤维素转淀粉体系分为2个模块:在纤维素水解模块中,纤维素在来自枯草芽胞杆菌的内切β-1, 4-葡聚糖酶(BsCel5)和来自Clostridium phytofermentans的纤维二糖水解(CpCel48)共同作用下生成中间产物纤维二糖;之后纤维二糖在淀粉合成模块中被来自Clostridium thermocellum的CtCBP磷解为葡萄糖和G-1-P;最后G-1-P被来自马铃薯的葡聚糖磷酸化酶加合到麦芽寡糖链上生成直链淀粉。此体系具有2个优点:(1) 纤维素水解模块对纤维素的降解仅进行到纤维二糖,大大减少了完全水解终产物葡萄糖对水解酶的产物抑制作用,降低了纤维素水解的难度;(2) 淀粉合成模块中的G-1-P是由CtCBP催化纤维二糖磷解过程中产生的,整个体系的反应过程中没有任何高能物质的加入。相比之下,生物体合成淀粉均需要ATP或者GTP参与活化葡萄糖,是一个高耗能的过程。体外酶法纤维素转淀粉体系具有原料易得、反应易放大、无ATP参与等优势,产生的淀粉不仅可以用作实验研究,还可用作功能食品、动物饲料等,具有重要的研究和开发价值,是今后人工合成淀粉的一个重要途径。
4.3 酶法合成特殊寡糖由于CBP和CDP能催化独特的可逆磷解反应,因此,CBP和CDP被认为是合成各种特殊糖类的有效酶。近年来已有研究人员在大肠杆菌中共表达来自Bifidobacterium adolescentis的蔗糖磷酸化酶和来自Cellulomonas uda的CBP,利用它们催化的合成反应将蔗糖和葡萄糖高效地转化为纤维二糖,此方法有望用于纤维二糖的工业化生产[51]。Ubiparip等[52]通过对来自Cellulomonas uda的CBP进行饱和突变,获得的优化突变体能以高达73%的产率将纤维二糖转化为纤维三糖,并且在最终产物中纤维三糖的含量达到82%,获得了迄今为止已报道的最高纯度的纤维三糖产物。
可溶性纤维寡糖的酶法合成同样使用了CBP和CDP催化的合成反应。Zhong等[22]通过构建来自Cellulomonas uda的CBP和来自Clostridium cellulosi的CDP级联反应体系,以廉价底物葡萄糖为起点,合成了聚合度在3-6之间的可溶性纤维寡糖,这为批量化生产可溶性纤维寡糖提供了新思路。
此外,由于CBP在合成方向上催化生成与α构型的糖基供体G-1-P具有相反端基构型的产物,因此其可以被用于合成多种β构型的特殊糖类,例如β-葡萄糖苷。自然界中有许多生物大分子以β-葡萄糖苷的形式存在,其被广泛地用于药物、洗涤剂和食品添加剂中。Kino等使用来自Clostridium thermocellum的CBP合成烷基β-葡萄糖苷[53],而烷基β-葡萄糖苷是有效的非离子型表面活性剂,具有良好的抗菌功能。De Winter等论证了CBP具有非常高的酶促催化合成β-葡萄糖苷的潜在价值,通过酶的固定化技术和离子溶液与有机助溶剂的加入,扩展了来自Clostridium thermocellum的CtCBP的糖基化潜力并提高了产物收率,具有出色的可操作性[54]。
因为CBP和CDP具有非常广泛的受体特异性,它们还可被用于合成各种特殊二糖和支链三糖。已有文献报道CtCBP可以有效合成4-o-β-d-吡喃葡萄糖基-d-阿拉伯糖等5种杂二糖[55],CgCBP可以合成β-d-Gclp(1→4)-[α-d-Galp-(1→6)]-d-Gclp等3种支链三糖[56]。这些特殊寡糖作为重要的药物前体,具有广泛的应用。另一项研究证明CtCDP具有产生新型寡糖β-Glcp-[(1→4)-β-Glcp]n-(1→2)- Glcp和β-Glcp-[(1→4)-β-Glcp]n-(1→3)-Glcp (n=1–7)的能力,研究也表明CtCBP和CtCDP在分别以葡萄糖和纤维二糖为糖基受体催化合成方向反应时,以氟化物G-1-Fluoride代替G-1-P作为糖基供体时,产物收率分别提高到98%和68%[57],这为CBP和CDP合理的工程化生产应用奠定了基础。
5 结语世界能源危机和粮食危机的加剧使得人们意识到利用木质纤维素进行可持续发展的必要性。CBP与CDP作为纤维素降解酶系中重要的组成部分,由于其特殊的催化方式以及在ATP生成方面的优势,受到了研究人员的格外关注。截至目前,已有多种CBP和CDP被发现并显示其应用价值。
对CBP和CDP的进一步研究和应用需要特别关注以下工作:(1) 发掘新型CBP和CDP。根据已有资料,CBP和CDP的来源菌种均属于厌氧菌,这为我们寻找新酶提供了依据。当我们基于KEGG数据库寻找新酶时,就可以将重点放在来自深海、肠道或油层等极端环境的厌氧菌种上。例如超嗜热细菌Thermotoga naphthophila RKU-10和Thermotoga sp. RQ2在KEGG数据库中就具有被注释为CBP的基因,对这些候选基因进行表达和分析,有很大可能鉴定出新型CBP酶。(2) 通过定向进化提高酶的催化活性和热稳定性。自然界中天然CBP和CDP的活性及热稳定性是与其菌株的生长代谢状态相适应的,具有更高的催化活力和更强的热稳定性是体外酶催化体系中所期望的特性,通过定向进化的策略可以有效地对酶进行改造以达到这个目的,同时定向进化还可以拓宽酶的底物谱、扩展酶的应用范围等。(3) 寻找新酶或改造已有酶以获得纤维寡糖通用型的磷酸化酶,这样仅用一种酶便可以磷解纤维二糖、纤维三糖和纤维四糖等纤维寡糖。这种兼具有CBP和CDP功能的双功能磷酸化酶用于构建直接利用纤维寡糖的工程菌株或应用于酶法纤维素转淀粉体系中,则可以使纤维素部分水解的所有中间产物都能进入下一步的转化,从而大大提高原料的利用率和终产物得率。(4) 对于构建直接利用纤维寡糖的工程菌株来说,还要同时开展高效纤维寡糖转运蛋白的筛选工作。因为酿酒酵母不具有吸收纤维寡糖的能力,若使工程菌株胞内磷解途径正常进行,还需要同时构建一个与CBP/CDP磷解过程相适配的纤维寡糖转运体系。因此,获得一种可以在酿酒酵母中高效表达、具有强大的纤维寡糖转运能力并较少扰动细胞代谢的纤维寡糖转运蛋白,对于直接利用纤维寡糖的酵母工程菌的构建和优化十分必要。CBP和CDP也因此会在纤维乙醇研发中显现更重要的作用。
| [1] |
Tian C, Beeson WT, Iavarone AT, Sun JP, Marletta MA, Cate JHD, Glass NL. Systems analysis of plant cell wall degradation by the model filamentous fungus Neurospora crassa[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52): 22157-22162. DOI:10.1073/pnas.0906810106 |
| [2] |
Feng Y, Jiang J, Zhu L. Recent developments in activities, utilization and sources of cellulase[J]. Forestry Studies in China, 2009, 11(3): 202-207. DOI:10.1007/s11632-009-0028-z |
| [3] |
Reichenbecher M, Lottspeich F, Bronnenmeier K. Purification and properties of a cellobiose phosphorylase (CepA) and a cellodextrin phosphorylase (CepB) from the cellulolytic thermophile Clostridium stercorarium[J]. European Journal of Biochemistry, 1997, 247(1): 262-267. DOI:10.1111/j.1432-1033.1997.00262.x |
| [4] |
Devendran S, Abdel-Hamid AM, Evans AF, Iakiviak M, Kwon IH, Mackie RI, Cann I. Multiple cellobiohydrolases and cellobiose phosphorylases cooperate in the ruminal bacterium Ruminococcus albus 8 to degrade cellooligosaccharides[J]. Scientific Reports, 2016, 6: 35342. DOI:10.1038/srep35342 |
| [5] |
Ketudat Cairns JR, Esen A. β-Glucosidases[J]. Cellular and Molecular Life Sciences, 2010, 67(20): 3389-3405. DOI:10.1007/s00018-010-0399-2 |
| [6] |
Salgado JCS, Meleiro LP, Carli S, Ward RJ. Glucose tolerant and glucose stimulated β-glucosidases: a review[J]. Bioresource Technology, 2018, 267: 704-713. DOI:10.1016/j.biortech.2018.07.137 |
| [7] |
Hammerstrom RA, Claus KD, Coghlan JW, McBee RH. The constitutive nature of bacterial cellulases[J]. Archives of Biochemistry & Biophysics, 1955, 56(1): 123-129. |
| [8] |
Tanaka K, Kawaguchi T, Imada Y, Ooi T, Arai M. Purification and properties of cellobiose phosphorylase from Clostridium thermocellum[J]. Journal of Fermentation and Bioengineering, 1995, 79(3): 212-216. DOI:10.1016/0922-338X(95)90605-Y |
| [9] |
Mieyal JJ, Abeles RH. 17 disaccharide phosphorylases[J]. The Enzymes, 1972, 7: 515-532. |
| [10] |
Schimz KL, Broll B, John B. Cellobiose phosphorylase (EC 2.4.1.20) of Cellulomonas: occurrence, induction, and its role in cellobiose metabolism[J]. Archives of Microbiology, 1983, 135(4): 241-249. DOI:10.1007/BF00413475 |
| [11] |
Hüttermann A, Volger C. Cellobiose phosphorylase in Fomes annosus[J]. Nature New Biology, 1973, 245(141): 64. DOI:10.1038/newbio245064a0 |
| [12] |
Yernool DA, McCarthy JK, Eveleigh DE, Bok JD. Cloning and characterization of the glucooligosaccharide catabolic pathway β-glucan glucohydrolase and cellobiose phosphorylase in the marine hyperthermophile Thermotoga neapolitana[J]. Journal of Bacteriology, 2000, 182(18): 5172-5179. DOI:10.1128/JB.182.18.5172-5179.2000 |
| [13] |
Nidetzky B, Eis C, Albert M. Role of non-covalent enzyme-substrate interactions in the reaction catalysed by cellobiose phosphorylase from Cellulomonas uda[J]. Biochemical Journal, 2000, 351(3): 649-659. DOI:10.1042/bj3510649 |
| [14] |
Rajashekhara E, Kitaoka M, Kim YK, Hayashi K. Characterization of a cellobiose phosphorylase from a hyperthermophilic eubacterium, Thermotoga maritima MSB8[J]. Bioscience, Biotechnology, and Biochemistry, 2002, 66(12): 2578-2586. DOI:10.1271/bbb.66.2578 |
| [15] |
Hamura K, Saburi W, Abe S, Morimoto N, Taguchi H, Mori H, Matsui H. Enzymatic characteristics of cellobiose phosphorylase from Ruminococcus albus NE1 and kinetic mechanism of unusual substrate inhibition in reverse phosphorolysis[J]. Bioscience, Biotechnology, and Biochemistry, 2012, 76(4): 812-818. DOI:10.1271/bbb.110954 |
| [16] |
Ayers WA. Phosphorolysis and synthesis of cellobiose by cell extracts from Ruminococcus flavefaciens[J]. Journal of Biological Chemistry, 1959, 234(11): 2819-2822. DOI:10.1016/S0021-9258(18)69677-2 |
| [17] |
Fan HY. Discovery of novel cellobiose phosphorylase and study on enzymology properties[D]. Zhengzhou: Master's Thesis of Henan Agricultural University, 2015 (in Chinese) 樊海妍. 新型纤维二糖磷酸化酶的发现及酶学性质研究[D]. 郑州: 河南农业大学硕士学位论文, 2015 |
| [18] |
Sheth K, Alexander JK. Purification and properties of beta- 1, 4-oligoglucan: orthophosphate glucosyltransferase from Clostridium thermocellum[J]. Journal of Biological Chemistry, 1969, 244(2): 457-464. DOI:10.1016/S0021-9258(18)94451-0 |
| [19] |
Kitaoka M, Taniguchi H, Hayashi K. Characterization of cellobiose phosphorylase and cellodextrin phosphorylase[J]. Journal of Applied Glycoscience, 2002, 49(2): 221-227. DOI:10.5458/jag.49.221 |
| [20] |
Taylor LE, Henrissat B, Coutinho PM, Ekborg NA, Hutcheson SW, Weiner RM. Complete cellulase system in the marine bacterium Saccharophagus degradans strain 2-40T[J]. Journal of Bacteriology, 2006, 188(11): 3849-3861. DOI:10.1128/JB.01348-05 |
| [21] |
Sawano T, Saburi W, Hamura K, Matsui H, Mori H. Characterization of Ruminococcus albus cellodextrin phosphorylase and identification of a key phenylalanine residue for acceptor specificity and affinity to the phosphate group[J]. FEBS Journal, 2013, 280(18): 4463-4473. DOI:10.1111/febs.12408 |
| [22] |
Zhong C, Luley-Goedl C, Nidetzky B. Product solubility control in cellooligosaccharide production by coupled cellobiose and cellodextrin phosphorylase[J]. Biotechnology and Bioengineering, 2019, 116(9): 2146-2155. DOI:10.1002/bit.27008 |
| [23] |
Zhang YHP, Lynd LR. Kinetics and relative importance of phosphorolytic and hydrolytic cleavage of cellodextrins and cellobiose in cell extracts of Clostridium thermocellum[J]. Applied and Environmental Microbiology, 2004, 70(3): 1563-1569. DOI:10.1128/AEM.70.3.1563-1569.2004 |
| [24] |
Lou JR, Dawson KA, Strobel HJ. Cellobiose and cellodextrin metabolism by the ruminal bacterium Ruminococcus albus[J]. Current Microbiology, 1997, 35(4): 221-227. DOI:10.1007/s002849900242 |
| [25] |
Kitaoka M, Sasaki T, Taniguchi H. Synthetic reaction of Cellvibrio gilvus cellobiose phosphorylase[J]. Journal of Biochemistry, 1992, 112(1): 40-44. DOI:10.1093/oxfordjournals.jbchem.a123862 |
| [26] |
Hidaka M, Kitaoka M, Hayashi K, Wakagi T, Shoun H, Fushinobu S. Structural dissection of the reaction mechanism of cellobiose phosphorylase[J]. Biochemical Journal, 2006, 398(1): 37-43. DOI:10.1042/BJ20060274 |
| [27] |
Sasaki T, Tanaka T, Nakagawa S, Kainuma K. Purification and properties of Cellvibrio gilvus cellobiose phosphorylase[J]. Biochemical Journal, 1983, 209(3): 803-807. DOI:10.1042/bj2090803 |
| [28] |
Sasaki T. Cellobiose phosphorylase from Cellvibrio gilvus[J]. Methods in Enzymology, 1988, 160: 468-472. |
| [29] |
Tanaka K, Kawaguchi T, Imada Y, Ooi T, Arai M. Purification and properties of cellobiose phosphorylase from Clostridium thermocellum[J]. Journal of Fermentation and Bioengineering, 1995, 79(3): 212-216. DOI:10.1016/0922-338X(95)90605-Y |
| [30] |
Chomvong K, Kordić V, Li X, Bauer S, Gillespie AE, Ha SJ, Oh E, Galazka JM, Jin YS, Cate JHD. Overcoming inefficient cellobiose fermentation by cellobiose phosphorylase in the presence of xylose[J]. Biotechnology for Biofuels, 2014, 7(1): 1-11. DOI:10.1186/1754-6834-7-1 |
| [31] |
Arai M, Tanaka K, Kawaguchi T. Purification and properties of cellodextrin phosphorylase from Clostridium thermocellum[J]. Journal of Fermentation & Bioengineering, 1994, 77(3): 239-242. |
| [32] |
Krishnareddy M, Kim YK, Kitaoka M, Mori Y, Hayashi K. Cellodextrin phosphorylase from Clostridium thermocellum YM4 strain expressed in Escherichia coli[J]. Journal of Applied Glycoscience, 2002, 49(1): 1-8. DOI:10.5458/jag.49.1 |
| [33] |
Wu YY, Mao GT, Fan HY, Song AD, Zhang YH, Chen HG. Biochemical properties of GH94 cellodextrin phosphorylase THA_1941 from a thermophilic Eubacterium Thermosipho africanus TCF52B with cellobiose phosphorylase activity[J]. Scientific Reports, 2017, 7: 4849. DOI:10.1038/s41598-017-05289-x |
| [34] |
Kuhaudomlarp S, Pergolizzi G, Patron NJ, Henrissat B, Field RA. Unraveling the subtleties of β-(1→3)-glucan phosphorylase specificity in the GH94, GH149, and GH161 glycoside hydrolase families[J]. Journal of Biological Chemistry, 2019, 294(16): 6483-6493. DOI:10.1074/jbc.RA119.007712 |
| [35] |
Kitaoka M, Sasaki T, Taniguchi H. Phosphorolytic reaction of Cellvibrio gilvus cellobiose phosphorylase[J]. Bioscience, Biotechnology, and Biochemistry, 1992, 56(4): 652-655. DOI:10.1271/bbb.56.652 |
| [36] |
Kim YK, Kitaoka M, Krishnareddy M, Mori Y, Hayashi K. Kinetic studies of a recombinant cellobiose phosphorylase (CBP) of the Clostridium thermocellum YM4 strain expressed in Escherichia coli[J]. Journal of Biochemistry, 2002, 132(2): 197-203. DOI:10.1093/oxfordjournals.jbchem.a003210 |
| [37] |
Bianchetti CM, Elsen NL, Fox BG, Phillips GN. Structure of cellobiose phosphorylase from Clostridium thermocellum in complex with phosphate[J]. Acta Crystallographica Section F Structural Biology and Crystallization Communications, 2011, 67(11): 1345-1349. DOI:10.1107/S1744309111032660 |
| [38] |
Hidaka M, Kitaoka M, Hayashi K, Wakagi T, Shoun H, Fushinobu S. Crystallization and preliminary X-ray analysis of cellobiose phosphorylase from Cellvibrio gilvus[J]. Acta Crystallographica Section D Biological Crystallography, 2004, 60(10): 1877-1878. DOI:10.1107/S0907444904017767 |
| [39] |
Van Hoorebeke A, Stout J, Kyndt J, De Groeve M, Dix I, Desmet T, Soetaert W, Van Beeumen J, Savvides SN. Crystallization and X-ray diffraction studies of cellobiose phosphorylase from Cellulomonas uda[J]. Acta Crystallographica Section F Structural Biology and Crystallization Communications, 2010, 66(3): 346-351. DOI:10.1107/S1744309110002642 |
| [40] |
O'Neill EC, Pergolizzi G, Stevenson CEM, Lawson DM, Nepogodiev SA, Field RA. Cellodextrin phosphorylase from Ruminiclostridium thermocellum: X-ray crystal structure and substrate specificity analysis[J]. Carbohydrate Research, 2017, 451: 118-132. DOI:10.1016/j.carres.2017.07.005 |
| [41] |
Mao GT, Wang YY, Li B, Liu Q, Zhang HS, Xie H, Chen HG, Song AD. Analysis of crystallization and X-ray crystallographic of the cellodextrin phosphorylase from Thermosipho africanus[J]. Journal of Henan Agricultural University, 2019, 53(3): 400-405. (in Chinese) 毛国涛, 王园园, 李斌, 刘茜, 张宏森, 谢慧, 陈红歌, 宋安东. 非洲栖热腔菌(Thermosipho africanus)纤维寡糖磷酸化酶的结晶及衍射分析[J]. 河南农业大学学报, 2019, 53(3): 400-405. |
| [42] |
Chauve M, Mathis H, Huc D, Casanave D, Monot F, Ferreira N. Comparative kinetic analysis of two fungal β-glucosidases[J]. Biotechnology for Biofuels, 2010, 3(1): 3. DOI:10.1186/1754-6834-3-3 |
| [43] |
Sekar R, Shin HD, Chen R. Engineering Escherichia coli cells for cellobiose assimilation through a phosphorolytic mechanism[J]. Applied and Environmental Microbiology, 2012, 78(5): 1611-1614. DOI:10.1128/AEM.06693-11 |
| [44] |
Rutter C, Chen R. Improved cellobiose utilization in E. coli by including both hydrolysis and phosphorolysis mechanisms[J]. Biotechnology Letters, 2014, 36(2): 301-307. DOI:10.1007/s10529-013-1355-7 |
| [45] |
Zhang F, Rodriguez S, Keasling JD. Metabolic engineering of microbial pathways for advanced biofuels production[J]. Current Opinion in Biotechnology, 2011, 22(6): 775-783. DOI:10.1016/j.copbio.2011.04.024 |
| [46] |
Machida M, Ohtsuki I, Fukui S, Yamashita I. Nucleotide sequences of Saccharomycopsis fibuligera genes for extracellular beta-glucosidases as expressed in Saccharomyces cerevisiae[J]. Applied and Environmental Microbiology, 1988, 54(12): 3147-3155. DOI:10.1128/aem.54.12.3147-3155.1988 |
| [47] |
Lee WH, Nan H, Kim HJ, Jin YS. Simultaneous saccharification and fermentation by engineered Saccharomyces cerevisiae without supplementing extracellular β-glucosidase[J]. Journal of Biotechnology, 2013, 167(3): 316-322. DOI:10.1016/j.jbiotec.2013.06.016 |
| [48] |
Sadie CJ, Rose SH, Haan RD, Van Zyl WH. Co-expression of a cellobiose phosphorylase and lactose permease enables intracellular cellobiose utilisation by Saccharomyces cerevisiae[J]. Applied Microbiology and Biotechnology, 2011, 90(4): 1373-1380. DOI:10.1007/s00253-011-3164-z |
| [49] |
Ha SJ, Galazka JM, Joong Oh E, Kordić V, Kim H, Jin YS, Cate JHD. Energetic benefits and rapid cellobiose fermentation by Saccharomyces cerevisiae expressing cellobiose phosphorylase and mutant cellodextrin transporters[J]. Metabolic Engineering, 2013, 15: 134-143. DOI:10.1016/j.ymben.2012.11.005 |
| [50] |
You C, Chen H, Myung S, Sathitsuksanoh N, Ma H, Zhang XZ, Li J, Zhang YHP. Enzymatic transformation of nonfood biomass to starch[J]. Proceeding of the National Academy of the Sciences of United States of America, 2013, 110(18): 7182-7187. DOI:10.1073/pnas.1302420110 |
| [51] |
Schwaiger KN, Voit A, Dobiašová H, Luley C, Wiltschi B, Nidetzky B. Plasmid design for tunable two-enzyme co-expression promotes whole-cell production of cellobiose[J]. Biotechnology Journal, 2020, 15(11): e2000063. DOI:10.1002/biot.202000063 |
| [52] |
Ubiparip Z, Moreno DS, Beerens K, Desmet T. Engineering of cellobiose phosphorylase for the defined synthesis of cellotriose[J]. Applied Microbiology and Biotechnology, 2020, 104(19): 8327-8337. DOI:10.1007/s00253-020-10820-8 |
| [53] |
Kino K, Satake R, Morimatsu T, Kuratsu S, Shimizu Y, Sato M, Kirimura K. A new method of synthesis of alkyl β-glycosides using sucrose as sugar donor[J]. Bioscience, Biotechnology, and Biochemistry, 2008, 72(9): 2415-2417. DOI:10.1271/bbb.80097 |
| [54] |
De Winter K, Van Renterghem L, Wuyts K, Pelantová H, Křen V, Soetaert W, Desmet T. Chemoenzymatic synthesis of β-D-glucosides using cellobiose phosphorylase from Clostridium thermocellum[J]. Advanced Synthesis & Catalysis, 2015, 357(8): 1961-1969. |
| [55] |
Alexander JK. Synthesis of 4-o-β-d-glucopyranosyl- d-xylose, 4-o-β-d-glucopyranosyl-d-arabinose, 4-o-β-d- glucopyranosyl-2-deoxy-d-glucose, 4-o-β-d-glucopyranosyl- d-mannose, and 4-o-β-d-glucopyranosyl-d-glucosamine by cellobiose phosphorylase from Clostridium thermocellum[J]. Archives of Biochemistry and Biophysics, 1968, 123(2): 240-246. DOI:10.1016/0003-9861(68)90130-6 |
| [56] |
Percy A, Ono H, Hayashi K. Acceptor specificity of cellobiose phosphorylase from Cellvibrio gilvus: synthesis of three branched trisaccharides[J]. Carbohydrate Research, 1998, 308(3/4): 423-429. |
| [57] |
Nakai H, Hachem MA, Petersen BO, Westphal Y, Mannerstedt K, Baumann MJ, Dilokpimol A, Schols HA, Duus JØ, Svensson B. Efficient chemoenzymatic oligosaccharide synthesis by reverse phosphorolysis using cellobiose phosphorylase and cellodextrin phosphorylase from Clostridium thermocellum[J]. Biochimie, 2010, 92(12): 1818-1826. DOI:10.1016/j.biochi.2010.07.013 |
 2021, Vol. 48
2021, Vol. 48




