
扩展功能
文章信息
- 程彤, 薛亮, 吴清平, 张菊梅
- CHENG Tong, XUE Liang, WU Qingping, ZHANG Jumei
- 诺如病毒反向遗传系统的研究进展与应用
- Research progress and application of norovirus reverse genetics systems
- 微生物学通报, 2021, 48(3): 974-983
- Microbiology China, 2021, 48(3): 974-983
- DOI: 10.13344/j.microbiol.china.201086
-
文章历史
- 收稿日期: 2020-11-18
- 接受日期: 2021-01-20
- 网络首发日期: 2021-01-28
2. 广东省科学院广东省微生物研究所 华南应用微生物国家重点实验室 广东省微生物安全与健康重点实验室 广东 广州 510070
2. State Key Laboratory of Applied Microbiology Southern China, Guangdong Institute of Microbiology, Guangdong Academy of Sciences, Key Laboratory of Microbial Safety and Health of Guangdong Province, Guangzhou, Guangdong 510070, China
诺如病毒(Norovirus,NoV)是导致人类急性胃肠炎的主要病原体,具有传播迅速、易变异等特点。全球每年超过20%的食源性疾病由NoV引起,感染人次高达6.84亿,导致20多万人死亡,是当前普遍关注的公共卫生安全问题[1-2]。目前尚未批准有效疫苗或抗病毒药物用于预防或治疗NoV感染。我国是NoV流行的主要地区之一,近年来,我国NoV的发病率呈逐年上升趋势。本课题组通过对贝类、地表水、临床样本等的长期监测,证实了NoV是我国广东地区冬季散发性胃肠炎的主要致病原[3-5]。长期以来,对NoV的研究主要受限于标准的体外细胞培养系统的缺乏;此外,由于受体识别等因素带来的物种特异性,导致人源NoV缺乏合适的小动物感染模型,严重限制了对其感染、复制机理、致病机制及病毒-宿主相互作用等的研究,并阻碍了其有效疫苗和抗病毒药物的研发。过去几十年里,研究人员尝试利用人源原代免疫细胞和永生化细胞在体外培养人源NoV,但均未成功。2016年,NoV的体外培养取得突破性进展,Ettayebi等首次在干细胞衍生的非转化人类肠道单层细胞培养系统中成功培养出了多种人源NoV毒株[6],使NoV体外研究成为可能。然而,由于该系统构建过程的复杂性,目前还未得到广泛应用。虽然NoV已被证明可在黑猩猩、猪和牛等大型动物体内复制,然而这些动物模型在临床症状、肠道生理以及对NoV的敏感性方面都与人类存在明显差异,加之伦理学等方面的原因,因此在应用上仍存在一定的局限性。近年来,研究人员相继构建的新型NoV反向遗传操作体系为系统研究该病原提供了有力工具,加深了对其复制周期、致病机制及病毒-宿主相互作用等的了解。基于反向遗传学操作技术,目前已经能成功拯救出具有感染性的NoV,实现了人源NoV的体外增殖。本文主要综述了反向遗传学技术用于NoV研究的工作进展(表 1),并讨论了该技术在NoV疫苗制备和抗病毒药物开发中的应用及发展前景,以期为NoV防控策略的制定及药物靶点的挖掘提供有益参考,推进我国食品安全风险防控工作。
| Virus strain | Method | Promoter | Helper virus | Transfected cells | Viral yield | References |
| GI.1 NoV | Plasmid transfection | T7 | MVA | HEK293T | 8.5×104 virion from 12 T-75 flasks | [7] |
| GII.3 U201 NoV | Plasmid transfection | T7 | MVA | HEK293T | 1.32 g/cm3 | [8] |
| GI.1 NoV | In vitro transcription | T7 | / | Huh7 and BHK21 | 2.6×1011 g.e./µg-RNA and 8.0×108 g.e./µg-RNA | [9] |
| MNV-1 | Plasmid transfection | T7 | FPV | BHK21 | 5×104 TCID50 per 35 mm dish | [10] |
| MNV | Plasmid transfection | pol Ⅱ | / | HepG2 | 103 pfu/ml | [11] |
| MNV-1 | In vitro transcription | T7 | / | Raw 264.7 | 4.5×105 TCID50 per 35 mm dish | [12] |
| MNV-3 | Plasmid transfection | T7 | FPV | BSR-T7 | 6.3×103 TCID50/ml | [13] |
| MNV | In vitro transcription | T7 | / | BSR-T7 | 1×106 TCID50/ml | [14] |
| MNV | Plasmid transfection | T7 | FPV | BSR-T7 | 1×104 TCID50/ml | [14] |
| GII.3 U201 NoV | Plasmid transfection | EF-1 | / | COS7, HEK293T, Huh7, Caco-2 | 8.0×104, 1.4×104, 2.4×102 and 1.3×101 copies per 106 cells, respectively | [15] |
| MNV-1 CW1 | Plasmid transfection | T7 | / | BSR-T7 | 2.6×103 pfu/ml | [16] |
| GII.4 Sydeny NoV | Plasmid transfection | CMV | / | Caco-2 | Not reported | [17] |
| Note: HEK293T: Human embryonic kidney 293T cells; Huh7: Human hepatocellular carcinoma cell line; BHK21: Baby hamster kidney fibroblast cells; HepG2: Human hepatocellular carcinoma cell line G2 cells; Raw 264.7: Mouse leukemia cells of monocyte macrophage; BSR-T7: Baby hamster kidney cells expressing T7 RNA polymerase; COS7: African green monkey kidney cell; Caco-2: Human colorectal adenocarcinoma cells; MVA: Modified vaccinia ankara; FPV: Fowlpox virus; pfu/ml: Plaque forming units/mL; g.e.: Genome equivalents; /: The system does not require the presence of the helper viruses | ||||||
反向遗传学技术能实现在DNA水平上对RNA病毒基因组的体外操作,可从基因水平上探索病毒毒力,是研究RNA病毒基因表达调控机制、进化机理、特定蛋白结构与功能、致病机制等的重要方法,在抗病毒药物筛选和疫苗制备等领域具有十分广泛的用途(图 1)。目前,利用反向遗传学技术已成功拯救出多种动物RNA病毒,其构建策略包括病毒全长cDNA克隆载体的构建、体外转录、细胞转染及拯救病毒的表型鉴定等。现有的NoV反向遗传系统主要可分为T7 RNA聚合酶体内转录系统、T7 RNA聚合酶体外转录系统、稳定表达T7 RNA聚合酶的哺乳动物细胞系体内转录系统及真核细胞启动子体内转录系统。
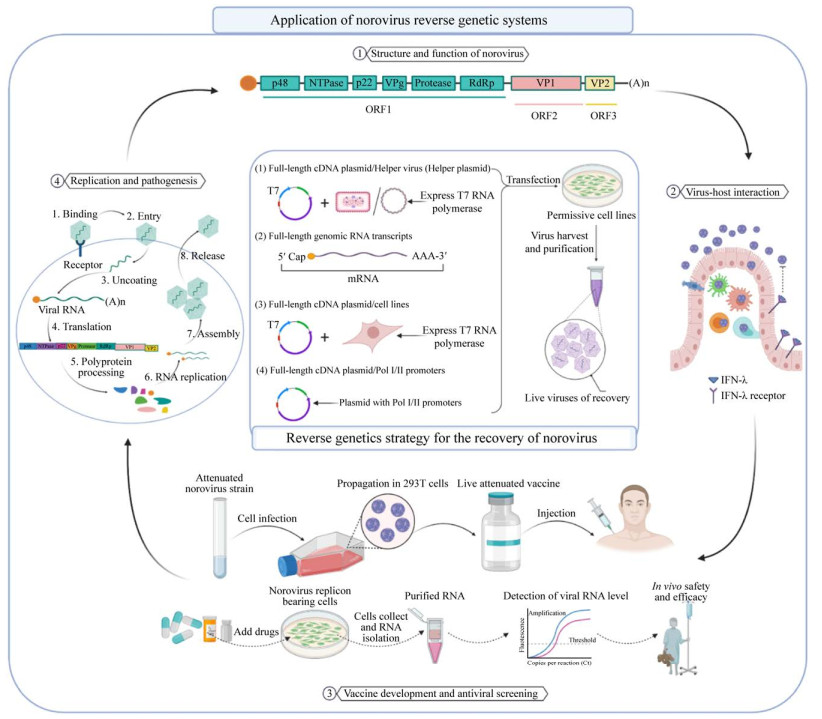
|
| 图 1 诺如病毒反向遗传学系统的应用 Figure 1 Application of norovirus reverse genetic systems (created with BioRender.com) |
|
|
体内转录是指直接将含有全长或分段病毒cDNA序列的质粒转染至合适细胞系,使其在胞内直接转录复制,进而装配出感染性的子代病毒[18]。T7 RNA聚合酶体内转录系统即通过辅助质粒或辅助病毒(如牛痘病毒、禽痘病毒)表达的T7 RNA聚合酶在细胞中产生具有感染性的子代病毒。2005年,Asanaka等利用该方法首次实现了对GI.1型人源NoV的体外拯救[7],从12个T-75培养瓶中共回收到了约8.5×104个病毒颗粒。此后,采用相同的方法实现了GII.3型人源NoV的拯救[8],然而用该方法产生的病毒密度低于临床样本中的病毒密度,并且牛痘病毒的使用导致了细胞病变,因此不适用于病毒拯救。在后来的研究中,Chaudhry等发现牛痘病毒也会抑制鼠诺如病毒(Murine Norovirus,MNV)的复制,相比之下,基于T7 RNA聚合酶的禽痘病毒系统可成功用于正链、负链及双链RNA病毒的拯救;2007年,Chaudhry等将MNV的全长cDNA转染到预先用禽痘病毒(FPV-T7)感染过的细胞中,第一次在体外拯救出了MNV,并且发现FPV-T7的感染对MNV的复制无有害影响[10],该系统随后被用于分析NoV生命周期的许多方面。Bailey等利用该系统构建了MNV减毒毒株的感染性cDNA克隆突变体,对拯救病毒的序列分析发现MNV衣壳蛋白第296位谷氨酸到赖氨酸的替换足以恢复病毒毒力,首次鉴定出了影响MNV毒力的病毒编码因子[19]。Simmonds等将该系统与生物信息学相结合,发现MNV亚基因组启动子区和3′端RNA结构的破坏会导致MNV复制能力的完全丧失,而对RNA二级结构进行修复后病毒感染性可得到恢复,证明了RNA二级结构在病毒复制中发挥重要的生物学功能[20]。
依赖辅助病毒的体内病毒拯救法效率不高,因为拯救出的病毒还需经过复杂耗时的步骤才能从辅助病毒中分离出来,而且辅助病毒导致的宿主细胞病变还会影响拯救病毒的增殖,从而限制了该系统的应用。此外,感染性克隆的构建复杂且困难,需要考虑多种因素,如质粒拷贝数、宿主菌的选择、培养温度、插入物的大小和启动子选择等。因此,在后来的研究中,该系统应用较少。
1.2 T7 RNA聚合酶体外转录系统T7 RNA聚合酶体外转录系统是构建由T7启动子控制的病毒全长cDNA克隆,并在全长cDNA 3′端引入限制性酶切位点用于体外转录,以获得病毒RNA[21]。由于正链RNA病毒基因组可直接作为mRNA翻译病毒蛋白,因此只需将RNA转录本直接转染敏感细胞即可自行组装出感染性的病毒粒子。构建该系统时,获得高保真的病毒全长cDNA至关重要。此外,由于天然NoV基因组5′端存在帽状结构,该结构对病毒的感染性具有重要作用,而体外转录所得RNA 5′端无帽状结构,因此,通过这种策略产生的RNA还需要在体外转录体系中添加帽子类似物,以使病毒RNA获得感染性。1995年,Sosnovtsev等利用该方法在猫肾细胞中拯救出了感染性的猫杯状病毒[22],证明了帽状类似物的添加可替代VPg蛋白建立感染,这一发现加速了杯状病毒反向遗传学系统的发展。2010年,Yunus等在对体外加帽过程及鼠巨噬细胞系(Raw 264.7)的电转条件进行优化后,建立了一种高效拯救MNV的方法,每6×106个细胞的病毒产量可达109 TCID50[12]。2012年,Arias等利用该系统也成功拯救出了感染性MNV[14]。
T7体外转录系统的应用为体外获得感染性的病毒粒子提供了有力工具,可用于研究突变对病毒复制及宿主致病能力的影响,具有十分重要的意义。然而,由于体外加帽效率较低的原因,部分RNA转录本会缺少5′帽子结构或出现帽状类似物与RNA错误结合的情况,从而导致RNA翻译效率低下,难以获得子代病毒。此外,未加帽的RNA转录本还会在细胞内诱导干扰素产生,这也会对病毒产量产生重大影响。总体而言,该系统的病毒拯救效率较低、耗时耗力,需要昂贵的体外转录试剂及帽状类似物,所得RNA转录本的感染性较亲本毒株弱,操作过程中极易受到污染而降解,因此感染性病毒的拯救不易成功。
1.3 稳定表达T7 RNA聚合酶的哺乳动物细胞系体内转录系统通过构建稳定表达T7 RNA聚合酶的哺乳动物细胞系,在细胞内直接驱动感染性cDNA克隆的转录及复制,也可以得到感染性的子代病毒。Arias等于2012年通过质粒转染的方法,发现MNV RNA可直接在表达T7 RNA聚合酶的细胞中产生[14]。2015年,Sandoval-Jaime等将包含MNV全长的cDNA质粒转染到表达T7 RNA聚合酶的细胞中,也获得了感染性MNV[16]。
该系统的应用可以使T7 RNA聚合酶基因在不同靶细胞内高水平表达,能有效促进大量RNA在体内转录且能表达较大的外源基因,使病毒拯救更加便利、有效。由于病毒基因组直接在细胞内转录,可避免RNA降解,保证了RNA转录本的正确性,并且获得的RNA更加稳定、感染性较强。但前提是要获得可稳定表达T7 RNA聚合酶的动物靶细胞系,而该细胞系的构建过程比较耗时,需要经过复杂的构建、筛选及检验过程。稳定表达T7 RNA聚合酶靶细胞系的获得将大大提高病毒拯救效率,只需将质粒转染至合适细胞系,即可实现一步法快速拯救。因此,目前不同RNA病毒的拯救大多采用此方法,表现了其广泛的应用前景。
1.4 真核细胞启动子体内转录系统真核细胞中广泛分布着RNA聚合酶Ⅰ、Ⅱ,在构建RNA病毒全长cDNA克隆时,可在载体中引入真核细胞特异识别的启动子替代T7启动子,直接驱动病毒基因组在胞内转录。通过这种策略构建的病毒cDNA不再需要体外转录,可直接导入宿主细胞;在体内产生的病毒转录本可充当信使RNA,利用宿主细胞的翻译系统表达病毒蛋白[23]。虽然真核细胞RNA聚合酶启动外源基因转录的功能不如T7 RNA聚合酶强大,但其具有校正功能、广泛的细胞适应性、可避免体外长时间操作RNA等优点。2007年,Ward等开发了一种RNA聚合酶Ⅰ启动子驱动的MNV反向遗传学系统[11],然而用该系统得到的病毒产量不足FPV-T7系统的十分之一。2014年,Katayama等成功构建了由EF-1α启动子驱动的人源NoV GII.3反向遗传学系统,该系统随后被证明也适用于其他人源NoV及MNV的拯救,表明了其较高的稳定性及通用性[15]。2018年,Oliveira等构建了在巨细胞病毒(CMV)启动子控制下的人源NoV GII.4悉尼亚型的感染性cDNA克隆和含GFP报告基因的复制子系统,转染人结肠癌(Caco-2)细胞后病毒结构蛋白得到了表达;然而,研究人员发现GFP基因的插入通过一种未知机制对病毒结构蛋白的合成产生了负面影响[17]。
尽管真核聚合酶拯救系统操作简单,但其转录能力有限,转录本存在被剪切的风险,病毒拯救效率不高,因此没能广泛应用于各类RNA病毒的拯救,但其在基因组较小的正链RNA病毒拯救中应用较多。与从粪便中或MNV反向遗传学系统中回收的病毒相比,现有的人源NoV反向遗传学系统回收病毒的效率均较低。此外,虽然已证实NoV可在人类来源的B细胞系及干细胞衍生的非转化人类肠道单层细胞培养系统中进行复制,然而因其仅支持少量病毒株的复制且培养条件苛刻等,较难广泛用于NoV的连续传代,仍然存在一定的局限性,目前未能得到广泛应用。因此,仍需开发稳健的NoV体外细胞培养系统,以充分配合反向遗传学系统,阐明NoV的相关生物学问题并促进其有效疗法的开发。
2 诺如病毒反向遗传学系统的应用 2.1 病毒基因组结构与功能研究NoV反向遗传系统是研究病毒基因组结构与功能的有力工具,可用于确定病毒蛋白功能、新型开放阅读框、转录调控序列,病毒的毒力及致病性等。Sandoval-Jaime等发现NoV基因组5′端结构的完整性对病毒拯救具有重要影响,缺失5′非编码区的载体无法拯救出病毒粒子[16]。Robinson等利用反向遗传学系统证明了MNV持久株CR6非结构蛋白NS1/2的裂解对MNV持久感染肠上皮细胞至关重要,而且NS1/2的裂解可促进细胞凋亡[24]。此外,MNV的VPg蛋白被证明可通过与宿主翻译因子相互作用来介导病毒基因组的翻译[25-26]。长期以来,NoV亚基因组RNA合成的机制一直未被深入阐明,Yunus等通过构建MNV的感染性克隆,发现MNV负链RNA上存在的保守茎环结构对MNV的复制至关重要,茎环序列区的氨基酸替换会导致其复制能力的显著下降,进一步的体外实验表明该茎环结构可作为MNV的亚基因启动子起始病毒RNA的合成[27]。McFadden等[28]发现,与人源NoV相比,MNV亚基因组还编码额外的开放阅读框4,其翻译后产生的蛋白主要定位于线粒体并且可拮抗先天免疫应答,是MNV毒力和感染力的重要决定因子。Yen等[29]对GII.4型人源NoV非结构蛋白NTPase的生物学功能进行了系统表征,发现其N端的179个氨基酸序列在病毒复制和致病过程中发挥多种作用,如介导囊泡形成、内质网共定位及诱导细胞凋亡等。前期研究中,本课题组开发了一种细菌表面展示系统,基于该系统发现NoV P蛋白可以特异性识别并结合组织血型抗原(Histoblood Group Antigen,HBGA),体现了该系统在捕获并分离人源NoV受体中的广泛应用前景[30]。
2.2 病毒宿主相互作用研究了解病毒宿主间的相互作用有利于了解病毒的致病机制,可为疫苗开发及抗病毒药物研发提供一定思路。病毒通过劫持宿主防御系统及调节细胞内环境的稳态,从而在宿主细胞内进行基因组复制、增殖并导致机体致病。相应地,细胞也通过各种防御机制来检测、摧毁及清除病毒,如通过调节细胞内信号通路来抵抗病毒感染。Chang等利用人源NoV复制子系统证明了外源干扰素IFN-α可有效抑制NoV的体外复制[9]。Peterson等[31]研究发现Ⅲ型干扰素基因Ifnl2和Ifnl3的破坏可间接导致Ⅲ型干扰素受体受损,从而阻止干扰素信号表达。Mears等[32]研究了Ifit1蛋白在控制MNV感染中的作用,证明了Ifit1是MNV感染期间的一种抗病毒因子,其不能直接抑制病毒翻译,但Ifit1可通过增强鼠巨噬细胞中的干扰素反应来限制MNV的复制。Lin等[33]研究了NoV与宿主间的相互作用机制,研究结果显示在病毒感染人肠道细胞后,IFN-β的表达没有改变,IFN-λ、ISG-15及ISG-45的表达在感染后显著升高,这凸显了IFN-λ信号在NoV与宿主间相互作用中的重要作用。病毒与宿主细胞的相互作用对RNA病毒的复制至关重要。宿主细胞中存在对病毒感染及复制具有促进或抑制作用的宿主细胞因子,现已发现的影响NoV复制的宿主细胞因子主要包括组织血型抗原(HBGA)[34]、细胞蛋白La、多聚嘧啶区结合蛋白(PTB)及polyA结合蛋白(PABP)[35]等。与MNV复制相关的宿主因子包括唾液酸、糖脂、糖蛋白[36]、DDX3、La、PTB[37-38]、polyC结合蛋白(PCBP)、异质核糖核蛋白A1 (hnRNPA1)[39]、信号转导和转录激活因子1 (STAT1)[40]、干扰素调节因子3 (IRF3)[41]等。此外,病毒对宿主细胞信号通路的破坏也可实现其在宿主细胞中的有效复制,基于人源NoV复制子系统,研究人员发现复制子细胞的胆固醇和碳水化合物的生物合成途径都有所改变[42]。Arthur等[43]利用NoV复制子系统研究了影响病毒复制的宿主因子,发现Ⅲ型干扰素在抑制NoV体外复制中起重要作用,而且干扰素λ受体IFNLR1的缺失会增强NoV的复制。
2.3 病毒致病机制研究以病毒的cDNA体外转录来获得拯救病毒是研究RNA病毒致病机制的有效手段。将病毒序列与反向遗传学技术结合可用于研究病毒致病性。2006年,Chang等建立的NoV复制子系统被广泛用于研究外源干扰素及药物对病毒复制的影响,研究发现NoV对IFNα、IFNγ、利巴韦林、霉酚酸等物质较为敏感[44],核苷类似物2C甲基胞苷处理可抑制病毒的体外复制[45]。Lee等[46]发现MNV的NS1蛋白的分泌对于MNV感染引起的肠道发病机制和抵抗内源性IFN至关重要。研究人员发现有些人群不易感染NoV,是因为他们体内缺乏一种功能性岩藻糖基转移酶,该酶的缺乏导致机体不能正确表达岩藻糖基聚糖及HBGA受体[47]。CD300受体家族被证明参与了许多疾病的致病过程,并参与病毒逃逸机制。此外,病毒的细胞趋向性也是决定病毒致病机制的重要因素,MNV感染簇状细胞,但不感染其他肠上皮细胞类型,这是因为簇状细胞特异性表达了MNV受体CD300lf[48]。NoV的附着和侵袭与HBGA有关,Zhang等[49]从生菜中分离到了一种可特异性捕获NoV的HBGA类物质,发现其作为NoV的关键配体在病毒感染中发挥重要作用。
2.4 抗病毒药物筛选复制子系统是重要的病毒反向遗传学研究手段,能实现病毒的体外高效复制,为一些难培养病毒的研究提供有效的解决方案。例如,HCV的研究工作同样曾长期受制于缺乏合适的体外细胞培养体系,但Lohmann等[50]在1999年提出的亚基因组复制子系统,解决了HCV病毒难以体外复制的难题,被公认是HCV研究的里程碑事件,为HCV致病机制及抗病毒药物研究提供了重要条件。Chang等替换人源NoV病毒复制子系统中的衣壳蛋白建立的稳转细胞系,已广泛应用于抗病毒药物筛选工作[9],基于该方法证明了胆固醇水平在NoV复制中的重要作用[42]。
NoV复制子系统已广泛应用于抗病毒化合物的评价并加速了抗病毒感染药物的研究进程。蛋白酶已被证明是抗NoV病毒的有效靶点,Kitano等[51]利用MNV反向遗传学系统研究了病毒蛋白酶抑制剂Rupintrivir对病毒复制的影响,研究发现,病毒蛋白酶中单一I109V替代的重组MNV株对Rupintrivir的敏感性降低,从而证明了NoV蛋白酶的突变可导致耐药突变体的产生。Chang等[52]成功构建了NoV 3C样蛋白酶的抑制剂,利用NoV复制子评估了其抗病毒活性,结果表明基因组P1区谷氨酰胺替代物的加入对抑制剂抑制病毒复制至关重要。Van Dycke等证明了7-去氮-2′-碳-甲基腺苷可直接作用于NoV的RdRp而抑制其体外复制[45]。
2.5 疫苗研制NoV极易发生变异,目前尚未批准有效疫苗或抗病毒药物用于预防或治疗NoV感染,几乎所有的NoV候选疫苗都是由病毒样颗粒(VLP)产生的亚单位疫苗,目前已有2种VLP疫苗进入临床试验阶段。利用反向遗传学技术,可直接对病毒基因组进行片段缺失、置换、定点突变等操作,通过获救病毒的表型变化推断相关基因操作的影响,可用于确定RNA病毒的毒力基因,促进减毒病毒或改进的可用疫苗的发展,也可用于开发嵌合疫苗或多价疫苗[53]。基于对病毒基因组感染性cDNA克隆的改造,以获救病毒作为载体表达外源基因可用于研制分子标记疫苗和嵌合体疫苗。Lee等在2019年发表的论文首次提出了MNV NS1疫苗,他们发现单独用NS1接种疫苗可有效地保护小鼠不受MNV感染,表明了其作为疫苗的潜在效果;然而由于NSI区域保守度较低,因此该区域可能面临巨大的进化压力,影响NSI作为疫苗抗原的稳定性[46]。
3 展望反向遗传学技术能实现在DNA水平上对RNA病毒基因组进行体外操作,在RNA病毒复制、感染、致病机制、追溯病毒起源、药物筛选及疫苗开发等方面有着十分广泛的用途。此外,反向遗传学技术还可用于构建靶向病毒载体及研发基因工程疫苗,在基因治疗及病毒致弱等方面具有巨大潜力。近年来,中外研究学者利用该技术对NoV开展了大量的研究工作,基于反向遗传学技术,目前已经能成功拯救出具有感染性的NoV,实现了NoV的体外增殖。然而,与从粪便中或MNV反向遗传学系统中回收的病毒相比,目前所有的人源NoV反向遗传系统的病毒回收效率均较低。由于缺乏适用于测试NoV候选疫苗的标准动物模型及细胞培养体系,迄今尚无批准的NoV有效疫苗及抗病毒药物。当前,NoV的研究仍然存在很多尚未解决的问题,如人源与动物源NoV复制的相关性、病毒感染细胞的详细过程、先天免疫及适应性免疫在抵抗NoV感染中的作用、病毒基因组翻译和复制所必需的细胞蛋白的功能及宿主细胞因子的作用等。与人源NoV结构蛋白VP1的深入研究相比,非结构蛋白在病毒复制周期、发病机制和宿主免疫反应中的作用还未被深入阐明。此外,在疫苗研制方面,目前可用的候选疫苗是否能够预防所有的异源NoV毒株及最常见的可引起疫情暴发的NoV突变株、疫苗是否对所有年龄组有效以及疫苗在免疫缺陷患者体内诱导的保护时间的长短等问题仍然急需解决。NoV反向遗传系统取代了传统的感染模型,为体外感染提供了一个平台,可实现NoV在体外培养细胞中的有效复制,在NoV致病机制研究和疫苗开发方面发挥着极大的作用。因此,未来仍需开发更加稳健的NoV新型反向遗传学系统,配合现有的NoV研究手段,以深入阐明NoV的相关生物学问题,推进NoV防控策略的制定,为设计安全有效的抗NoV药物和开发灭活及减毒NoV疫苗提供平台。
| [1] |
Lopman BA, Steele D, Kirkwood CD, Parashar UD. The vast and varied global burden of norovirus: Prospects for prevention and control[J]. PLoS Medicine, 2016, 13(4): e1001999. DOI:10.1371/journal.pmed.1001999 |
| [2] |
Kirk MD, Pires SM, Black RE, Caipo M, Crump JA, Devleesschauwer B, Döpfer D, Fazil A, Fischer-Walker CL, Hald T, et al. World Health Organization estimates of the global and regional disease burden of 22 foodborne bacterial, protozoal, and viral diseases, 2010: A data synthesis[J]. PLoS Medicine, 2015, 12(12): e1001921. DOI:10.1371/journal.pmed.1001921 |
| [3] |
Xue L, Dong RM, Wu QP, Li YL, Cai WC, Kou XX, Zhang JM, Guo WP. Molecular epidemiology of noroviruses associated with sporadic gastroenteritis in Guangzhou, China, 2013-2015[J]. Archives of Virology, 2016, 161(5): 1377-1384. DOI:10.1007/s00705-016-2784-0 |
| [4] |
Xue L, Wu QP, Dong RM, Kou XX, Li YL, Zhang JM, Guo WP. Genetic analysis of noroviruses associated with sporadic gastroenteritis during winter in Guangzhou, China[J]. Foodborne Pathogens and Disease, 2013, 10(10): 888-895. DOI:10.1089/fpd.2013.1521 |
| [5] |
Xue L, Cai WC, Gao JS, Zhang L, Dong RM, Li YL, Wu HM, Chen MT, Zhang JM, Wang J, et al. The resurgence of the norovirus GII.4 variant associated with sporadic gastroenteritis in the post-GII.17 period in South China, 2015 to 2017[J]. BMC Infectious Diseases, 2019, 19(1): 696. DOI:10.1186/s12879-019-4331-6 |
| [6] |
Ettayebi K, Crawford SE, Murakami K, Broughman JR, Karandikar U, Tenge VR, Neill FH, Blutt SE, Zeng XL, Qu L, et al. Replication of human noroviruses in stem cell-derived human enteroids[J]. Science, 2016, 353(6306): 1387-1393. DOI:10.1126/science.aaf5211 |
| [7] |
Asanaka M, Atmar RL, Ruvolo V, Crawford SE, Neill FH, Estes MK. Replication and packaging of Norwalk virus RNA in cultured mammalian cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(29): 10327-10332. DOI:10.1073/pnas.0408529102 |
| [8] |
Katayama K, Hansman GS, Oka T, Ogawa S, Takeda N. Investigation of norovirus replication in a human cell line[J]. Archives of Virology, 2006, 151(7): 1291-1308. DOI:10.1007/s00705-005-0720-9 |
| [9] |
Chang KO, Sosnovtsev SV, Belliot G, King AD, Green KY. Stable expression of a Norwalk virus RNA replicon in a human hepatoma cell line[J]. Virology, 2006, 353(2): 463-473. DOI:10.1016/j.virol.2006.06.006 |
| [10] |
Chaudhry Y, Skinner MA, Goodfellow IG. Recovery of genetically defined murine norovirus in tissue culture by using a fowlpox virus expressing T7 RNA polymerase[J]. The Journal of General Virology, 2007, 88(Pt 8): 2091-2100. |
| [11] |
Ward VK, Mccormick CJ, Clarke IN, Salim O, Wobus CE, Thackray LB, Virgin IV HW, Lambden PR. Recovery of infectious murine norovirus using pol Ⅱ-driven expression of full-length cDNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(26): 11050-11055. DOI:10.1073/pnas.0700336104 |
| [12] |
Yunus MA, Chung LMW, Chaudhry Y, Bailey D, Goodfellow I. Development of an optimized RNA-based murine norovirus reverse genetics system[J]. Journal of Virological Methods, 2010, 169(1): 112-118. DOI:10.1016/j.jviromet.2010.07.006 |
| [13] |
Arias A, Bailey D, Chaudhry Y, Goodfellow I. Development of a reverse-genetics system for murine norovirus 3: long-term persistence occurs in the caecum and colon[J]. The Journal of General Virology, 2012, 93(Pt 7): 1432-1441. |
| [14] |
Arias A, Ureña L, Thorne L, Yunus MA, Goodfellow I. Reverse genetics mediated recovery of infectious murine norovirus[J]. Journal of Visualized Experiments: JoVE, 2012(64): 4145. |
| [15] |
Katayama K, Murakami K, Sharp TM, Guix S, Oka T, Takai-Todaka R, Nakanishi A, Crawford SE, Atmar RL, Estes MK. Plasmid-based human norovirus reverse genetics system produces reporter-tagged progeny virus containing infectious genomic RNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(38): E4043-E4052. DOI:10.1073/pnas.1415096111 |
| [16] |
Sandoval-Jaime C, Green KY, Sosnovtsev SV. Recovery of murine norovirus and feline calicivirus from plasmids encoding EMCV IRES in stable cell lines expressing T7 polymerase[J]. Journal of Virological Methods, 2015, 217: 1-7. DOI:10.1016/j.jviromet.2015.02.003 |
| [17] |
Oliveira LM, Blawid R, Orilio AF, Andrade BYG, Souza ACA, Nagata T. Development of an infectious clone and replicon system of norovirus GII.4[J]. Journal of Virological Methods, 2018, 258: 49-53. DOI:10.1016/j.jviromet.2018.05.011 |
| [18] |
Stobart CC, Moore ML. RNA virus reverse genetics and vaccine design[J]. Viruses, 2014, 6(7): 2531-2550. DOI:10.3390/v6072531 |
| [19] |
Bailey D, Thackray LB, Goodfellow IG. A single amino acid substitution in the murine norovirus capsid protein is sufficient for attenuation in vivo[J]. Journal of Virology, 2008, 82(15): 7725-7728. DOI:10.1128/JVI.00237-08 |
| [20] |
Simmonds P, Karakasiliotis I, Bailey D, Chaudhry Y, Evans DJ, Goodfellow IG. Bioinformatic and functional analysis of RNA secondary structure elements among different genera of human and animal caliciviruses[J]. Nucleic Acids Research, 2008, 36(8): 2530-2546. DOI:10.1093/nar/gkn096 |
| [21] |
Beckert B, Masquida B. Synthesis of RNA by in vitro transcription[J]. Methods in Molecular Biology, 2011, 703: 29-41. |
| [22] |
Sosnovtsev S, Green KY. RNA transcripts derived from a cloned full-length copy of the feline calicivirus genome do not require VpG for infectivity[J]. Virology, 1995, 210(2): 383-390. DOI:10.1006/viro.1995.1354 |
| [23] |
Xia W, Bringmann P, Mcclary J, Jones PP, Manzana W, Zhu Y, Wang SJ, Liu Y, Harvey S, Madlansacay MR, et al. High levels of protein expression using different mammalian CMV promoters in several cell lines[J]. Protein Expression and Purification, 2006, 45(1): 115-124. DOI:10.1016/j.pep.2005.07.008 |
| [24] |
Robinson BA, Van Winkle JA, Mccune BT, Peters AM, Nice TJ. Caspase-mediated cleavage of murine norovirus NS1/2 potentiates apoptosis and is required for persistent infection of intestinal epithelial cells[J]. PLoS Pathogens, 2019, 15(7): e1007940. DOI:10.1371/journal.ppat.1007940 |
| [25] |
Daughenbaugh KF, Wobus CE, Hardy ME. VPg of murine norovirus binds translation initiation factors in infected cells[J]. Virology Journal, 2006, 3: 33. DOI:10.1186/1743-422X-3-33 |
| [26] |
Goodfellow I, Chaudhry Y, Gioldasi I, Gerondopoulos A, Natoni A, Labrie L, Laliberte JF, Roberts L. Calicivirus translation initiation requires an interaction between VPg and eIF4 E[J]. EMBO Reports, 2005, 6(10): 968-972. DOI:10.1038/sj.embor.7400510 |
| [27] |
Yunus MA, Lin XY, Bailey D, Karakasiliotis I, Chaudhry Y, Vashist S, Zhang G, Thorne L, Kao CC, Goodfellow I. The murine norovirus core subgenomic RNA promoter consists of a stable stem-loop that can direct accurate initiation of RNA synthesis[J]. Journal of Virology, 2015, 89(2): 1218-1229. DOI:10.1128/JVI.02432-14 |
| [28] |
McFadden N, Bailey D, Carrara G, Benson A, Chaudhry Y, Shortland A, Heeney J, Yarovinsky F, Simmonds P, Macdonald A, et al. Norovirus regulation of the innate immune response and apoptosis occurs via the product of the alternative open reading frame 4[J]. PLoS Pathogens, 2011, 7(12): e1002413. DOI:10.1371/journal.ppat.1002413 |
| [29] |
Yen JB, Wei LH, Chen LW, Chen LY, Hung CH, Wang SS, Chang PJ. Subcellular localization and functional characterization of GII.4 norovirus-encoded NTPase[J]. Journal of Virology, 2018, 92(5): e01824-17. |
| [30] |
Xu Q, Ni PE, Liu DL, Yin Y, Li QQ, Zhang J, Wu QP, Tian P, Shi XM, Wang DP. A bacterial surface display system expressing cleavable capsid proteins of human norovirus: A novel system to discover candidate receptors[J]. Frontiers in Microbiology, 2017, 8: 2405. DOI:10.3389/fmicb.2017.02405 |
| [31] |
Peterson ST, Kennedy EA, Brigleb PH, Taylor GM, Urbanek K, Bricker TL, Lee S, Shin H, Dermody TS, Boon ACM, et al. Disruption of type Ⅲ interferon (IFN) genes Ifnl2 and Ifnl3 recapitulates loss of the type Ⅲ IFN receptor in the mucosal antiviral response[J]. Journal of Virology, 2019, 93(22): e01073-19. |
| [32] |
Mears HV, Emmott E, Chaudhry Y, Hosmillo M, Goodfellow IG, Sweeney TR. Ifit1 regulates norovirus infection and enhances the interferon response in murine macrophage-like cells[J]. Wellcome Open Research, 2019, 4: 82. DOI:10.12688/wellcomeopenres.15223.1 |
| [33] |
Lin L, Han JG, Yan TB, Li LL, Li JS, Ao YY, Duan ZJ, Hou YD. Replication and transcriptionomic analysis of human noroviruses in human intestinal enteroids[J]. American Journal of Translational Research, 2019, 11(6): 3365-3374. |
| [34] |
Donaldson EF, Lindesmith LC, Lobue AD, Baric RS. Norovirus pathogenesis: mechanisms of persistence and immune evasion in human populations[J]. Immunological Reviews, 2008, 225(1): 190-211. DOI:10.1111/j.1600-065X.2008.00680.x |
| [35] |
Gutiérrez-Escolano AL, Brito ZU, Del Angel RM, Jiang X. Interaction of cellular proteins with the 5′ end of Norwalk virus genomic RNA[J]. Journal of Virology, 2000, 74(18): 8558-8562. DOI:10.1128/JVI.74.18.8558-8562.2000 |
| [36] |
Taube S, Perry JW, Mcgreevy E, Yetming K, Perkins C, Henderson K, Wobus CE. Murine noroviruses bind glycolipid and glycoprotein attachment receptors in a strain-dependent manner[J]. Journal of Virology, 2012, 86(10): 5584-5593. DOI:10.1128/JVI.06854-11 |
| [37] |
Vashist S, Urena L, Chaudhry Y, Goodfellow I. Identification of RNA-protein interaction networks involved in the norovirus life cycle[J]. Journal of Virology, 2012, 86(22): 11977-11990. DOI:10.1128/JVI.00432-12 |
| [38] |
Bailey D, Karakasiliotis I, Vashist S, Chung LMW, Reese J, Mcfadden N, Benson A, Yarovinsky F, Simmonds P, Goodfellow I. Functional analysis of RNA structures present at the 3′ extremity of the murine norovirus genome: the variable polypyrimidine tract plays a role in viral virulence[J]. Journal of Virology, 2010, 84(6): 2859-2870. DOI:10.1128/JVI.02053-09 |
| [39] |
López-Manríquez E, Vashist S, Ureña L, Goodfellow I, Chavez P, Mora-Heredia JE, Cancio-Lonches C, Garrido E, Gutiérrez-Escolano AL. Norovirus genome circularization and efficient replication are facilitated by binding of PCBP2 and hnRNP A1[J]. Journal of Virology, 2013, 87(21): 11371-11387. DOI:10.1128/JVI.03433-12 |
| [40] |
Mumphrey SM, Changotra H, Moore TN, Heimann-Nichols ER, Wobus CE, Reilly MJ, Moghadamfalahi M, Shukla D, Karst SM. Murine norovirus 1 infection is associated with histopathological changes in immunocompetent hosts, but clinical disease is prevented by STAT1-dependent interferon responses[J]. Journal of Virology, 2007, 81(7): 3251-3263. DOI:10.1128/JVI.02096-06 |
| [41] |
Thackray LB, Duan EN, Lazear HM, Kambal A, Schreiber RD, Diamond MS, Virgin HW. Critical role for interferon regulatory factor 3(IRF-3) and IRF-7 in type I interferon-mediated control of murine norovirus replication[J]. Journal of Virology, 2012, 86(24): 13515-13523. DOI:10.1128/JVI.01824-12 |
| [42] |
Chang KO. Role of cholesterol pathways in norovirus replication[J]. Journal of Virology, 2009, 83(17): 8587-8595. DOI:10.1128/JVI.00005-09 |
| [43] |
Arthur SE, Sorgeloos F, Hosmillo M, Goodfellow IG. Epigenetic suppression of interferon lambda receptor expression leads to enhanced human norovirus replication in vitro[J]. mBio, 2019, 10(5): e02155-19. |
| [44] |
Chang KO, George DW. Interferons and ribavirin effectively inhibit Norwalk virus replication in replicon-bearing cells[J]. Journal of Virology, 2007, 81(22): 12111-12118. DOI:10.1128/JVI.00560-07 |
| [45] |
Van Dycke J, Arnoldi F, Papa G, Vandepoele J, Burrone OR, Mastrangelo E, Tarantino D, Heylen E, Neyts J, Rocha-Pereira J. A single nucleoside viral polymerase inhibitor against norovirus, rotavirus, and sapovirus-induced diarrhea[J]. The Journal of Infectious Diseases, 2018, 218(11): 1753-1758. DOI:10.1093/infdis/jiy398 |
| [46] |
Lee S, Liu HJ, Wilen CB, Sychev ZE, Desai C, Hykes BL Jr, Orchard RC, Mccune BT, Kim KW, Nice TJ, et al. A secreted viral nonstructural protein determines intestinal norovirus pathogenesis[J]. Cell Host & Microbe, 2019, 25(6): 845-857. |
| [47] |
Hutson AM, Atmar RL, Graham DY, Estes MK. Norwalk virus infection and disease is associated with ABO histo-blood group type[J]. The Journal of Infectious Diseases, 2002, 185(9): 1335-1337. DOI:10.1086/339883 |
| [48] |
Wilen CB, Lee S, Hsieh LL, Orchard RC, Desai C, Hykes BL Jr, Mcallaster MR, Balce DR, Feehley T, Brestoff JR, et al. Tropism for tuft cells determines immune promotion of norovirus pathogenesis[J]. Science, 2018, 360(6385): 204-208. DOI:10.1126/science.aar3799 |
| [49] |
Zhang ZL, Liu DL, Wu QP, Lu Y, Tian P, Wang ZF, Wang DP. Characterization of a histo-blood group antigen-like substance in romaine lettuce that contributes to human norovirus attachment[J]. Journal of Agricultural and Food Chemistry, 2020, 68(5): 1207-1212. DOI:10.1021/acs.jafc.9b05887 |
| [50] |
Lohmann V, Körner F, Koch JO, Herian U, Theilmann L, Bartenschlager R. Replication of subgenomic hepatitis C virus RNAs in a hepatoma cell line[J]. Science, 1999, 285(5424): 110-113. DOI:10.1126/science.285.5424.110 |
| [51] |
Kitano M, Hosmillo M, Emmott E, Lu J, Goodfellow I. Selection and characterization of rupintrivir-resistant Norwalk virus replicon cells in vitro[J]. Antimicrobial Agents and Chemotherapy, 2018, 62(5): e00201-18. |
| [52] |
Chang KO, Kim Y, Lovell S, Rathnayake AD, Groutas WC. Antiviral drug discovery: Norovirus proteases and development of inhibitors[J]. Viruses, 2019, 11(2): 197. DOI:10.3390/v11020197 |
| [53] |
Xiong Q, Peng YH. Progress in human enteroviruses replicons[J]. Progress in Microbiology and Immunology, 2017, 45(5): 48-52. (in Chinese) 熊庆, 彭宜红. 人肠道病毒复制子的研究进展[J]. 微生物学免疫学进展, 2017, 45(5): 48-52. |
 2021, Vol. 48
2021, Vol. 48




