扩展功能
文章信息
- 韩淑梅, 李欣, 张芝元, 董旋, 韩燕峰, 梁宗琦
- HAN Shumei, LI Xin, ZHANG Zhiyuan, DONG Xuan, HAN Yanfeng, LIANG Zongqi
- 微生物角蛋白酶的特性及其应用研究进展
- Research progress on the characteristics and application of microbial keratinase
- 微生物学通报, 2021, 48(11): 4315-4326
- Microbiology China, 2021, 48(11): 4315-4326
- DOI: 10.13344/j.microbiol.china.210152
-
文章历史
- 收稿日期: 2021-02-05
- 接受日期: 2021-05-20
- 网络首发日期: 2021-07-01
角蛋白主要存在于哺乳动物、两栖动物、爬行动物和鸟类的表皮中,其是形成蹄、指甲、角、喙、羽毛、爪、毛发和人类皮肤外层的主要结构,包含较多的二硫键、氢键和疏水作用力,具有化学结构稳定、不溶于水及难降解的特点[1-2]。数百万吨的羽毛由于自然堆积时降解缓慢,附着的粪便、血液等含氮磷物质也易成为其他致病菌的营养源,处理不当将造成环境污染,这些“难降解”的角蛋白处置和管理方式是家禽养殖场、屠宰场等行业面临的主要问题之一[3]。但从另一方面来说羽毛等角蛋白废弃物中含有丰富的蛋白质,其中粗蛋白含量在80%以上,角蛋白还含有大量动植物生长必需的氨基酸,如胱氨酸、赖氨酸、脯氨酸等,含量可达氨基酸总量的70%[4]。目前工业上常用填埋法、物理法和化学法处理废弃羽毛[5],但具有环境污染严重、必需氨基酸破坏、耗能高等缺点。此外,由于人类皮肤病原真菌很多是嗜角蛋白真菌,如毛癣菌属(Trichophyton)、小孢子菌属(Microsporum)及表皮癣菌属(Epidermophyton)等,随意丢弃羽毛会使环境中富集这类病原真菌,极大地增加人类疾病的发生概率[6-8]。因此,迫切需要一种新型环保的生物催化剂来降解角蛋白。鉴于这一事实,微生物角蛋白酶作为一种有效的生物降解法引起了人们的关注,因其低成本、易获得和无污染的特性已被应用于皮革、纺织、制药等工业领域[9]。角蛋白酶的微生物来源广泛、性质各异,因此,选择合适微生物来源的角蛋白酶用于工业生产意义重大。
产角蛋白酶的微生物分布于不同生境中,尤其在羽毛倾倒场、家禽废物、屠宰场废物等生境中较为丰富[10]。目前已报道的产角蛋白酶的微生物主要是细菌和真菌,如苏云金芽孢杆菌(Bacillus thuringiensis)、枯草芽孢杆菌(B. subtilis)、地衣芽孢杆菌(B. licheniformis)等[11],它们产生的热稳定角蛋白酶具有活性较高、分解羽毛效果好的特点[12];已报道的产角蛋白酶放线菌主要是链霉菌属(Streptomyces)菌株,如白色链霉菌(Str. albus)、弗氏链霉菌(Str. fradiae)、革兰氏链霉菌(Str. graminofaciens)、热硝化链霉菌(Str. thermonitrificans)和古巴尔古链霉菌(Str. gulbarguensis)等,它们能利用不同的角蛋白底物产生角蛋白酶[13]。最早发现的角蛋白酶来源于真菌——马爪甲团囊菌(Onygena equina)[14],随后越来越多能产生角蛋白酶的真菌被报道,如小孢子菌属(Microsporum)、金孢霉属(Chrysosporium)、地丝霉属(Geomyces)、毛癣菌属(Trichophyton)、枝顶孢属(Acremonium)、拟青霉属(Paecilomyces)和侧齿霉属(Engyodontium)等[15-17]。某些真菌角蛋白酶能破坏动物及人体皮肤外层角蛋白,从而引起头癣、体癣和脚癣等皮肤类疾病[6];此外,由于真菌产生的角蛋白酶生物化学性质多样,已备受关注。基于此,本文主要综述了不同微生物角蛋白酶的生化特性、分子生物学特性,并对微生物角蛋白酶的应用进行了介绍,以期为角蛋白酶的相关研究提供参考。
1 角蛋白酶的生物化学特性 1.1 一般特性角蛋白酶的特性因微生物的来源而异。多数微生物角蛋白酶是中性或碱性蛋白酶,pH在7.5−9.0之间,一些角蛋白酶在极端碱性或微酸性条件下也具有最佳活性。少数角蛋白酶在很宽的pH范围内也能保持活性稳定。如拟诺卡氏菌属(Nocardiopsis) strain TOA-1,所产角蛋白酶能在30 ℃、pH 1.5−12.0范围内保持酶活性24 h[18]。
角蛋白酶的作用温度与来源微生物的生存环境有关,多数微生物来源的角蛋白酶最适温度在40−70 ℃之间。一株嗜热厌氧菌闪烁杆菌属(Fervidobacterium sp.)角蛋白酶最适温度是80 ℃,嗜麦芽寡养单胞菌(Stenotrophomonas maltophila) DHHJ的角蛋白酶在40 ℃活性最大[19];在一些特殊条件下,来源于海岛闪烁杆菌(F. islandicum) AW-1角蛋白酶的最佳活性为100 ℃[20]。
角蛋白酶的分子量范围是18−240 kD[21-22],但大多数角蛋白酶的分子量小于50 kD。多数角蛋白酶是单体酶,也存在多聚角蛋白酶[20],有些角蛋白酶的分子量高达440 kD[23],详见表 1。
| 微生物 Microorganisms |
来源 Source |
水解类型 Catalytic type |
最适pH Optimum pH |
最适温度 Optimum temperature (℃) |
分子量 Molecular weight (kD) |
参考文献 References |
| Bacillus licheniformis PWD-1 | Poultry waste digester | Serine proteases | 7.5 | 50 | 33.00 | [24] |
| Fervidobacterium | Hot spring | Serine proteases | 10.0 | 80 | 130.00 | [25] |
| Bacillus sp. BK111 | Poultry feather | Serine proteases | 9.5 | 60 | 42.00 | [26] |
| Bacillus subtilis S1-4 | Chicken feather | Serine proteases | 9.0 | 50 | 36.10 | [27] |
| Bacillus thuringiensis MT1 | Cattle-yard | Metallo protease | 9.0 | 50 | 80.00 | [28] |
| Streptomyces sp. AB1 | Soil collected from dump yards, slaughter house, and poultry farms | Serine proteases | 11.5 | 75 | 29.85 | [29] |
| Actinomadura keratinilytica Cpt 29 | Poultry compost | Serine proteases | 8.0 | 85 | 135.00 | [30] |
| Streptomyces minutiscleroticus DNA38 | Soil of poultry farm | Serine proteases | 9.0 | 50 | 29.00 | [31] |
| Streptomyces albidoflavus Fea-10 | Poultry waste | Serine proteases | 10.0 | 50 | 36.00 | [32] |
| Scopulariopsis brevicaulis | Poultry farm soil | Serine proteases | 8.0 | 40 | 36.00−39.00 | [33] |
| Actinomadura viridilutea DZ50 | Algerian fishing port | Serine proteases | 11.0 | 80 | 9.54 | [34] |
| Candida parapsilosis | Poultry waste | − | 7.4 | 37 | 100.00 | [35] |
| Trichophyton sp. 95 | Rotted Feather soil | − | 7.0 | 60 | 43.00 | [36] |
| Paecilomyces marquandii | Soil | Serine proteases | 8.0 | 60–65 | 33.00 | [37] |
| Doratomyces microsporus | Soil | Serine proteases | 8.0 | 50 | 30.00 | [37] |
| Note: –: Not mentioned | ||||||
底物特异性是定义“角蛋白酶”的主要标准之一。角蛋白酶能有效水解酪蛋白、明胶、牛血清白蛋白等可溶性蛋白底物,也能水解羽毛、羊毛、指甲等不溶性蛋白底物。有研究者提出将K: C (Keratinolytic: Caseinolytic)活性的比率作为参数来判断某种酶作为角蛋白酶的潜力,即在标准条件下,用羽毛角蛋白作为角蛋白分解活性底物,酪蛋白作为酪蛋白分解活性底物[38]。K: C=0.5被认为是角蛋白酶的理想比率,K: C > 0.5是一种潜在的角蛋白酶,K: C < 0.5是非角蛋白酶。
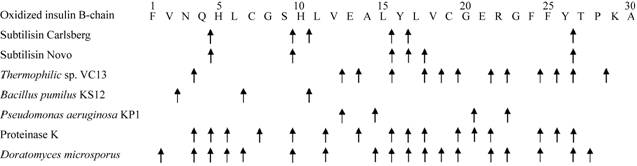
另外,角蛋白酶区别于其他蛋白酶的一个显著特征就是能够切割疏水性和芳香族氨基酸[39]。角蛋白酶的底物特异性可通过胰岛素氧化B链水解得到证实(图 1)。胰岛素B链由大量疏水氨基酸残基组成,还含有几个带电荷的侧链残基,半胱氨酸残基被氧化后能产生2个额外的负电荷,来自Thermophilic sp. VC13的角蛋白酶在氧化半胱氨酸裂解后水解肽键;Doratomyces microsporus角蛋白酶表现出广泛的特异性,切割位点与蛋白酶K的切割位点相似,对亮氨酸(L)、缬氨酸(V)、酪氨酸(Y)和苯丙氨酸(F)的P1位点上的疏水和芳香氨基酸具有优先选择性。据报道,真菌D. microsporus角蛋白酶的切割位点最多,有19个[37]。
|
| 图 1 胰岛素B链水解显示多种角蛋白酶的裂解位点 Figure 1 Cleavage sites of various keratinases as revealed by insulin B chain hydrolysis |
|
|
根据催化类型,角蛋白酶主要分为丝氨酸蛋白酶或金属蛋白酶。丝氨酸蛋白酶能被苯甲基磺酰氟(Phenylmethylsulfonyl Fluoride,PMSF)强烈抑制,金属蛋白酶活性中心含有金属离子(Ca2+、Zn2+),能被乙二胺四乙酸(Ethylene Diamine Tetraacetic Acid,EDTA)和1, 10-邻菲罗啉抑制[40]。Streptomyces sp.角蛋白酶被PMSF和EDTA抑制,因此被鉴定为丝氨酸金属蛋白酶[41]。
不同金属离子对角蛋白酶活性影响各异。一些金属离子会对角蛋白酶活性起促进作用,如Ca2+、Mg2+能增强角蛋白酶的活性,Fe2+及Fe3+能提高酶活力,有研究表明Fe2+能增加嗜角蛋白金孢霉(C. keratinophilum)角蛋白酶活性[42]。一些过渡金属离子及重金属离子Cu+、Hg2+、Ag+、Pb+、Zn2+、Ba2+、Co+可能会抑制角蛋白酶的活性[20, 40],这与金属离子和底物竞争酶分子的活性位点有关。5 mmol/L的K+、Ca2+、Cu2+能提高B. subtilis Y3-4角蛋白酶的活性,其中,Cu2+提高了其25%的酶活力,而5 mmol/L的Zn2+、Ba2+、Co+、Ni2+、Mn2+明显降低了该菌株角蛋白酶活性,Co+、Ni2+抑制了其40%左右的酶活,而同样浓度的Sr2+、Li+、Na+、Mg2+则没有显著性影响[41]。
非离子洗涤剂和溶剂对角蛋白酶活性具有抑制、激发或稳定的作用。角蛋白酶通常在有机溶剂、SDS、DMSO、Triton X-100中是稳定存在的[40]。对于嗜麦芽窄食单胞菌(Ste. maltophilia)菌株BBE11-1分泌的3种角蛋白酶中的K1、K2来说,非离子去垢剂Triton X-100和吐温-20促进角蛋白酶活性,而离子型去垢剂SDS显著抑制其活性[43]。
据报道,还原剂如二硫苏糖醇(Dithiothreitol,DTT)、β-巯基乙醇(β-Mercaptoethanol,β-ME)、还原型谷胱甘肽、半胱氨酸和亚硫酸钠能增强角蛋白酶的活性,表明这些酶能够被硫醇激活,对于B. licheniformis MUK3所分泌的角蛋白酶,还原剂DTT和β-ME使角蛋白酶活性增加49%−73%[44]。事实上,还原剂通过破坏二硫键来促进角蛋白酶的水解,体外纯化的角蛋白酶只有通过加入还原剂才能发挥降解作用[18, 45]。
2 角蛋白酶的分子生物学特性 2.1 角蛋白酶结构特性对角蛋白酶核苷酸序列的研究,有助于解释其结构-功能的关系。多数微生物角蛋白酶与枯草杆菌蛋白酶有很高的序列同源性。目前,已有部分菌株的角蛋白酶基因成功克隆并测序,如地衣芽孢杆菌PWD-1[45]、链霉菌SCUT-3[46]、发癣菌KERB[36]等,其中,以地衣芽孢杆菌角蛋白酶研究最为深入。
除了少数角蛋白酶基因序列,多数角蛋白酶氮端区域(AQTVPY)高度保守。地衣芽孢杆菌PWD-1角蛋白酶的氮端序列与Subtilisin Carlsberg相同(表 2),他们同属于丝氨酸蛋白酶家族,其编码的基因kerA与Subtilisin Carlsberg相似性达99%,与Subtilisin E、Subtilisin BPN相似性为65%,与Fervidolysin相似性为30%[18]。
| 蛋白酶类型及来源 Protease type and source |
N-端氨基酸序列 N-terminal amino acid sequence |
参考文献 References |
| Keratinase A (B. licheniformis PWD-1) | AQTVPYGIPLIKADK | [45] |
| Subtilisin Carlsberg (B. licheniformis) | AQTVPYGIPLIKADK | [47] |
| Subtilisin E (B. subtilis) | AQSVPYGISQIKAPA | [48] |
| Subtilisin BPN (B. amyloliquefaciens) | AQSVPYGVSQIKAPA | [49] |
| Streptomyces griseus protease B (Str. griseus) | ISGGDAIYSSTGRCS | [21] |
| SFase-2 (Str. fradiae ATCC 14544) | IAGGEAIYAAGGGRC | [50] |
| NAPase (Nocardiopsis sp. TOA-1) | ADIIGGLAYTMGGRCS | [51] |
| Fervidolysin (F. pennivorans) | SKAKDLASLPEIKSQ | [52] |
| Keratinase Pm (P. marquandii) | ALTQQPGAPWGLG | [38] |
| Keratinase Dm (D. microsporus) | ATVTQNNAPWGLG | [38] |
| Keratinase Af (A. fumigatus) | ALTTQKGAPWGLGSI | [53] |
| Keratinase Pa (P. aeruginosa) | AEAGGPGG | [54] |
尽管F. pennavorans产生的嗜热角蛋白酶N端氨基酸序列与枯草杆菌蛋白酶不同,但其活性位点区域与枯草杆菌蛋白酶样丝氨酸蛋白酶类似。真菌角蛋白酶N端氨基酸序列的比较表明它们与枯草杆菌蛋白酶的关系比放线菌角蛋白酶的关系更密切。来自链霉菌的角蛋白酶更接近灰色链霉菌(Str. griseus)蛋白酶B,后者是链霉素蛋白酶的主要成分[21]。来自铜绿假单胞菌(P. aeruginosa)的角蛋白酶N端氨基酸序列显示出与其他微生物角蛋白酶的相似性较小。
Wu等报道了台湾亚栖热菌(Meiothermus taiwanensis) WR-220角蛋白酶MtaKer 1.50 Å晶体结构,其由信号肽、N端前肽和成熟蛋白酶3部分组成,该酶是由Asp39、His72和Ser224残基组成的高度保守的催化三联体结构,并且在成熟的rMtaKer结构中还发现了2个钙离子结合位点及2个二硫键,其中,钙离子对蛋白质正确折叠和结构稳定起着重要作用,二硫键可以增强该蛋白的热稳定性,由Ser130-Leu131-Gly132组成的S1结合口袋是角蛋白酶与底物的结合位点[55]。Kim等分析了F. pennivorans 1.7 Å角蛋白酶晶体结构,其由1个催化结构域、1个前肽结构域和2个β-三明治结构域组成;其催化结构域与枯草芽孢杆菌蛋白酶类似,其中,催化结构域内的钙结合位点与Subtilisin E完全一致[23],高分子量的角蛋白酶可能由多结构域或寡聚蛋白酶组成,这种多结构域Fervidolysin晶体结构为深入了解酶原激活和非催化结构域作用提供了依据。
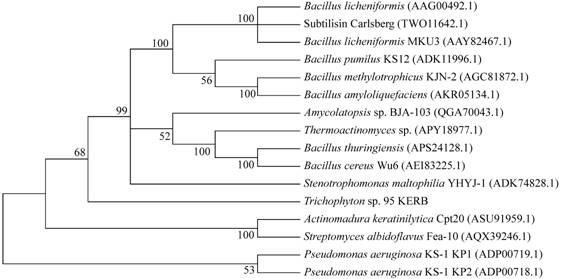
2.2 角蛋白酶氨基酸序列分子进化树使用MEGA 6.0软件中邻接(Neighbor-Joining,NJ)法构建部分角蛋白酶的系统发育树。其中15个角蛋白酶的氨基酸序列从NCBI下载,发癣菌KERB氨基酸序列从文献[36]获得,他们均有角蛋白酶活性。从图 2可以看出这些角蛋白酶分为2个簇,第一个簇属于S8蛋白酶家族,其中,Actinomadura keratinilytica和Str. albidoflavus来源的角蛋白酶独立于其他分支,第二个簇中P. aeruginosa KS-1 KP1属于M28蛋白酶家族,P. aeruginosa KS-1 KP2属于M4蛋白酶家族,虽然他们都来源于P. aeruginosa KS-1菌株,但亲缘关系十分疏远。P. aeruginosa角蛋白酶独立于其他角蛋白酶,从而扩大了角蛋白酶的范围。
|
| 图 2 部分角蛋白酶氨基酸序列分子系统树 Figure 2 Phylogenetic tree based on partial keratinase amino acid sequences 注:分支上的数据表示Bootstrap检验的支持百分率,自展支持值(Bootstrap)≥50%的显示在各个进化分支节点上;括号内的GenBank登录号 Note: The confidence values over 50% from 1 000 replicate bootstrap samplings are shown at each node; GenBank accession numbers are shown in the parentheses |
|
|
据文献报道,S8蛋白酶家族成员通过催化三联体机制工作,Asp、His和Ser的催化三联体及含氧阴离子空穴Asn(N)周围的区域在角蛋白酶中都是高度保守的[56-57]。可对这些保守区域设计通用引物来克隆新的角蛋白酶。
2.3 角蛋白酶的异源表达随着越来越多的角蛋白酶基因被鉴定出来,角蛋白酶在工程表达系统中的异源表达引起了人们的极大关注。
大肠杆菌由于生长迅速、易于操作、遗传背景简单等优点,是最常用的表达宿主[58],来源于Ste. maltophilia BBE11-1的2个角蛋白酶基因kerSMD和kerSMF在大肠杆菌BL21(DE3)中成功表达[42],P. aeruginosa KS-1的角蛋白酶基因kerP在HB101中成功表达[59]。但大肠杆菌表达系统仍存在一些问题,如形成无活性的包涵体和分泌效率低,角蛋白酶在宿主细胞中的大量累积会导致细胞毒性和不正确的蛋白质折叠,最终导致细胞溶解和目标蛋白前体失活[58]。
芽孢杆菌因具有外源蛋白易分泌到胞外、易分离纯化、无致病性等优势而被广泛研究[60]。B. licheniformis PWD-1角蛋白酶基因kerA首次在枯草芽孢杆菌DB104中表达[61],Radha等在巨大芽孢杆菌MS941和巨大芽孢杆菌ATCC14945中实现了B. licheniformis MKU3角蛋白酶基因的稳定表达[43]。但是多余的内源蛋白酶及质粒的不稳定性是芽孢杆菌表达系统面临的主要问题。
毕赤酵母因具有完善的分泌表达体系,能将外源蛋白完全分泌至胞外,并且具有外源蛋白翻译后修饰功能,更适用于真核生物角蛋白酶基因的表达[62]。来自Aspergillus fumigatus和P. aeruginosa的角蛋白酶基因在毕赤酵母SMD1165和毕赤酵母SMD1168H中成功表达[53]。但毕赤酵母系统中的表达水平强烈依赖于密码子使用偏好性,Hu等实现了B. licheniformis S90的角蛋白酶在毕赤酵母中的表达,并且与在大肠杆菌系统中的表达相比,通过密码子优化策略提高了12倍的角蛋白酶产量[63]。
人们也在尝试将新型宿主用于角蛋白酶的表达,昆虫细胞杆状病毒表达载体系统(Baculovirus Expression Vector System,BEVS)具有高水平的细胞内表达、对外源基因翻译后修饰充分、生物安全性好的优势,已被广泛用于医学研究、药物研发、疫苗生产等众多领域。Huang等首次使用昆虫细胞Spodoptera frugiperda (sf9)成功表达了来自B. licheniformis PWD-1的角蛋白酶基因,酶活635 U/mg,这为人们选择表达宿主提供了新思路[64]。
3 角蛋白酶的应用微生物角蛋白酶是用于角蛋白降解的特异酶,已在动物饲料、生物肥料、洗涤工业、皮革和纺织工业等方面得到应用[18, 57, 65],在其他生物技术领域如制药和生物医学等方面也具有重要应用潜力。
3.1 饲料及肥料中的应用角蛋白水解产物富含改善动物饲料的必需氨基酸[1],当角蛋白酶与动物饲料一起添加时,能有效改善动物对饲料的吸收利用,加速动物生长,提高消化率。据报道,在含有羽毛粉的动物饲料中添加玫瑰考克氏菌(Kocuria rosea),能有效提高组氨酸、蛋氨酸和赖氨酸的含量[66]。角蛋白酶还能够分解朊病毒蛋白(Prion Protein,PrpSc),使肉制品安全地供动物食用[67],Versazyme和Cibenza DP100TM是从地衣芽孢杆菌PWD-1中获得的2种商业角蛋白酶,在肉鸡饲料中添加Versazyme能够促进肉鸡生长及消化。
金黄杆菌属(Chryseobacterium spp.)菌株水解羽毛产物已经用于提高香蕉植物的含氮量,并能促进其他植物根和芽的生长[68]。
3.2 沼气生产将角蛋白废弃物经微生物处理可转化为可再生能源沼气,用于改善卫生条件、保护生态环境、解决能源短缺等问题[9]。有研究表明可通过两步法来产生沼气:第一步:由角蛋白酶产生菌水解角蛋白底物;第二步:由热球菌进一步将水解产物降解为小分子,如氢气;此外,通过补充角蛋白水解产物,进一步产生甲烷气体[69]。有研究表明用B. megaterium作为角蛋白酶产生菌处理鸡毛,能够产生甲烷气体,将羽毛水解物与稀释的猪粪在厌氧条件下消化,比单独用猪粪发酵能产生更多的甲烷[70]。
3.3 洗涤剂中的应用角蛋白酶的生物化学性质决定了其能够耐受各种表面活性剂、洗涤剂及各种有机化学试剂。有研究表明B. subtilis产生的角蛋白酶能清除棉布衣服上的血液污渍,将角蛋白酶加入商业洗涤剂中,有强化去除污渍的效果,将B. pumilus GRK产生的角蛋白酶加入洗涤剂,在不损坏纺织品前提下,成功地去除了衣服上的血渍[71]。
3.4 皮革和纺织工业中的应用在皮革工业中,去除动物身上的毛发而不损坏皮肤是脱毛过程遇到的巨大挑战之一,角蛋白酶由于其底物特异性,可被用于皮革工业的脱毛过程,具有绿色、无二次污染、经济等显著优点[72]。来自B. paralicheniformis MKU3的角蛋白酶能有效对羊皮进行100%的脱毛而不损坏皮革表层,这个过程因为没有用硫化物等进行预处理,因此被称为无化学物质脱毛过程[9]。
3.5 医药方面的应用角化过度通常发生在手指背面的外层皮肤,其主要成分是角蛋白,基于角蛋白酶的药物Keratoclean®Hydra PB及Pure100可用于治疗皮层角化过度症状。痤疮是由于过多的角蛋白阻塞了皮脂腺而形成。Keratoclean Sensitive PB和Keratopeel PB已用于痤疮治疗[67]。朊病毒是导致传染性和致命性脑部疾病的关键传染性蛋白质之一[73]。羽毛分解产生的角蛋白酶具有切割β-角蛋白结构的功能;因此,它们可以降解具有β折叠片的朊病毒蛋白(PrPSc)。地衣芽孢杆菌PWD-1是发现的首个能产生降解PrPSc角蛋白酶的菌株,另外,Pure100已被用于水解手术器械中的朊病毒。由于角蛋白酶可以分解指甲等基质中的角蛋白,因此可以增强药物的渗透性,提高疾病的治疗效果[67]。
综上,角蛋白酶的应用范围非常广,除上述领域外,角蛋白酶也可以应用到化妆品和护肤品中更好地帮助营养成分的吸收[74],今后仍需在更多的应用领域继续探索。
4 展望 4.1 角蛋白降解微生物资源筛选尽管目前已陆续报道了一些角蛋白降解微生物,但不同的微生物对不同底物的降解效果不同。因此仍有大量资源值得进一步筛选并进行降解效果评价,同时,不同角蛋白酶在不同环境条件下的酶活不同,导致降解效果不同;为适应不同条件下角蛋白酶对底物进行高效降解,开发具有广泛底物特异性和较高催化活性的新型角蛋白酶势在必行。因此极端环境中的角蛋白降解微生物如嗜热和嗜盐微生物等,值得广泛关注。此外,随着高通量测序技术的发展,筛选的技术手段也需要改进,利用高通量测序技术、基因组数据库及生物信息学的方法来分析和发现新的角蛋白酶和新的角蛋白降解微生物是一个有效途径。
4.2 微生物角蛋白酶的生物降解机理迄今角蛋白的生物降解机制还未探明,但有多种假设,如酶解理论、硫解理论及物理压力理论等。但确定的是一个有效的角蛋白降解过程必然包括:二硫键的还原以及角蛋白多肽链的水解[55]。二硫键的破坏是角蛋白酶进入角蛋白底物的先决条件。酶解理论认为,二硫键还原酶先对底物中的二硫键进行还原[73],然后,角蛋白酶能顺利进入角蛋白底物内部进行水解。尽管羽毛角蛋白很难被蛋白水解酶降解,但角蛋白酶和真菌菌丝体机械破坏相结合可以较好地完成这项复杂工作,真菌菌丝在含有角蛋白底物的培养基中不断生长并侵入角蛋白,其肽键逐渐暴露于角蛋白酶下,进而被降解。鸡毛中的β-角蛋白很难被普通蛋白水解酶水解,研究发现Microsporum fulvum菌丝对角蛋白的物理压力及其分泌的角蛋白酶对角蛋白的水解增强了其对底物的降解能力,最后将含角蛋白的基质全部降解[75]。
因此,不同微生物产生的角蛋白酶特性不同,再加上角蛋白结构的复杂性,使角蛋白酶对底物的降解可能有不同机制,未来仍需深入研究。
4.3 角蛋白酶的蛋白质工程在工业应用当中,角蛋白酶需要更高效、高产和耐热的特性。蛋白质工程中关键技术的成熟,为改造蛋白质结构使其具有新的特征和更高的产能提供了机会。
提高异源宿主中角蛋白酶产量最重要的因素是筛选高分泌效率的信号肽,信号肽是指导新合成的蛋白质转移到分泌途径的短肽链。Tian等对B. subtilis SCK6信号肽进行优化,发现使用SPLipA信号肽的角蛋白酶的胞外活性比野生型高1.95倍[76]。Fu等构建了包含173种不同Sec型信号肽的文库,认为几乎没有任何通用的最佳信号肽适用于所有异源蛋白质[77]。因此,系统优化最佳信号肽是促进不同异源表达系统产生角蛋白酶的有力策略。
在前肽自主加工过程中,前肽结构在辅助蛋白质成熟和结构重排中起到重要作用,“前肽工程”聚焦于对角蛋白酶前肽的修饰,以提高酶的产量及催化特性[78]。Sharma等对角蛋白酶KerP的N末端区域进行随机截短,发现随着截短长度的延长,酶活性逐渐降低,只有187 bp前序列区域是角蛋白酶KerP正确折叠并且具有活性所需的最小长度。因此,前序列的大小直接影响蛋白质的构象,从而导致酶动力学的改变[59]。角蛋白酶Sfp2的氮端前序列的定点突变使酶的比活性和蛋白质产量提高了9倍[79]。“前肽工程”可利用定点突变、交换、嵌合体和基因重组手段对蛋白质的前肽区域进行修饰,其是提高蛋白酶产量和产生特定突变体很有前途的技术。
据报道,一些不必要的氨基酸或结构域存在于酶的碳末端,影响催化效率和酶的性质。部分截短角蛋白酶的C末端序列可以提高其催化效率、嗜热性、耐盐性和耐洗涤剂性[80]。因此,碳末端修饰已被探索为一种新的蛋白质工程策略。
蛋白酶最重要的区域是其催化中心以及和底物的结合区。对蛋白酶催化中心的电子分析以及定点突变可以提高酶的催化效率[81],通过计算机模拟角蛋白酶KerSMD的3D结构发现,该酶口袋的入口或底部的4个残基可能与底物特异性相关,通过突变这些残基使S1口袋扩大,发现酶的催化效率提高且可水解大分子底物[80]。这些研究为角蛋白酶中催化中心结合囊、催化能力和底物特异性之间的互作提供了依据。
多种突变策略的组合可能会通过协同作用对角蛋白酶产生叠加效应。用具有更高催化效率的同源蛋白酶KerSMF的前肽和碳末端结构域取代KerSMD的相关区域,发现角蛋白酶KerSMD的催化能力和热稳定性都有提高[80]。此外,同时进行多位点突变可以作为一种有效的方法来改善和定制角蛋白酶。
| [1] |
Onifade AA, Al-Sane NA, Al-Musallam AA, Al-Zarban S. A review: potentials for biotechnological applications of keratin-degrading microorganisms and their enzymes for nutritional improvement of feathers and other keratins as livestock feed resources[J]. Bioresource Technology, 1998, 66(1): 1-11. DOI:10.1016/S0960-8524(98)00033-9 |
| [2] |
Bagewadi ZK, Mulla SI, Ninnekar HZ. Response surface methodology based optimization of keratinase production from Trichoderma harzianum isolate HZN12 using chicken feather waste and its application in dehairing of hide[J]. Journal of Environmental Chemical Engineering, 2018, 6(4): 4828-4839. DOI:10.1016/j.jece.2018.07.007 |
| [3] |
Bhari R, Kaur M, Singh RS, Pandey A, Larroche C. Bioconversion of chicken feathers by Bacillus aerius NSMk2: a potential approach in poultry waste management[J]. Bioresource Technology Reports, 2018, 3: 224-230. DOI:10.1016/j.biteb.2018.07.015 |
| [4] |
Shen X. Dynamic changes of fungal community during the degradation of chicken feathers under artificial conditions[D]. Guiyang: Master's Thesis of Guizhou University, 2019 (in Chinese) 沈鑫. 人工条件下鸡毛降解过程中真菌群落的动态变化[D]. 贵阳: 贵州大学硕士学位论文, 2019 |
| [5] |
Bai QW, Zhang SB, Li R, Ma XY, Nordin NI, Chen SE, Ding GT. Advances in keratin degradation technology[J]. Journal of Microbiology, 2020, 40(3): 119-123. (in Chinese) 摆倩文, 张邵博, 李儒, 马咸莹, 诺如·以扎·诺丁, 陈士恩, 丁功涛. 角蛋白酵解工艺研究进展[J]. 微生物学杂志, 2020, 40(3): 119-123. DOI:10.3969/j.issn.1005-7021.2020.03.020 |
| [6] |
Zhang ZY, Zheng H, Han YF, Chen WH, Liang ZQ. Diversity of keratin-degrading fungi from body surfaces of different pet dogs[J]. Mycosystema, 2017, 36(5): 563-572. (in Chinese) 张芝元, 郑欢, 韩燕峰, 陈万浩, 梁宗琦. 不同宠物狗体表一些部位角蛋白降解真菌物种多样性[J]. 菌物学报, 2017, 36(5): 563-572. |
| [7] |
Yusuf I, Ahmad SA, Phang LY, Syed MA, Shamaan NA, Abdul Khalil K, Dahalan FA, Shukor MY. Keratinase production and biodegradation of polluted secondary chicken feather wastes by a newly isolated multi heavy metal tolerant bacterium-Alcaligenes sp. AQ05-001[J]. Journal of Environmental Management, 2016, 183: 182-195. DOI:10.1016/j.jenvman.2016.08.059 |
| [8] |
Jana A, Halder SK, Dasgupta D, Hazra S, Mondal P, Bhaskar T, Ghosh D. Keratinase biosynthesis from waste poultry feathers for proteinaceous stain removal[J]. ACS Sustainable Chemistry & Engineering, 2020, 8(48): 17651-17663. |
| [9] |
Hassan MA, Abol-Fotouh D, Omer AM, Tamer TM, Abbas E. Comprehensive insights into microbial keratinases and their implication in various biotechnological and industrial sectors: a review[J]. International Journal of Biological Macromolecules, 2020, 154: 567-583. DOI:10.1016/j.ijbiomac.2020.03.116 |
| [10] |
Korniłłowicz-Kowalska T, Bohacz J. Biodegradation of keratin waste: theory and practical aspects[J]. Waste Management, 2011, 31(8): 1689-1701. DOI:10.1016/j.wasman.2011.03.024 |
| [11] |
Lateef A, Adelere IA, Gueguim-Kana EB. Bacillus safensis LAU 13: a new source of keratinase and its multi-functional biocatalytic applications[J]. Biotechnology & Biotechnological Equipment, 2015, 29(1): 54-63. |
| [12] |
Kanoksilapatham W, Pasomsup P, Keawram P, Cuecas A, Portillo MC, Gonzalez JM. Fervidobacterium thailandense sp. nov., an extremely thermophilic bacterium isolated from a hot spring[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(12): 5023-5027. DOI:10.1099/ijsem.0.001463 |
| [13] |
Nouioui I, Carro L, García-López M, Meier-Kolthoff JP, Woyke T, Kyrpides NC, Pukall R, Klenk HP, Goodfellow M, Göker M. Genome-based taxonomic classification of the phylum Actinobacteria[J]. Frontiers in Microbiology, 2018, 9: 2007. DOI:10.3389/fmicb.2018.02007 |
| [14] |
Jain PC, Agrawal SC. A note on the keratin decomposing capability of some fungi on pig hair and peacock feather[J]. Transactions of the Mycological Society of Japan, 1980, 21(4): 513-517. |
| [15] |
Sharma R, Devi S. Versatility and commercial status of microbial keratinases: a review[J]. Reviews in Environmental Science and Bio/Technology, 2018, 17(1): 19-45. DOI:10.1007/s11157-017-9454-x |
| [16] |
El-Gendy MMA. Keratinase production by endophytic Penicillium spp. Morsy1 under solid-state fermentation using rice straw[J]. Applied Biochemistry and Biotechnology, 2010, 162(3): 780-794. DOI:10.1007/s12010-009-8802-x |
| [17] |
Zhang YW, Zeng GP, Zou X, Han YF, Liang ZQ, Qiu SY. Two new keratinophilic fungal species[J]. Phytotaxa, 2017, 303(2): 173. DOI:10.11646/phytotaxa.303.2.7 |
| [18] |
Gupta R, Ramnani P. Microbial keratinases and their prospective applications: an overview[J]. Applied Microbiology and Biotechnology, 2006, 70(1): 21-33. DOI:10.1007/s00253-005-0239-8 |
| [19] |
Cao ZJ, Zhang Q, Wei DK, Chen L, Wang J, Zhang XQ, Zhou MH. Characterization of a novel Stenotrophomonas isolate with high keratinase activity and purification of the enzyme[J]. Journal of Industrial Microbiology & Biotechnology, 2009, 36(2): 181-188. |
| [20] |
Nam GW, Lee DW, Lee HS, Lee NJ, Kim BC, Choe EA, Hwang JK, Suhartono MT, Pyun YR. Native-feather degradation by Fervidobacterium islandicum AW-1, a newly isolated keratinase-producing thermophilic anaerobe[J]. Archives of Microbiology, 2002, 178(6): 538-547. DOI:10.1007/s00203-002-0489-0 |
| [21] |
Bressollier P, Letourneau F, Urdaci M, Verneuil B. Purification and characterization of a keratinolytic serine proteinase from Streptomyces albidoflavus[J]. Applied and Environmental Microbiology, 1999, 65(6): 2570-2576. DOI:10.1128/AEM.65.6.2570-2576.1999 |
| [22] |
Bernal C, Cairó J, Coello N. Purification and characterization of a novel exocellular keratinase from Kocuria rosea[J]. Enzyme and Microbial Technology, 2006, 38(1/2): 49-54. |
| [23] |
Kim JS, Kluskens LD, De Vos WM, Huber R, Van Der Oost J. Crystal structure of fervidolysin from Fervidobacterium pennivorans, a keratinolytic enzyme related to subtilisin[J]. Journal of Molecular Biology, 2004, 335(3): 787-797. DOI:10.1016/j.jmb.2003.11.006 |
| [24] |
Lin X, Lee CG, Casale ES, Shih JC. Purification and characterization of a keratinase from a feather-degrading Bacillus licheniformis strain[J]. Applied and Environmental Microbiology, 1992, 58(10): 3271-3275. DOI:10.1128/aem.58.10.3271-3275.1992 |
| [25] |
Friedrich AB, Antranikian G. Keratin degradation by Fervidobacterium pennavorans, a novel thermophilic anaerobic species of the order thermotogales[J]. Applied and Environmental Microbiology, 1996, 62(8): 2875-2882. DOI:10.1128/aem.62.8.2875-2882.1996 |
| [26] |
Moridshahi R, Bahreini M, Sharifmoghaddam M, Asoodeh A. Biochemical characterization of an alkaline surfactant-stable keratinase from a new keratinase producer, Bacillus zhangzhouensis[J]. Extremophiles, 2020, 24(5): 693-704. DOI:10.1007/s00792-020-01187-9 |
| [27] |
Yong B, Fei XT, Shao HH, Xu P, Hu YW, Ni WM, Xiao QJ, Tao X, He XY, Feng H. Recombinant expression and biochemical characterization of a novel keratinase BsKER71 from feather degrading bacterium Bacillus subtilis S1-4[J]. AMB Express, 2020, 10(1): 9. DOI:10.1186/s13568-019-0939-6 |
| [28] |
Hassan MA, Taha TH, Hamad GM, Hashem M, Alamri S, Mostafa YS. Biochemical characterisation and application of keratinase from Bacillus thuringiensis MT1 to enable valorisation of hair wastes through biosynthesis of vitamin B-complex[J]. International Journal of Biological Macromolecules, 2020, 153: 561-572. DOI:10.1016/j.ijbiomac.2020.03.032 |
| [29] |
Jaouadi B, Abdelmalek B, Fodil D, Ferradji FZ, Rekik H, Zaraî N, Bejar S. Purification and characterization of a thermostable keratinolytic serine alkaline proteinase from Streptomyces sp. strain AB1 with high stability in organic solvents[J]. Bioresource Technology, 2010, 101(21): 8361-8369. DOI:10.1016/j.biortech.2010.05.066 |
| [30] |
Riessen S, Antranikian G. Isolation of Thermoanaerobacter keratinophilus sp. nov., a novel thermophilic, anaerobic bacterium with keratinolytic activity[J]. Extremophiles, 2001, 5(6): 399-408. DOI:10.1007/s007920100209 |
| [31] |
Nandini A, Madhusudhan DN, Dayanand A. Enhanced production, purification and characterization of alkaline keratinase from Streptomyces minutiscleroticus DNA38[J]. International Letters of Natural Sciences, 2015, 43(4): 27-37. |
| [32] |
Ma YM, Ke X, Li XX, Shu WX, Yang WH, Liu YY, Yan X, Jia LH, Yan H. Expression and characterization of a keratinase encoding gene gm2886 in Streptomyces pactum ACT12 strain[J]. Chinese Journal of Biotechnology, 2017, 33(12): 1968-1978. (in Chinese) 马怡茗, 柯欣, 李晓霞, 舒伟学, 杨文翰, 刘亚勇, 颜霞, 贾良辉, 颜华. 角蛋白酶基因gm2886在密旋链霉菌ACT12中的表达及鉴定[J]. 生物工程学报, 2017, 33(12): 1968-1978. |
| [33] |
Anbu P, Gopinath SCB, Hilda A, Priya TL, Annadurai G. Purification of keratinase from poultry farm isolate-Scopulariopsis brevicaulis and statistical optimization of enzyme activity[J]. Enzyme and Microbial Technology, 2005, 36(5/6): 639-647. |
| [34] |
Ben Elhoul M, Zaraî Jaouadi N, Rekik H, Omrane Benmrad M, Mechri S, Moujehed E, Kourdali S, El Hattab M, Badis A, Bejar S, et al. Biochemical and molecular characterization of new keratinoytic protease from Actinomadura viridilutea DZ50[J]. International Journal of Biological Macromolecules, 2016, 92: 299-315. DOI:10.1016/j.ijbiomac.2016.07.009 |
| [35] |
Duarte TR, Oliveira SS, MacRae A, Cedrola SML, Mazotto AM, Souza EP, Melo ACN, Vermelho AB. Increased expression of keratinase and other peptidases by Candida parapsilosis mutants[J]. Brazilian Journal of Medical and Biological Research, 2011, 44(3): 212-216. DOI:10.1590/S0100-879X2011007500011 |
| [36] |
Yao B, Wang YR, Cao SS, Shi XY, Yuan TZ, Fan YL. Efficient, broad-spectrum keratinase and its gene: China, CN1405305A[P]. 2003-03-26 (in Chinese) 姚斌, 王亚茹, 操时树, 史秀云, 袁铁峥, 范云六. 一种高效、广谱角蛋白酶及其基因: 中国, CN1405305A[P]. 2003-03-26 |
| [37] |
Gradisar H, Friedrich J, Krizaj I, Jerala R. Similarities and specificities of fungal keratinolytic proteases: comparison of keratinases of Paecilomyces marquandii and Doratomyces microsporus to some known proteases[J]. Applied and Environmental Microbiology, 2005, 71(7): 3420-3426. DOI:10.1128/AEM.71.7.3420-3426.2005 |
| [38] |
Brandelli A, Daroit DJ, Riffel A. Biochemical features of microbial keratinases and their production and applications[J]. Applied Microbiology and Biotechnology, 2010, 85(6): 1735-1750. DOI:10.1007/s00253-009-2398-5 |
| [39] |
Riffel A, Lucas F, Heeb P, Brandelli A. Characterization of a new keratinolytic bacterium that completely degrades native feather keratin[J]. Archives of Microbiology, 2003, 179(4): 258-265. DOI:10.1007/s00203-003-0525-8 |
| [40] |
Zhu YX. Biochemical characterization and application of keratinase from keratin-degrading bacteria Bacillus strains[D]. Jinan: Master's Thesis of Shandong University, 2018 (in Chinese) 朱耀霞. 芽孢杆菌角蛋白降解菌及其胞外角蛋白酶的性质、应用研究[D]. 济南: 山东大学硕士学位论文, 2018 |
| [41] |
Tatineni R, Doddapaneni KK, Potumarthi RC, Vellanki RN, Kandathil MT, Kolli N, Mangamoori LN. Purification and characterization of an alkaline keratinase from Streptomyces sp.[J]. Bioresource Technology, 2008, 99(6): 1596-1602. DOI:10.1016/j.biortech.2007.04.019 |
| [42] |
Fang Z, Zhang J, Liu BH, Du GC, Chen J. Biochemical characterization of three keratinolytic enzymes from Stenotrophomonas maltophilia BBE11-1 for biodegrading keratin wastes[J]. International Biodeterioration & Biodegradation, 2013, 82: 166-172. |
| [43] |
Radha S, Gunasekaran P. Purification and characterization of keratinase from recombinant Pichia and Bacillus strains[J]. Protein Expression and Purification, 2009, 64(1): 24-31. DOI:10.1016/j.pep.2008.10.008 |
| [44] |
Yamamura S, Morita Y, Hasan Q, Yokoyama K, Tamiya E. Keratin degradation: a cooperative action of two enzymes from Stenotrophomonas sp.[J]. Biochemical and Biophysical Research Communications, 2002, 294(5): 1138-1143. DOI:10.1016/S0006-291X(02)00580-6 |
| [45] |
Lin X, Kelemen DW, Miller ES, Shih JC. Nucleotide sequence and expression of kerA, the gene encoding a keratinolytic protease of Bacillus licheniformis PWD-1[J]. Applied and Environmental Microbiology, 1995, 61(4): 1469-1474. DOI:10.1128/aem.61.4.1469-1474.1995 |
| [46] |
Li ZW, Liang S, Ke Y, Deng JJ, Zhang MS, Lu DL, Li JZ, Luo XC. The feather degradation mechanisms of a new Streptomyces sp. isolate SCUT-3[J]. Communications Biology, 2020, 3(1): 191. DOI:10.1038/s42003-020-0918-0 |
| [47] |
Jacobs M, Eliasson M, Uhlén M, Flock JI. Cloning, sequencing and expression of subtilisin Carlsberg from Bacillus licheniformis[J]. Nucleic Acids Research, 1985, 13(24): 8913-8926. DOI:10.1093/nar/13.24.8913 |
| [48] |
Stahl ML, Ferrari E. Replacement of the Bacillus subtilis subtilisin structural gene with an in vitro-derived deletion mutation[J]. Journal of Bacteriology, 1984, 158(2): 411-418. DOI:10.1128/jb.158.2.411-418.1984 |
| [49] |
Wells JA, Ferrari E, Henner DJ, Estell DA, Chen EY. Cloning, sequencing, and secretion of Bacillus amyloliquefaciens subtillisin in Bacillus subtilis[J]. Nucleic Acids Research, 1983, 11(22): 7911-7925. DOI:10.1093/nar/11.22.7911 |
| [50] |
Kitadokoro K, Tsuzuki H, Nakamura E, Sato T, Teraoka H. Purification, characterization, primary structure, crystallization and preliminary crystallographic study of a serine proteinase from Streptomyces fradiae ATCC 14544[J]. European Journal of Biochemistry, 1994, 220(1): 55-61. DOI:10.1111/j.1432-1033.1994.tb18598.x |
| [51] |
Mitsuiki S, Ichikawa M, Oka T, Sakai M, Moriyama Y, Sameshima Y, Goto M, Furukawa K. Molecular characterization of a keratinolytic enzyme from an alkaliphilic Nocardiopsis sp. TOA-1[J]. Enzyme and Microbial Technology, 2004, 34(5): 482-489. DOI:10.1016/j.enzmictec.2003.12.011 |
| [52] |
Kluskens LD, Voorhorst WG, Siezen RJ, Schwerdtfeger RM, Antranikian G, Van Der Oost J, De Vos WM. Molecular characterization of fervidolysin, a subtilisin-like serine protease from the thermophilic bacterium Fervidobacterium pennivorans[J]. Extremophiles, 2002, 6(3): 185-194. DOI:10.1007/s007920100239 |
| [53] |
Noronha EF, De Lima BD, De Sá CM, Roberto Felix C. Heterologous production of Aspergillus fumigatus keratinase in Pichia pastoris[J]. World Journal of Microbiology and Biotechnology, 2002, 18(6): 563-568. DOI:10.1023/A:1016341702908 |
| [54] |
Lin HH, Yin LJ, Jiang ST. Cloning, expression, and purification of Pseudomonas aeruginosa keratinase in Escherichia coli AD494(DE3)pLysS expression system[J]. Journal of Agricultural and Food Chemistry, 2009, 57(9): 3506-3511. DOI:10.1021/jf803752j |
| [55] |
Wu WL, Chen MY, Tu IF, Lin YC, EswarKumar N, Chen MY, Ho MC, Wu SH. The discovery of novel heat-stable keratinases from Meiothermus taiwanensis WR-220 and other extremophiles[J]. Scientific Reports, 2017, 7: 4658. DOI:10.1038/s41598-017-04723-4 |
| [56] |
Qiu JW, Wilkens C, Barrett K, Meyer AS. Microbial enzymes catalyzing keratin degradation: classification, structure, function[J]. Biotechnology Advances, 2020, 44: 107607. DOI:10.1016/j.biotechadv.2020.107607 |
| [57] |
Gupta R, Rajput R, Sharma R, Gupta N. Biotechnological applications and prospective market of microbial keratinases[J]. Applied Microbiology and Biotechnology, 2013, 97(23): 9931-9940. DOI:10.1007/s00253-013-5292-0 |
| [58] |
Zelena K, Eisele N, Berger RG. Escherichia coli as a production host for novel enzymes from basidiomycota[J]. Biotechnology Advances, 2014, 32(8): 1382-1395. DOI:10.1016/j.biotechadv.2014.08.006 |
| [59] |
Sharma R, Gupta R. Extracellular expression of keratinase Ker P from Pseudomonas aeruginosa in E. coli[J]. Biotechnology Letters, 2010, 32(12): 1863-1868. DOI:10.1007/s10529-010-0361-2 |
| [60] |
Zhang XY, Shen ZY, Duan XG, Li HX. The research progress on Bacillus expression system[J]. Genomics and Applied Biology, 2020, 39(7): 3110-3118. (in Chinese) 张心怡, 沈镇炎, 段绪果, 李慧星. 芽孢杆菌表达系统研究进展[J]. 基因组学与应用生物学, 2020, 39(7): 3110-3118. |
| [61] |
Lin X, Wong SL, Miller ES, Shih JCH. Expression of the Bacillus licheniformis PWD-1 keratinase gene in B. subtilis[J]. Journal of Industrial Microbiology and Biotechnology, 1997, 19(2): 134-138. DOI:10.1038/sj.jim.2900440 |
| [62] |
Yang ZL, Zhang ZS. Engineering strategies for enhanced production of protein and bio-products in Pichia pastoris: a review[J]. Biotechnology Advances, 2018, 36(1): 182-195. DOI:10.1016/j.biotechadv.2017.11.002 |
| [63] |
Hu H, Gao J, He J, Yu B, Zheng P, Huang ZQ, Mao XB, Yu J, Han GQ, Chen DW. Codon optimization significantly improves the expression level of a keratinase gene in Pichia pastoris[J]. PLoS One, 2013, 8(3): e58393. DOI:10.1371/journal.pone.0058393 |
| [64] |
Huang MR, Chen RA, Ren GC. Secretory expression and purification of Bacillus licheniformis keratinase in insect cells[J]. PLoS One, 2017, 12(8): e0183764. DOI:10.1371/journal.pone.0183764 |
| [65] |
Brandelli A. Bacterial keratinases: useful enzymes for bioprocessing agroindustrial wastes and beyond[J]. Food and Bioprocess Technology, 2008, 1(2): 105-116. DOI:10.1007/s11947-007-0025-y |
| [66] |
Bertsch A, Coello N. A biotechnological process for treatment and recycling poultry feathers as a feed ingredient[J]. Bioresource Technology, 2005, 96(15): 1703-1708. DOI:10.1016/j.biortech.2004.12.026 |
| [67] |
Gupta R, Sharma R, Beg QK. Revisiting microbial keratinases: next generation proteases for sustainable biotechnology[J]. Critical Reviews in Biotechnology, 2013, 33(2): 216-228. DOI:10.3109/07388551.2012.685051 |
| [68] |
Gurav RG, Jadhav JP. A novel source of biofertilizer from feather biomass for banana cultivation[J]. Environmental Science and Pollution Research, 2013, 20(7): 4532-4539. DOI:10.1007/s11356-012-1405-z |
| [69] |
Bálint B, Bagi Z, Tóth A, Rákhely G, Perei K, Kovács KL. Utilization of keratin-containing biowaste to produce biohydrogen[J]. Applied Microbiology and Biotechnology, 2005, 69(4): 404-410. DOI:10.1007/s00253-005-1993-3 |
| [70] |
Schommer VA, Wenzel BM, Daroit DJ. Anaerobic co-digestion of swine manure and chicken feathers: effects of manure maturation and microbial pretreatment of feathers on methane production[J]. Renewable Energy, 2020, 152: 1284-1291. DOI:10.1016/j.renene.2020.01.154 |
| [71] |
Ramakrishna Reddy M, Sathi Reddy K, Ranjita Chouhan Y, Bee H, Reddy G. Effective feather degradation and keratinase production by Bacillus pumilus GRK for its application as bio-detergent additive[J]. Bioresource Technology, 2017, 243: 254-263. DOI:10.1016/j.biortech.2017.06.067 |
| [72] |
Paul T, Das A, Mandal A, Jana A, Maity C, Adak A, Halder SK, DasMohapatra PK, Pati BR, Mondal KC. Effective dehairing properties of keratinase from Paenibacillus woosongensis TKB2 obtained under solid state fermentation[J]. Waste and Biomass Valorization, 2014, 5(1): 97-107. DOI:10.1007/s12649-013-9217-z |
| [73] |
Wang P, Cui L, Wang Q, Fan XR, Zhao XF, Wu J. Combined use of mild oxidation and cutinase/lipase pretreatments for enzymatic processing of wool fabrics[J]. Engineering in Life Sciences, 2010, 10(1): 19-25. DOI:10.1002/elsc.200900041 |
| [74] |
Zhang R, Ling XN, Li KT. Research progress in the mechanism of keratin degradation by microorganisms and the application of keratinase[J]. Biological Disaster Science, 2020, 43(4): 331-337. (in Chinese) 张荣, 凌晓宁, 李昆太. 微生物降解角蛋白机制及角蛋白酶应用研究进展[J]. 生物灾害科学, 2020, 43(4): 331-337. DOI:10.3969/j.issn.2095-3704.2020.04.61 |
| [75] |
Cao L, Tan H, Liu Y, Xue X, Zhou S. Characterization of a new keratinolytic Trichoderma atroviride strain F6 that completely degrades native chicken feather[J]. Letters in Applied Microbiology, 2008, 46(3): 389-394. DOI:10.1111/j.1472-765X.2008.02327.x |
| [76] |
Tian JW, Long XF, Tian YQ, Shi B. Enhanced extracellular recombinant keratinase activity in Bacillus subtilis SCK6 through signal peptide optimization and site-directed mutagenesis[J]. RSC Advances, 2019, 9(57): 33337-33344. DOI:10.1039/C9RA07866E |
| [77] |
Fu G, Liu JL, Li JS, Zhu BW, Zhang DW. Systematic screening of optimal signal peptides for secretory production of heterologous proteins in Bacillus subtilis[J]. Journal of Agricultural and Food Chemistry, 2018, 66(50): 13141-13151. DOI:10.1021/acs.jafc.8b04183 |
| [78] |
Su C, Gong JS, Sun YX, Qin JF, Zhai S, Li H, Li H, Lu ZM, Xu ZH, Shi JS. Combining pro-peptide engineering and multisite saturation mutagenesis to improve the catalytic potential of keratinase[J]. ACS Synthetic Biology, 2019, 8(2): 425-433. DOI:10.1021/acssynbio.8b00442 |
| [79] |
Li JX, Chen DD, Yu ZQ, Zhao LM, Zhang RJ. Improvement of expression level of keratinase Sfp2 from Streptomyces fradiae by site-directed mutagenesis of its N-terminal pro-sequence[J]. Biotechnology Letters, 2013, 35(5): 743-749. DOI:10.1007/s10529-013-1139-0 |
| [80] |
Fang Z, Zhang J, Liu BH, Du GC, Chen J. Enhancement of the catalytic efficiency and thermostability of Stenotrophomonas sp. keratinase KerSMD by domain exchange with KerSMF[J]. Microbial Biotechnology, 2016, 9(1): 35-46. DOI:10.1111/1751-7915.12300 |
| [81] |
Su C, Gong JS, Qin JF, Li H, Li H, Xu ZH, Shi JS. The tale of a versatile enzyme: molecular insights into keratinase for its industrial dissemination[J]. Biotechnology Advances, 2020, 45: 107655. DOI:10.1016/j.biotechadv.2020.107655 |
 2021, Vol. 48
2021, Vol. 48