
扩展功能
文章信息
- 段亮杰, 沙雨婷, 罗意, 夏小乐
- DUAN Liang-Jie, SHA Yu-Ting, LUO Yi, XIA Xiao-Le
- 叠氮溴化丙锭-荧光定量PCR法实时快速检测5种乳杆菌活菌数方法的建立与应用
- Quantitative PCR combined with propidium monoazide treatment for real-time and rapid determination of five viable Lactobacillus
- 微生物学通报, 2020, 47(12): 4317-4327
- Microbiology China, 2020, 47(12): 4317-4327
- DOI: 10.13344/j.microbiol.china.200050
-
文章历史
- 收稿日期: 2020-01-17
- 接受日期: 2020-03-09
- 网络首发日期: 2020-04-26
2. 江南大学生物工程学院 江苏 无锡 214122
2. School of Biotechnology, Jiangnan University, Wuxi, Jiangsu 214122, China
乳杆菌属(Lactobacillus)是一类革兰氏阳性细菌,广泛存在于食品、饲料、植物、脊椎动物、无脊椎动物和人类中[1]。由于其出色的发酵性能和“益生”特性[2]而被广泛应用于发酵食品领域。一方面乳杆菌产生的有机酸、胞外多糖、γ-氨基丁酸等物质极大地增强了发酵食品的风味和保健功能;另一方面乳杆菌是导致食品发生腐败变质、影响食品安全的主要微生物之一[3-4]。与大多数发酵食品一致,黄酒具有丰富的微生物群落,而乳杆菌的占比很大(约占细菌总量的0.59%-65.17%)[5]。丰富的乳杆菌群落与复杂的发酵环境共同作用,最终形成了黄酒独特的风味与品质。因此,解析乳杆菌在黄酒乃至其他发酵食品中的实时组成情况,尤其是检测乳杆菌的实时活菌数对于发酵食品的安全制造及新型发酵食品的开发都具有重要意义。
目前已经建立了如基因组测序[6]、荧光原位杂交[7]、末端限制性片段长度多态性分析(terminal- restriction fragment length polymorphism,T-RFLP)[8]、磷脂脂肪酸检测(phospholipid fatty acid,PLFA)[9]、荧光定量PCR (quantitative PCR,qPCR)[10]等多种非培养的乳杆菌检测方法。其中,荧光定量PCR是一种基于特异性引物定量检测微生物的方法,因其具有快速、灵敏、特异等特点而被广泛应用于各种样品中微生物的检测[10-12]。但传统qPCR既能扩增活菌DNA,又能扩增死菌DNA,无法区分活细胞与死细胞。反转录荧光定量PCR (reverse transcription qPCR,RT-qPCR)虽然能够达到检测活菌的效果,但因RNA易降解等特性而不适宜于复杂样品中微生物的检测。叠氮溴化丙锭(propidium monoazide,PMA)是一种常见的活菌染料,能够穿透死亡细胞/膜损伤细胞的细胞膜插入核酸,在强光照射下能够与DNA形成稳定的共价氮碳键不可逆地修饰DNA,阻止死细胞/膜损伤细胞的DNA进行PCR扩增[13]。PMA耦合qPCR不仅能够选择性地检测目标微生物的活菌数,而且具有稳定、快速、灵敏等特点,成为了检测样品中乳杆菌活菌数的有利方法。尽管PMA耦合qPCR在微生物活菌检测中已有广泛应用[10-11, 14-15],但不同微生物对PMA的敏感度不同[16],因此应当针对乳杆菌建立特定的PMA处理方式,以获得更为准确的活菌检测结果。
本研究以发酵食品中常见的5种乳杆菌为目标菌株,筛选得到高特异性乳杆菌引物,通过PMA-qPCR检测,建立一种实时定量检测5种不同乳杆菌活菌数的特异性方法,并将其应用于黄酒酿造过程。该方法对于其他样品中乳杆菌乃至乳酸菌的种水平的实时定量活菌数检测也具有一定的参考价值。
1 材料与方法 1.1 材料 1.1.1 黄酒样品实验用黄酒发酵醪液全部取自江苏某黄酒厂,黄酒酿造工艺和取样参考文献[4]。
1.1.2 主要试剂和仪器及培养基叠氮溴化丙锭(PMA),Biotium公司;细菌基因组DNA提取试剂盒(TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit),宝生物工程(大连)有限公司;DNA提取试剂盒(Fast DNA® Spin Kit for Soil),MP Biomedicals公司;ChamQ Universal SYBR qPCR Master Mix,南京诺唯赞生物科技股份有限公司。650 W卤素灯,OSRAM公司;实时荧光定量基因扩增仪,Bio-Rad公司;核酸与蛋白分析仪,赛默飞世尔科技公司。MRS肉汤培养基,北京索莱宝科技有限公司;MSA培养基(g/L):牛肉膏1.0,蛋白胨10.0,甘露糖醇10.0,氯化钠75.0,pH 7.4;GY培养基(g/L):葡萄糖10.0,酵母提取物10.0,pH自然;LB培养基(g/L):蛋白胨10.0,酵母粉5.0,氯化钠10.0,pH自然。
1.1.3 菌株及培养方式植物乳杆菌(Lactobacillus plantarum) ACBC271购自酿造微生物应用中心,短乳杆菌(Lactobacillus brevis) ATCC367和巴氏醋酸杆菌(Acetobacter pasteurianus) ATCC33445购自美国菌种保藏中心,嗜酸乳杆菌(Lactobacillus acidophilus) CICC20244和乳杆菌(Lactobacillus sp.) CICC6251购自中国工业微生物菌种保藏中心,木糖葡萄球菌(Staphylococcus xylosus) CGMCC1.8382购自中国普通微生物菌种保藏管理中心;大肠杆菌(Escherichia coli) JM109由本实验室提供;巴氏醋酸杆菌(Acetobacter pasteurianus) CICIM B7003分离自酿造工厂,发酵乳杆菌(Lactobacillus fermentum)、干酪乳杆菌(Lactobacillus casei)、4株乳杆菌(Lactobacillus sp.)和10株肠球菌(Enterococcus spp.)分离自黄酒发酵醪液,4株芽孢杆菌(Bacillus spp.)分离自豆瓣酱;以上菌株由本实验室保存。乳杆菌和肠球菌采用MRS肉汤培养基37 ℃静置培养,木糖葡萄球菌采用MSA培养基37 ℃静置培养,醋酸杆菌采用GY培养基30 ℃、180 r/min振荡培养,芽孢杆菌和大肠杆菌采用LB培养基37 ℃、180 r/min振荡培养。
1.2 方法 1.2.1 基因组DNA的提取微生物纯培养物基因组DNA按照TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit产品说明书进行提取,黄酒发酵醪液微生物基因组DNA按照Fast DNA® Spin Kit for Soil产品说明书提取。所有基因组DNA通过核酸与蛋白分析仪检测其纯度与浓度后置于-20 ℃保存待用。
1.2.2 PMA处理与优化参考Gobert等[17]的方法制作热致死乳杆菌,具体方法:将乳杆菌在MRS培养基中37 ℃静置培养24 h后,用无菌生理盐水洗涤并稀释至104 CFU/mL,作为活菌菌液记为V4。另取稀释至107 CFU/mL的菌液于90 ℃水浴15 min (通过平板计数确保没有活菌),作为死菌菌液记为D7。取适量活菌菌液与死菌菌液混合作为待测样品,调整其浊度一致,记为V4+D7。
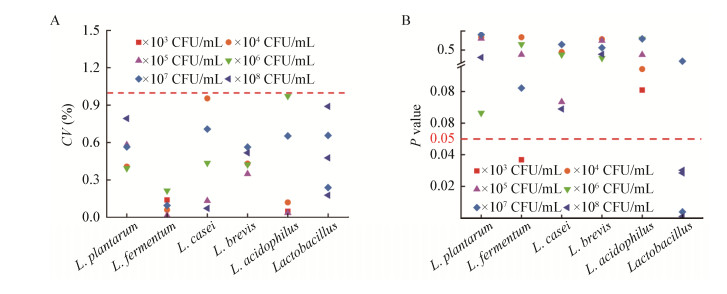
在待测样品中加入PMA,调整其终浓度为5、10、20、30、50 μmol/L,分别在黑暗环境反应5、10、15、20、25、30 min,然后在650 W卤素灯下照射5、10、15、20、25、30 min (样品置于冰上,距离灯管20 cm)后,室温、12 000 r/min离心2 min用于后续基因组提取,通过qPCR检测其对死菌DNA的抑制率,计算公式如下:
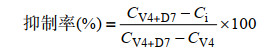
|
CV4+D7:待测样品未经PMA处理的菌落浓度,CFU/mL;CV4:V4样品的菌落浓度,CFU/mL;Ci:待测样品经PMA处理后的菌落浓度,CFU/mL。
1.2.3 引物获取及特异性检测所有引物均从已发表的文献中获取,引物来源及详细信息可见表 1,引物由生工生物工程(上海)股份有限公司合成。通过NCBI数据库、RDP数据库和TestPrime (https://www.arb-silva.de/search/testprime/)检索引物特异性,然后通过Oligo 7 (Molecular Biology Insights, USA)、silico PCR (http://insilico.ehu.es/PCR/)和Serial Cloner 2.6 (serialbasics.free.fr/Serial-Cloner.heml)模拟PCR并对引物评价分析。以1.1.3中的28株细菌为目标菌株,提取基因组后通过qPCR来验证引物特异性。
| 序号 No. |
引物名称 Primers name |
引物序列 Primers sequence (5′→3′) |
目标菌株 Target strains |
大小 Size (bp) |
参考文献 References |
| 1 | LactoF | TGGAAACAGRTGCTAATACCG | Lactobacillus | 233 | [18] |
| LactoR | GTCCATTGTGGAAGATTCCC | ||||
| 2 | LPrecAF | GTGGTGCGGTCGATATTTTAGTT | Lactobacillus plantarum | 108 | [19] |
| LPrecAR | TCAGCCGCGCTTGTAACC | ||||
| 3 | LFermF | GCACCTGATTGATTTTGGTCG | Lactobacillus fermentum | 103 | [20] |
| LFermR | GGTATTAGCATCTGTTTCCAAATG | ||||
| 4 | s-Lbre-F | ATTTTGTTTGAAAGGTGGCTTCGG | Lactobacillus brevis | 289 | [21] |
| s-Lbre-R | ACCCTTGAACAGTTACTCTCAAAGG | ||||
| 5 | Acidfor | AGCGAGCTGAACCAACAGAT | Lactobacillus acidophilus | 227 | [22] |
| Acidrev | AGGCCGTTACCCTACCAACT | ||||
| 6 | LcaseF | GCACCGAGATTCAACATGG | Lactobacillus casei | 117 | [18] |
| LcaseR | GGTTCTTGGATYTATGCGGTATTAG |
qPCR反应体系:ChamQ Universal SYBR qPCR Master Mix 10 μL,上、下游引物(10 μmol/L)各0.4 μL,DNA模板1.5 μL,添加双蒸水补足体积至20 μL。
qPCR反应条件:95 ℃ 3 min;95 ℃ 10 s,60 ℃ 30 s (收集荧光),共39个循环。循环结束后进入熔解曲线分析,程序为:65 ℃逐步升温至95 ℃,每升温0.5 ℃检测一次荧光信号,每次检测持续5 s。
1.2.5 标准曲线的建立乳杆菌在MRS培养基中37 ℃静置培养24 h后通过平板计数法测定原始浓度,同时用无菌生理盐水洗涤2-4次后按照1.2.1的方法将提取的微生物基因组DNA以10倍梯度进行稀释,通过qPCR测定对应的Cq值,建立菌落浓度(log10CFU/mL)与Cq值之间的线性关系,并通过公式E=10-1/s-1计算扩增效率,其中s为标准曲线的斜率。
1.2.6 qPCR方法的重复性实验及其与平板计数法的对比按照1.1.3的方式活化5种乳杆菌(Lactobacillus plantarum,Lactobacillus fermentum,Lactobacillus brevis,Lactobacillus acidophilus,Lactobacillus casei),用无菌生理盐水洗涤并梯度稀释,选取不同稀释倍数的菌液通过PMA-qPCR检测菌落浓度,重复3次,计算Cq值的变异系数,对该方法的稳定性进行评价。同时使用平板计数法检测5种乳杆菌的含量,通过配对样本t检验对比两种检测方法检测结果的差异,评估该方法的可靠性。
1.2.7 PMA-qPCR检测法在黄酒酿造中的应用黄酒发酵醪液经无菌生理盐水洗涤2-4次后重悬,调整其浊度与1.2.2微生物纯培养物的浊度一致,然后用最优的PMA处理条件进行处理,按照1.2.1的方法提取微生物基因组DNA,通过qPCR检测5种乳杆菌的活菌数。
1.3 数据分析所有实验均重复3次,数据结果以平均数±标准差表示。利用Excel 2016计算PMA-qPCR重复性实验的变异系数CV。PMA-qPCR检测法与平板计数检测法对同一样本检测结果的显著性差异分析通过Origin 8.0的配对样本t检验完成。
2 结果与分析 2.1 PMA处理条件的优化如图 1A所示,微量的PMA (5 μmol/L)已经能够抑制84.83%的死菌DNA扩增,随着PMA浓度的增加,死菌抑制率得到了进一步的提高;当浓度提升至20 μmol/L时,99.84%的死菌DNA与PMA结合而无法被扩增,随后PMA浓度的增加不再显著增加死菌抑制率,因此PMA最佳处理浓度为20 μmol/L。如图 1B所示,PMA具有相对“温和”的细胞膜渗透能力,暗孵育时间的增加并不会使PMA穿透活菌细胞膜造成假阴性结果,为提高检测结果的精确性,可将暗孵育时间设置为15 min,此时PMA对死菌DNA的抑制率达到了99.89%;当曝光时间分别为5 min和10 min时,死菌DNA与PMA交联不彻底,仍有41.76%和7.09%的死菌DNA未与PMA结合表现出PCR扩增能力;当曝光时间增加到15 min后,99.84%的死菌DNA被PMA抑制,达到了较好的活菌检测效果;随着曝光时间的继续增加,PMA对死菌DNA的抑制率不再继续上升,因此15 min的曝光时间对于死菌DNA信号的抑制是充分的。
|
| 图 1 PMA优化结果 Figure 1 Results of PMA treatment 注:A:PMA浓度对死菌DNA的抑制率;B:暗孵育时间和曝光时间对死菌DNA的抑制率. Note: A: Inhibiting effect of PMA concentration on DNA of non-viable bacteria; B: Inhibiting effect of incubation and photoactivation time on DNA of non-viable bacteria. |
|
|
减少或消除引物的非特异性结合是提高qPCR法检测生物量准确度的关键[14]。通过NCBI、RDP数据库和TestPrime检索证明了引物理论上的特异性,进一步通过qPCR实验对引物的特异性进行检测,结果见表 2。与乳杆菌亲缘关系较远的醋酸杆菌、芽孢杆菌和大肠杆菌与6对乳杆菌引物均表现出了阴性qPCR结果。以购买到的4株乳杆菌和从黄酒发酵醪液中筛选到的4株Lactobacillus sp.与10株Enterococcus spp.作为与目标微生物亲缘关系较近的菌种库,对引物特异性进行进一步验证,结果显示植物乳杆菌引物(LPrecAF/LPrecAR)、发酵乳杆菌引物(LFermF/ LFermR)、短乳杆菌引物(s-Lbre-F/s-Lbre-R)、嗜酸乳杆菌引物(Acidfor/Acidrev)和干酪乳杆菌引物(LcaseF/ LcaseR)具有较高的特异性,只与目标微生物特异性结合,熔解曲线单一。但实验发现基于16S rRNA基因设计的乳杆菌属引物(LactoF/LactoR)不仅能与10株乳杆菌结合,也能与8株肠球菌结合表现出扩增信号。肠球菌作为乳酸菌的一种,与乳杆菌具有较近的亲缘关系,在16S rRNA基因上具有较高的相似性[23],这使得基于16S rRNA基因设计的乳杆菌属引物无法准确地区分两个属,揭示这对引物只适用于肠球菌属含量较少样本中乳杆菌的定量检测。
| 菌种Species | 菌株Strains | 数量Amounts | 特异性检测Specificity tests | |||||
| LactoF/ LactoR | LPrecAF/ LPrecAR | LFermF/ LFermR | s-Lbre-F/ s-Lbre-R | Acidfor/ Acidrev | LcaseF/ LcaseR | |||
| Lactobacillus plantarum | ACBC271 | 1 | + | + | - | - | - | - |
| Lactobacillus brevis | ATCC367 | 1 | + | - | - | + | - | - |
| Lactobacillus fermentum | Laboratory strain | 1 | + | - | + | - | - | - |
| Lactobacillus acidophilus | CICC20244 | 1 | + | - | - | - | + | - |
| Lactobacillus casei | Laboratory strain | 1 | + | - | - | - | - | + |
| Lactobacillus sp. | Laboratory strain | 4 | + | - | - | - | - | - |
| Lactobacillus sp. | CICC6251 | 1 | + | - | - | - | - | - |
| Enterococcus spp. | Laboratory strain | 10 | 8/10a | - | - | - | - | - |
| Acetobacter pasteurianus | CICIM B7003 | 1 | - | - | - | - | - | - |
| Acetobacter pasteurianus | ATCC33445 | 1 | - | - | - | - | - | - |
| Bacillus spp. | Laboratory strain | 4 | - | - | - | - | - | - |
| Staphylococcus xylosus | CGMCC1.8382 | 1 | - | - | - | - | - | - |
| Escherichia coli | JM109 | 1 | - | - | - | - | - | - |
| 注:+:阳性扩增结果;-:阴性扩增结果;a:10株菌中有8株表现出阳性扩增结果.
Note: +: The positive amplification result; -: The negative amplification result; a: 8 of the 10 strains showed positive amplification results. | ||||||||
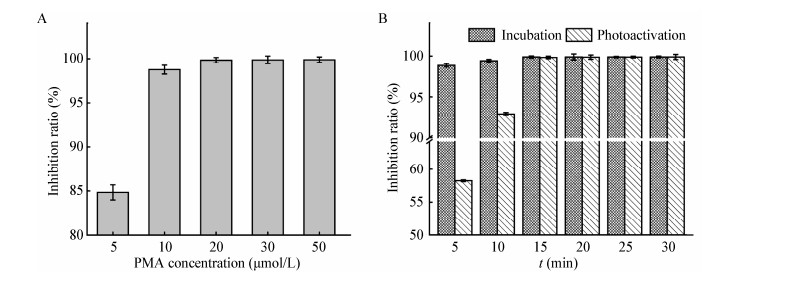
如图 2和表 3所示,qPCR扩增信号Cq值与菌落浓度的对数值(log10CFU/mL)之间具有良好的线性关系(R2 > 0.98),优化扩增体系与条件,扩增效率达到了90%-103%。检测限(limit of detection,LOD)通过公式Cq (LOD)=Cq (NTC)–3[10]来计算,不同乳杆菌的检测限在101.8-103.2 CFU/mL之间波动。其中嗜酸乳杆菌的限最低,为101.8 CFU/mL,具有最高的灵敏度。发酵乳杆菌的检测限相对较高,达到了103.2 CFU/mL。PMA-qPCR法的样品制备过程中可能造成目标微生物基因组的丢失,造成检测限的提高[14]。
|
| 图 2 不同乳杆菌的PMA-qPCR标准曲线 Figure 2 Standard curves of Lactobacillus constructed by PMA-qPCR 注:A:乳杆菌属;B:植物乳杆菌;C:发酵乳杆菌;D:短乳杆菌;E:嗜酸乳杆菌;F:干酪乳杆菌. Note: A: Lactobacillus; B: Lactobacillus plantarum; C: Lactobacillus fermentum; D: Lactobacillus brevis; E: Lactobacillus acidophilus; F: Lactobacillus casei. |
|
|
| 种类 Species |
扩增效率 Efficiency (%) |
检测限 Limit of detection (log10CFU/mL) |
| Lactobacillus | 90.4 | 2.6 |
| Lactobacillus plantarum | 102.1 | 2.1 |
| Lactobacillus fermentum | 94.3 | 3.2 |
| Lactobacillus brevis | 90.4 | 2.9 |
| Lactobacillus acidophilus | 90.6 | 1.8 |
| Lactobacillus casei | 92.4 | 2.9 |
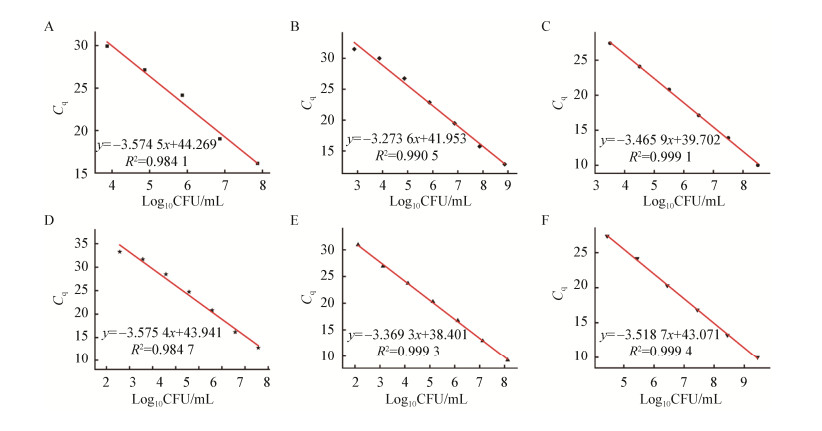
按照1.2.6的方法评估PMA-qPCR法检测乳杆菌含量的可靠性,结果如图 3A所示,Cq值的变异系数均小于1%,表明在实验浓度范围内,该方法具有良好的重复性。t检验证明在103-108 CFU/mL范围内,PMA-qPCR与平板计数法测得的不同乳杆菌的含量在统计学上没有显著性差异(P > 0.05) (Lactobacillus fermentum为103 CFU/mL时和Lactobacillus除外) (图 3B)。当样品中发酵乳杆菌的含量接近检测限103.2 CFU/mL时,PMA-qPCR的检测准确性出现降低(P < 0.05),尽管如此,其检测结果仍与平板计数检测结果处于同一数量级(表 4)。而较差的乳杆菌属定量检测结果反映了以16S rRNA基因为靶基因区域定量检测的局限性,这与16S rRNA基因在不同乳杆菌基因组DNA中的拷贝数不同有关。通过拷贝数矫正微生物组的丰度对于样品中微生物检测具有重要意义,文献[24]表明通过rrnDB数据库(https://rrndb.umms.med.umich.edu/)可以查找细菌和古菌的16S rRNA基因拷贝数,进而矫正相应拷贝数。
|
| 图 3 PMA-qPCR法的可靠性检测 Figure 3 Reliability of PMA-qPCR 注:A:变异系数;B:显著性检验. Note: A: Variation coefficients; B: Significance test. |
|
|
| 菌种 Species |
检测类型 Type |
Log10CFU/mL | ||||
| 组1 Group 1 |
组2 Group 2 |
组3 Group 3 |
组4 Group 4 |
组5 Group 5 |
||
| Lactobacillus plantarum | Plate count | 4.27±0.04 | 8.27±0.04 | 7.27±0.04 | 6.27±0.04 | 5.27±0.04 |
| PMA-qPCR | 4.25±0.11 | 8.32±0.12 | 7.27±0.10 | 6.21±0.09 | 5.27±0.14 | |
| Lactobacillus fermentum | Plate count | 7.51±0.11 | 6.51±0.11 | 5.51±0.11 | 4.51±0.11 | 3.51±0.11 |
| PMA-qPCR | 7.35±0.01 | 6.44±0.04 | 5.45±0.00 | 4.50±0.01 | 3.65±0.04 | |
| Lactobacillus casei | Plate count | 5.54±0.07 | 4.54±0.07 | 8.54±0.07 | 7.54±0.07 | 6.54±0.07 |
| PMA-qPCR | 5.46±0.03 | 4.53±0.04 | 8.42±0.01 | 7.51±0.12 | 6.48±0.09 | |
| Lactobacillus brevis | Plate count | 7.16±0.01 | 6.16±0.01 | 5.16±0.01 | 4.16±0.01 | 8.16±0.01 |
| PMA-qPCR | 7.19±0.10 | 6.10±0.09 | 5.17±0.09 | 4.13±0.13 | 8.19±0.08 | |
| Lactobacillus acidophilus | Plate count | 5.40±0.03 | 4.40±0.03 | 3.40±0.03 | 7.40±0.03 | 6.40±0.03 |
| PMA-qPCR | 5.37±0.01 | 4.43±0.03 | 3.44±0.01 | 7.38±0.08 | 6.36±0.15 | |
| Lactobacillus | Plate count | 7.67±0.07 | 8.28±0.04 | 8.56±0.06 | 7.79±0.04 | 8.18±0.01 |
| PMA-qPCR | 7.83±0.11 | 8.65±0.12 | 8.86±0.02 | 8.45±0.03 | 8.36±0.07 | |
黄酒是一种典型的多菌种混合发酵食品,具有复杂的发酵体系和微生物群落,检测其中的微生物组成,尤其是在种水平上检测乳杆菌的含量具有一定难度。本研究使用建立好的PMA-qPCR检测法在种水平上对黄酒酿造过程中5种乳杆菌的活菌数进行了定量检测。如图 4所示,随着发酵时间的延长,黄酒发酵醪液中总乳杆菌的含量从1.2×106 CFU/mL逐渐降低至1.1×105 CFU/mL,与实验室前期高通量测序测得的乳杆菌含量变化趋势[5]相似,较低的乳杆菌含量可能与较低的核酸提取率有关(17.3- 35.7 ng/μL)。发酵乳杆菌是黄酒中的优势乳杆菌,占总乳杆菌的25%-45%,尤其是在第3天时,发酵乳杆菌的含量达到了4.95×105 CFU/mL。干酪乳杆菌和短乳杆菌的含量虽然随着发酵时间的延长在不断下降,但其在乳杆菌属中的占比却在不断上升,在发酵第18天时,干酪乳杆菌和短乳杆菌分别占比33.76%和27.93%。植物乳杆菌和嗜酸乳杆菌在黄酒酿造过程中具有较低的含量,仅植物乳杆菌在第3天和第4天具有103.6 CFU/mL和102.9 CFU/mL,而在其他时间点的含量均低于检测限。王然然等[25]通过分离培养的方式从黄酒发酵醪液中筛选到了58株乳杆菌,通过菌种鉴定发现其中34株是发酵乳杆菌,占有绝对优势。Yu等利用PCR-DGGE方法分析了黄酒酿造过程中细菌群落多样性,发现发酵乳杆菌、短乳杆菌和植物乳杆菌在整个发酵过程中都具有较高的丰度[26],与本文研究结果相似。Lv等采用PMA-qPCR法检测了红曲黄酒中植物乳杆菌的含量,发酵开始阶段(1-5 d)植物乳杆菌的含量约为102.3-102.6 CFU/mL,随后其含量逐渐上升至约106.7 CFU/mL (30 d),到发酵结束其含量又下降至约106 CFU/mL (45 d)[10],与本研究结果具有一定差异。酿造环境及工艺的不同可能是导致乳杆菌含量及变化差异的主要原因。
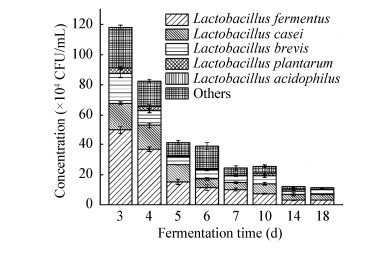
|
| 图 4 PMA-qPCR法检测黄酒酿造过程5种乳杆菌的含量 Figure 4 Quantitative analysis of 5 kinds of Lactobacillus during Chinese rice wine fermentation by PMA-qPCR |
|
|
活细菌不仅包括能够在传统培养基上生长并形成菌落的细菌,还包括大量具有活性但不可培养的细菌(viable but nonculturable,VBNC)[27]。VBNC状态的细菌虽然失去了在传统培养基上生长繁殖的能力,但依然具有细胞膜完整性和代谢活性[27],对于发酵的正常进行具有重要作用。细胞膜的完整与否是PMA鉴别活/死细胞的理论基础,完整的细胞膜能够帮助VBNC状态细菌抵抗PMA的侵入,有效提升了活菌检出率。但同时一些研究[13]表明PMA对细胞膜的渗透性相对“温和”,尤其是对于一些细胞膜轻微损伤的细菌,PMA难以透过细胞膜与DNA结合。通过优化PMA处理条件或适量添加促进物质(如脱氧胆酸钠、二甲亚砜和乙二胺四乙酸等)能够增强PMA对死亡细胞/膜损伤细胞细胞膜的渗透性[13],提高检测准确性。本研究针对5种乳杆菌,优化了包括PMA浓度、暗孵育时间及曝光时间在内的3个主要参数,最终抑制了99.89%的死菌DNA扩增,保证了活菌检测准确性。尽管如此,还有诸如细菌丰度及组成、活细胞与死细胞之间的比例及样品盐含量等因素[13, 28]可能影响PMA处理的有效性。不同微生物对于PMA的敏感度有所差异,Lai等[28]研究发现曝光时间从5 min增加到15 min能够显著增加PMA对Lactobacillus gasseri死菌的抑制效果,而不改变对Lactobacillus salivarius的处理效果,这可能是两种乳杆菌细胞膜组成的差异造成的。Lv等研究发现随着PMA浓度、暗孵育时间和曝光时间的增加,PMA对Lactobacillus plantarum死菌的抑制率逐渐增加,达到最佳条件后,浓度/时间的增加不再显著改善PMA对死菌的抑制效果[10],与本文研究结果相似。根据不同微生物采取不同的PMA处理方式能够获得更准确的检测结果[16]。
为了能够在种水平上区分不同的乳杆菌,我们检索文献及数据库,最终筛选出了5对高特异性的引物。其中4对(LFermF/LFermR、s-Lbre-F/s-Lbre-R、Acidfor/Acidrev、LcaseF/LcaseR)是基于16S rRNA基因设计的。16S rRNA基因是最常用于细菌鉴定与检测的核酸序列,也是qPCR法检测细菌含量最常用的区域。合理利用16S rRNA基因的不同分区[29],能够较准确地区分不同细菌。此外,基于管家基因recA设计的植物乳杆菌qPCR引物(LPrecAF/LPrecAR)也表现出了良好的特异性,表明用于qPCR法测生物量的引物不应局限于16S rRNA基因。大量基于16S-23S rRNA基因间区[30]、23S rRNA基因[31]以及各种管家基因[19]的引物被设计出来并用于细菌的检测,这也使得在菌种水平,甚至是菌株水平上检测某种微生物的含量成为了可能。根据不同微生物找到其最区别于其他微生物的靶基因区域,设计出高度特异的引物是未来的发展趋势。
基因组拷贝数与菌落浓度之间一一对应的关系使得可以通过基因组拷贝数反映菌落浓度。我们通过梯度稀释目标微生物基因组DNA,构建了扩增信号Cq值与菌落浓度(log10CFU/mL)之间的标准曲线,展现出了良好的线性关系(R2 > 0.98)。同时PMA-qPCR与平板计数法的检测结果在统计学上没有显著性(P > 0.05),且Cq值的变异系数小于1%,证明了该方法的稳定可靠性。5种乳杆菌的定量检测限如表 3所示,其变化范围从101.8 CFU/mL到103.2 CFU/mL,较高的检测限可能与样品制备过程中DNA的丢失有关。事实上,qPCR本身的检测限非常低(每个反应中含有1-10个目标基因即可),在100%扩增效率的前提下,其对微生物的检测限是12-120 CFU/mL[14]。为了提高检测的灵敏度,可以考虑将PMA处理与DNA提取融合到一个程序中,减少前处理的步骤,降低DNA的损失[15]。
我们建立的PMA-qPCR法在黄酒中乳杆菌活菌数检测中的成功应用,展现了其在检测活菌绝对含量中的巨大潜力。一方面,PMA-qPCR法克服了基因组测序和PCR-DGGE无法检测微生物绝对含量的缺点;另一方面,与传统的平板计数法相比,PMA-qPCR省去了1-2 d的细菌培养时间,使得整个检测过程能够在3 h内完成,而且能够检测到传统的平板计数法无法检测到的VBNC状态细菌,获得更为准确的活菌检测结果。PMA-qPCR方法的建立不仅能准确描述乳杆菌活菌数的实时原位动态,还可为研究其他发酵体系中不可培养的乳杆菌相关代谢功能提供参考。
| [1] |
Duar RM, Lin XB, Zheng JS, et al. Lifestyles in transition: evolution and natural history of the genus Lactobacillus[J]. FEMS Microbiology Reviews, 2017, 41(Supp_1): S27-S48. |
| [2] |
Hill C, Guarner F, Reid G, et al. The international scientific association for probiotics and prebiotics consensus statement on the scope and appropriate use of the term probiotic[J]. Nature Reviews Gastroenterology & Hepatology, 2014, 11(8): 506-514. |
| [3] |
Andreevskaya M, Jääskeläinen E, Johansson P, et al. Food spoilage-associated Leuconostoc, Lactococcus, and Lactobacillus species display different survival strategies in response to competition[J]. Applied and Environmental Microbiology, 2018, 84(13): e00554-18. |
| [4] |
Xia XL, Zhang QW, Zhang B, et al. Insights into the biogenic amine metabolic landscape during industrial semidry Chinese rice wine fermentation[J]. Journal of Agricultural and Food Chemistry, 2016, 64(39): 7385-7393. |
| [5] |
Huang Y. Metabolic control of biogenic amines and its application in semi-dry Chinese rice wine[D]. Wuxi: Master's Thesis of Jiangnan University, 2018 (in Chinese) 黄杨.半干型黄酒生物胺代谢调控研究及其应用[D].无锡: 江南大学硕士学位论文, 2018 |
| [6] |
Hong XT, Chen J, Liu L, et al. Metagenomic sequencing reveals the relationship between microbiota composition and quality of Chinese Rice Wine[J]. Scientific Reports, 2016, 6: 26621. |
| [7] |
Alqurashi RM, Alarifi SN, Walton GE, et al. In vitro approaches to assess the effects of acai (Euterpe oleracea) digestion on polyphenol availability and the subsequent impact on the faecal microbiota[J]. Food Chemistry, 2017, 234: 190-198. |
| [8] |
Qiu R, Lu J. Bacterial community dynamics during malting based on T-RFLP and 454 pyrosequencing profiles[J]. Journal of Food Science and Biotechnology, 2019, 38(4): 71-77. (in Chinese) 邱然, 陆健. T-RFLP和454焦磷酸测序方法分析制麦过程细菌群落的动态变化[J]. 食品与生物技术学报, 2019, 38(4): 71-77. |
| [9] |
Wu CD, Zheng J, Huang J, et al. Reduced nitrite and biogenic amine concentrations and improved flavor components of Chinese sauerkraut via co-culture of Lactobacillus plantarum and Zygosaccharomyces rouxii[J]. Annals of Microbiology, 2014, 64(2): 847-857. |
| [10] |
Lv XC, Li Y, Qiu WW, et al. Development of propidium monoazide combined with real-time quantitative PCR (PMA-qPCR) assays to quantify viable dominant microorganisms responsible for the traditional brewing of Hong Qu glutinous rice wine[J]. Food Control, 2016, 66: 69-78. |
| [11] |
Rizzotti L, Levav N, Fracchetti F, et al. Effect of UV-C treatment on the microbial population of white and red wines, as revealed by conventional plating and PMA-qPCR methods[J]. Food Control, 2015, 47: 407-412. |
| [12] |
Ling N, Shen JL, Guo JJ, et al. Rapid and accurate detection of viable Vibrio parahaemolyticus by sodium deoxycholate-propidium monoazide-qPCR in shrimp[J]. Food Control, 2020, 109: 106883. |
| [13] |
Emerson JB, Adams RI, Román CMB, et al. Schrödinger's microbes: Tools for distinguishing the living from the dead in microbial ecosystems[J]. Microbiome, 2017, 5(1): 86. |
| [14] |
Gensberger ET, Polt M, Konrad-Köszler M, et al. Evaluation of quantitative PCR combined with PMA treatment for molecular assessment of microbial water quality[J]. Water Research, 2014, 67: 367-376. |
| [15] |
Slimani S, Robyns A, Jarraud S, et al. Evaluation of propidium monoazide (PMA) treatment directly on membrane filter for the enumeration of viable but non cultivable Legionella by qPCR[J]. Journal of Microbiological Methods, 2012, 88(2): 319-321. |
| [16] |
Fittipaldi M, Nocker A, Codony F. Progress in understanding preferential detection of live cells using viability dyes in combination with DNA amplification[J]. Journal of Microbiological Methods, 2012, 91(2): 276-289. |
| [17] |
Gobert G, Cotillard A, Fourmestraux C, et al. Droplet digital PCR improves absolute quantification of viable lactic acid bacteria in faecal samples[J]. Journal of Microbiological Methods, 2018, 148: 64-73. |
| [18] |
Byun R, Nadkarni MA, Chhour KL, et al. Quantitative analysis of diverse Lactobacillus species present in advanced dental caries[J]. Journal of Clinical Microbiology, 2004, 42(7): 3128-3136. |
| [19] |
Costa GN, Vilas-Bôas GT, Vilas-Boas LA, et al. In silico phylogenetic analysis of lactic acid bacteria and new primer set for identification of Lactobacillus plantarum in food samples[J]. European Food Research and Technology, 2011, 233(2): 233-241. |
| [20] |
Schwendimann L, Kauf P, Fieseler L, et al. Development of a quantitative PCR assay for rapid detection of Lactobacillus plantarum and Lactobacillus fermentum in cocoa bean fermentation[J]. Journal of Microbiological Methods, 2015, 115: 94-99. |
| [21] |
Matsuda K, Tsuji H, Asahara T, et al. Establishment of an analytical system for the human fecal microbiota, based on reverse transcription-quantitative PCR targeting of multicopy rRNA molecules[J]. Applied and Environmental Microbiology, 2009, 75(7): 1961-1969. |
| [22] |
Tabasco R, Paarup T, Janer C, et al. Selective enumeration and identification of mixed cultures of Streptococcus thermophilus, Lactobacillus delbrueckii subsp. paracasei and Bifidobacterium lactis in fermented milk[J]. International Dairy Journal, 2007, 17(9): 1107-1114. |
| [23] |
Stiles ME, Holzapfel WH. Lactic acid bacteria of foods and their current taxonomy[J]. International Journal of Food Microbiology, 1997, 36(1): 1-29. |
| [24] |
Wu LW, Yang YF, Chen S, et al. Microbial functional trait of rRNA operon copy numbers increases with organic levels in anaerobic digesters[J]. The ISME Journal, 2017, 11(12): 2874-2878. |
| [25] |
Wang RR, Li XM, Chen L, et al. Detection of biogenic amines produced by lactic acid bacteria isolated from the fermentation process of Chinese rice wine[J]. Food and Fermentation Industries, 2017, 43(1): 12-17. (in Chinese) 王然然, 李晓敏, 陈柳, 等. 黄酒发酵过程中乳酸菌的分离及对其产生物胺能力的评价[J]. 食品与发酵工业, 2017, 43(1): 12-17. |
| [26] |
Yu W, Li XM, Lu J, et al. Citrulline production by lactic acid bacteria in Chinese rice wine[J]. Journal of the Institute of Brewing, 2018, 124(1): 85-90. |
| [27] |
Oliver JD. Recent findings on the viable but nonculturable state in pathogenic bacteria[J]. FEMS Microbiology Reviews, 2010, 34(4): 415-425. |
| [28] |
Lai CH, Wu SR, Pang JC, et al. Designing primers and evaluation of the efficiency of propidium monoazide - quantitative polymerase chain reaction for counting the viable cells of Lactobacillus gasseri and Lactobacillus salivarius[J]. Journal of Food and Drug Analysis, 2017, 25(3): 533-542. |
| [29] |
Chakravorty S, Helb D, Burday M, et al. A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria[J]. Journal of Microbiological Methods, 2007, 69(2): 330-339. |
| [30] |
Udomsil N, Chen S, Rodtong S, et al. Quantification of viable bacterial starter cultures of Virgibacillus sp. and Tetragenococcus halophilus in fish sauce fermentation by real-time quantitative PCR[J]. Food Microbiology, 2016, 57: 54-62. |
| [31] |
Wang D, Yamahara KM, Cao YP, et al. Absolute quantification of enterococcal 23S rRNA gene using digital PCR[J]. Environmental Science & Technology, 2016, 50(7): 3399-3408. |
 2020, Vol. 47
2020, Vol. 47




