细菌Lon蛋白酶研究进展
刘郁夫
,
董浩
,
孙石静
,
陈瑞爱
,
丁家波
微生物学通报  2019, Vol. 46 2019, Vol. 46 Issue (7): 1706−1711 Issue (7): 1706−1711
|

Lon蛋白是首个被鉴定的ATP依赖蛋白酶,早期科学家发现大肠杆菌在紫外照射下形成长线型、分裂受抑制的突变表型,因此把该突变基因取名为lon[1]。后续研究发现Lon蛋白具有蛋白酶活性,属于AAA+ (ATPases associated with a variety of cellular activities)蛋白家族成员[2]。该蛋白家族成员还包括Clp蛋白酶(ClpAP、ClpXP、ClpYQ)和FtsH蛋白酶等,它们可降解错误折叠蛋白,在调控细菌内蛋白质稳态过程中发挥重要作用[3]。
Lon蛋白广泛存在于自然界中,在细菌、真菌、古细菌等原核生物和真核生物基因组中均能比对找到Lon同源蛋白,提示ATP依赖蛋白酶的蛋白降解功能作为细菌内一类保守的蛋白调控机制,具有靶点多样性且发挥重要作用[4]。
最近研究表明Lon蛋白除了降解错误折叠蛋白外,还通过降解多种调节蛋白参与调节细菌毒力[5]、耐药性[6]、胞内压力应激反应[7]、DNA修复[8]等多个细菌生理过程。本文通过对Lon蛋白酶的结构、功能与上下游调控网络作一综述,旨在更全面清楚地了解Lon蛋白酶的生理功能及其精密的胞内调控网络,为基因工程弱毒疫苗的研发和潜在药物治疗靶点的筛选提供参考。
1 Lon蛋白结构及水解机制大肠杆菌(Escherichia coli) Lon蛋白由784个氨基酸组成,大小约为87 kD,在细胞质中形成六聚体环状复合物(图 1)[9]。每个Lon多肽链由3个结构域组成:N端结构域参与底物的识别;ATP酶结构域(AAA+)负责ATP结合和水解,为Lon蛋白酶发挥酶活性提供能量;酶活性中心由Ser679-Lys722二联体组成,具有蛋白水解活性[10]。
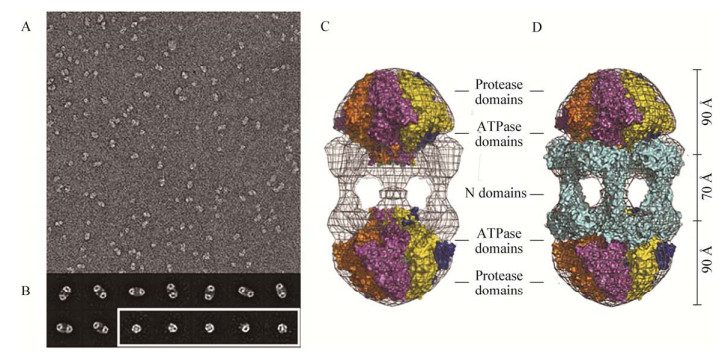
|
| 图 1 电镜观察Lon蛋白及Lon蛋白电子密度图[9] Figure 1 Electron microscopy and electron-density map of Lon protein[9] 注:A:电镜视野下观察Lon蛋白颗粒;B:电镜下Lon蛋白十二聚体(上排和左下)和六聚体(右下)的典型视野;C:B. subtilis Lon蛋白晶体结构的电子密度图;D:E. coli Lon蛋白晶体结构的电子密度图. Note: A: Representative field-of-view of electron micrographs of single wild-type Lon particles. (29 000× magnification); B: Representative class images of dodecamers (upper and lower left) and hexamers (lower right, enclosed in white box); C: The electron-density map of crystal structure of B. subtilis Lon; D: The electron-density map of crystal structure of E. coli Lon. |
|
|
根据Lon蛋白氨基酸序列的同源性和结构特征,可将Lon蛋白分为两类:存在于大部分细菌的LonA和主要存在于古细菌中的LonB;LonA含有大段的N端结构域,而LonB缺少N端结构域,但是在AAA+结构域中插入了一段转膜区(图 2)[2]。
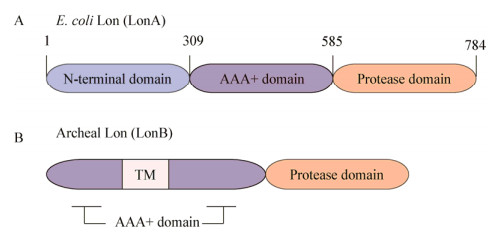
|
| 图 2 LonA和LonB的结构域[2] Figure 2 The structural domain of LonA and LonB[2] 注:A:细菌中Lon蛋白(主要为LonA)结构域示意图;B:古细菌中LonB蛋白结构域示意图,TM指代古细菌跨膜区. Note: A: Domain organization of Lon proteases found in bacteria (predominantly LonA); B: Domain organization of Lon proteases found in archaea (Lon B), TM refers to the trans-membrane anchor. |
|
|
Lon蛋白酶及其伴侣分子(主要是DanK系统)在降解细胞内错误折叠蛋白,以及维持胞内稳态过程中发挥重要作用[11]。由于Lon蛋白酶的底物切割位点位于圆柱状封闭空间的内部,与细胞质分隔开,所以也将Lon蛋白酶归类于“自独立” (Self-compartmentalized)蛋白酶[7]。Lon蛋白酶的水解机制与大多数蛋白酶类似:(1)在接头蛋白的协助下,Lon蛋白酶N端结构域识别并结合特异性蛋白底物,此过程不消耗ATP;(2) ATP结合水解位点使蛋白酶复合体的构象发生改变,底物多肽去折叠;(3)在移位酶的作用下,将多肽移至蛋白酶活性中心,蛋白酶切割多肽[10]。
2 Lon蛋白酶对细菌表型的影响研究人员起初发现E. coli Lon突变株的分裂受抑制,生长缓慢,菌体形成长线形,并且对紫外线的敏感性增强[1]。接着研究者们陆续发现Lon蛋白酶参与细胞内多个生理过程,起翻译后调控的作用[5, 12]。
Su等发现根瘤农杆菌(Agrobacterium tumefaciens) Lon突变株并没有表现出丝状细菌形态和紫外敏感性,但突变株生长缓慢,电镜观察发现超过80%的细菌形态发生改变,并且在37 ℃环境下可诱导lon基因转录和蛋白水平的表达上调[13]。幽门螺旋杆菌(Helicobacter pylori) lon基因突变后使得RdxA蛋白的甲硝唑还原酶活性下降44%,并最终导致H. pylori对甲硝唑的敏感性上升,亲本株对甲硝唑的最小抑菌浓度(Minimal inhibit concentration,MIC)是lon突变株的3倍[14]。铜绿假单胞菌(Pseudomonas aeruginosa) lon基因突变株可影响细菌的分裂过程,突变株导致细菌运动能力明显缺陷,对环丙沙星的敏感性增强8倍[15]。Xie等证明LonA是胸膜放线杆菌(Actinobacillus pleuropneumoniae)重要的压力耐受蛋白,该基因缺失在正常环境下(37 ℃)不影响细菌生长速度,但在低温(25 ℃)和高温(42 ℃)下导致缺失株均表现生长缺陷,环境应激实验表明lonA基因突变导致细菌对高渗、活性氧介质的敏感性增强[16]。
Pressler等系统研究了霍乱弧菌(Vibrio cholerae)胞内ATP蛋白酶的功能及作用[17];Δlon可分别通过上调VpsA基因和Ⅳ型鞭毛相关基因的转录,使细菌胞外多糖的产量上升,细菌运动性增强,同时下调FliA等毒力相关基因的表达,显著降低细菌在小鼠体内的定殖数量[17]。Fernández等发现P. aeruginosa lon基因缺失株和Hfq过表达菌株均表现出运动能力、生物被膜形成能力缺陷,提示Hfq蛋白在细菌胞内聚集是形成lon突变株表型的原因之一[18]。枯草芽孢杆菌(Bacillus subtilis) Lon蛋白通过接头分子SmiA介导,特异性地直接降解鞭毛合成的主要调控蛋白SwrA,从而控制细菌表面菌毛的密度,改变细菌的活动方式[19]。
在不同细菌中,Lon蛋白发挥着不完全一致的功能,lon基因突变可导致多种表型。总体来说,lon基因突变可导致细菌的生长速度、紫外敏感性、细菌形态、运动性、抗生素敏感性以及细菌生物被膜发生改变。
3 Lon蛋白对致病菌毒力的影响目前已报道lon基因在多种细菌,特别是革兰氏阴性菌的致病过程中发挥作用。Breidenstein等通过荧光定量PCR发现lon基因突变可下调P. aeruginosa三型分泌系统相关基因的表达,并利用人支气管上皮细胞模型、阿米巴原虫模型和CD-1小鼠模型证明P. aeruginosa毒力减弱,lon缺失株感染组小鼠的支气管载菌量为亲本株的1/500[15]。Xie等实验证明lonA基因缺失可通过改变细菌生物被膜的合成能力引起细菌毒力下降,亲本株在BALB/c小鼠肺脏、肝脏和肾脏中的载菌量为突变株的100−1 000倍,但是lonC基因不影响细菌毒力[16]。在沙门氏菌(Salmonella enterica)中lon基因突变可通过降低细菌胞外多糖的产量导致细菌毒力减弱,Lalsiamthara等以强毒株为亲本构建S. enterica Δlon基因缺失工程弱毒疫苗,免疫本体动物可诱导机体产生特异性IgG和外周血淋巴细胞介导的细胞免疫,同时提供动物机体对亲本毒株的抵抗力[20]。
细菌为适应宿主体内环境,可通过细菌分泌系统分泌毒力蛋白到宿主细胞质中,为自己复制繁殖提供最适条件。近期研究显示,Lon蛋白还可调控细菌分泌系统的表达,间接改变细菌的毒力[12]。Rogers等利用基因芯片技术发现与亲本毒株相比,V. cholerae LonA突变株中参与生物被膜形成的基因表达显著减少,而与Ⅵ型分泌系统相关的基因表达增加,引起Ⅵ型分泌系统活性增强,对小鼠致病性减弱,然而两者之间相互作用的机制尚不明确[21]。Zhou等的研究表明,Lon蛋白645位丝氨酸的磷酸化可导致Lon蛋白水解酶失效,可使HrpG更稳定,进而促进柑桔溃疡病菌(Xanthomonas citri) T3SS分泌系统的表达,对维持细菌的毒力至关重要[12]。Lee等发现,lon基因突变导致T3SS的表达上调和运动能力下降,后续研究发现Lon蛋白可间接作用Rcs和Gac-Csr调控系统,调节梨火疫病病原菌(Erwinia amylovora)的毒力[5]。
随着外界环境的改变,细菌在面对各种胁迫环境时会启动胞内的调控机制,改变胞内一系列的生理代谢途径以适应环境。而大量文献报道,lon基因突变后可导致致病菌毒力下降,提示Lon蛋白酶介导的翻译后调控作用对细菌在胁迫环境下的存活至关重要,是致病菌关键毒力基因之一,可作为基因工程致弱菌的潜在修饰靶点。
4 Lon蛋白的下游作用靶点研究E. coli MarA蛋白是Lon蛋白酶底物之一,在E. coli中lon基因突变导致MarA及其下游底物在细菌体内聚集,引起细菌对抗菌药物阿司匹林和多种抗生素的敏感性增强[22]。在大肠杆菌SOS应答过程中,E. coli细胞分裂抑制蛋白SulA的表达得到显著上调[23],lon基因突变后细菌丧失降解SulA的能力,导致细菌分裂周期调控紊乱,并使细菌的生长速度减缓[24]。
Lon蛋白在细胞内不仅能降解错误折叠蛋白,起到维持蛋白质动态平衡的作用,同时Lon蛋白还是一种重要压力应激蛋白,细菌在各种逆境的胁迫下,可降解各种不稳定的调节因子,通过调节细胞内一系列生理生化反应和代谢途径来适应不利环境[25]。
S. enterica铁离子摄入系统的FeoB和FeoC基因控制细菌Fe2+离子的摄入,Kim等研究表明Lon蛋白酶可水解S. enterica铁离子输入蛋白FeoC,调控细菌在胁迫环境下细菌体内的铁离子平衡,避免在遇到极端环境时金属离子的过度吸收[26]。Jonas等发现新月柄杆菌(Caulobacter crescentus)热应激可诱导Lon蛋白酶的合成,并在HSP70伴侣分子DnaK的介导下,直接降解细菌复制的启动蛋白DnaA,抑制细菌DNA的复制,从而形成细菌在热应激环境下抑制复制的保护机制[27]。Fernández等利用定量蛋白质组学方法探索Lon蛋白作用靶点,发现P. aeruginosa的lon基因突变株中,RNA结合蛋白Hfq的含量显著增加,并且在体外实验中证明了Lon蛋白对Hfq的降解作用,研究人员认为Lon蛋白可通过Hfq间接参与调控sRNA的表达[18]。
Lon蛋白酶的作用底物具有多样性,目前只鉴定出少数底物蛋白且进展缓慢。定量蛋白质组学作为一种新兴成熟的实验技术,在筛选Lon蛋白酶底物研究中具有天然的优势。Arends等利用Laber-free定量蛋白质组学方法在E. coli中新筛选出14个Lon蛋白作用底物,这些底物蛋白主要参与细菌氧化应激、核酸合成、氨基酸和能量代谢生理过程[28]。利用定量蛋白质组学技术筛选Lon蛋白酶的潜在底物可极大推进人们对Lon蛋白酶作用功能的认识。
5 Lon蛋白上游调控网络研究在先前研究报道中,lon基因属于热应激操纵子(Heat-shock regulon,HS),热应激可诱导lon基因的表达[29-30]。Robertson等通过Northern实验发现流产布鲁氏菌(Brucella abortus)在热休克以及其他压力环境下,lon基因的转录水平上调,但具体的调控方式仍需进一步研究[31]。然而也有例外,Myxococcus xanthus[32]中的lon同源基因并不是热诱导的,在不同的细菌中,lon基因的转录调控方式也不尽相同。
E. coli通过σ32因子激活HS的转录活性,在正常条件下DnaK分子伴侣系统和HflB蛋白酶使σ32因子处于非稳定状态,而在热应激环境下,细胞内积聚的非折叠蛋白竞争性与DnaK和蛋白酶结合,导致形成稳定状态的σ32因子,进而直接促进lon基因的表达[33]。B. subtilis在高渗、乙醇、H2O2和嘌呤霉素的环境胁迫下可诱导lonA基因的表达[29],但是这种转录激活作用并不依赖于压力应激σB因子,其内在的调控机制尚不明确。
变铅青链霉菌(Streptomyces lividans)除了通过HspR (Heat-shock protein regulator)负向调控lon基因表达外,还通过ClgR正向诱导Lon蛋白合成,发挥Lon蛋白酶活性降解错误折叠蛋白。同时ClgR还能被ClpP降解,形成负反馈调节机制,避免Lon蛋白过度表达[30],但是何种环境条件触发ClgR的表达仍不清楚。
正常环境下细菌胞内蛋白酶的量必须维持在恰当的水平,以维持细菌正常的生理过程,然而在特定情况下,细菌胞内Lon的蛋白水解作用有利于细菌在逆境中存活,然而人们对Lon蛋白表达调控的机制知之甚少[3]。
6 结论与展望细菌胞内Lon及其他ATP依赖蛋白酶介导的蛋白降解机制是一种保守机制,它可降解细菌胞内错误折叠蛋白,对于维持细菌胞内的蛋白质平衡具有重要意义。同时,Lon蛋白由于作用靶点具有多样性而在细菌胞内发挥多重作用。研究表明,lon基因缺失可影响细菌生长速度[26]、胁迫环境下的耐受能力[16]、抗生素敏感性[6]以及毒力[17]。
在先前研究报道中,B. abortus在热应激、酸性条件(pH 4.5)、EtOH、嘌呤霉素等应激环境下可引起lon基因的转录上调,lon基因缺失株在体外应激环境中表现出抵抗力下降,并影响B. abortus对小鼠模型的早期感染[31]。由于其中的分子机制尚不明确,因此本实验室采用原核细胞转录测序技术揭示了lon基因缺失与细菌对应激环境敏感性上升和毒力下降之间的分子机制[34]。研究发现lon基因的缺失可引起布鲁氏菌胞内大量与热应激、压力应激相关基因以及转录调控因子的转录发生变化,提示正常表达的Lon蛋白参与细菌胞内一系列重要生理过程,但是Lon蛋白调控这些生理过程的机制和方式有待后续进一步研究。
由于我们对Lon蛋白在B. abortus细菌内的作用靶点及其调控网络了解得不够清楚,接下来我们将利用蛋白质组学开展Lon蛋白下游靶点研究,并设计体外酶降解实验对Lon蛋白酶的底物进行鉴定。另外,在应用研究方面,我们将以lon基因为修饰靶点构建基因工程弱毒疫苗,利用动物模型评价基因工程弱毒疫苗的安全性和有效性,最终实现控制布鲁氏菌病流行传播的目的。
| [1] |
Swamy KHS, Goldberg AL. E. coli contains eight soluble proteolytic activities, one being ATP dependent[J]. Nature, 1981, 292(5824): 652-654. DOI:10.1038/292652a0 |
| [2] |
Gur E. The Lon AAA+ protease[A]//Dougan D. Regulated Proteolysis in Microorganisms[M]. Dordrecht: Springer, 2013, 66: 35-51
|
| [3] |
Tsilibaris V, Maenhaut-Michel G, van Melderen L. Biological roles of the Lon ATP-dependent protease[J]. Research in Microbiology, 2006, 157(8): 701-713. DOI:10.1016/j.resmic.2006.05.004 |
| [4] |
van Melderen L, Aertsen A. Regulation and quality control by Lon-dependent proteolysis[J]. Research in Microbiology, 2009, 160(9): 645-651. DOI:10.1016/j.resmic.2009.08.021 |
| [5] |
Lee JH, Ancona V, Zhao YF. Lon protease modulates virulence traits in Erwinia amylovora by direct monitoring of major regulators and indirectly through the Rcs and Gac-Csr regulatory systems[J]. Molecular Plant Pathology, 2018, 19(4): 827-840. DOI:10.1111/mpp.2018.19.issue-4 |
| [6] |
Nicoloff H, Andersson DI. Lon protease inactivation, or translocation of the lon gene, potentiate bacterial evolution to antibiotic resistance[J]. Molecular Microbiology, 2013, 90(6): 1233-1248. DOI:10.1111/mmi.2013.90.issue-6 |
| [7] |
Breidenstein EBM, Hancock REW. Armand-Frappier outstanding student award — role of ATP-dependent proteases in antibiotic resistance and virulence[J]. Canadian Journal of Microbiology, 2013, 59(1): 1-8. DOI:10.1139/cjm-2012-0681 |
| [8] |
Inoue M, Fukui K, Fujii Y, et al. The Lon protease-like domain in the bacterial RecA paralog RadA is required for DNA binding and repair[J]. The Journal of Biological Chemistry, 2017, 292(23): 9801-9814. DOI:10.1074/jbc.M116.770180 |
| [9] |
Vieux EF, Wohlever ML, Chen JZ, et al. Distinct quaternary structures of the AAA+ Lon protease control substrate degradation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(22): E2002-E2008. DOI:10.1073/pnas.1307066110 |
| [10] |
Lin CC, Su SC, Su MY, et al. Structural insights into the allosteric operation of the lon AAA+ protease[J]. Structure, 2016, 24(5): 667-675. DOI:10.1016/j.str.2016.03.001 |
| [11] |
Wohlever ML, Baker TA, Sauer RT. Roles of the N domain of the AAA+ Lon protease in substrate recognition, allosteric regulation and chaperone activity[J]. Molecular Microbiology, 2014, 91(1): 66-78. DOI:10.1111/mmi.2014.91.issue-1 |
| [12] |
Zhou XF, Teper D, Andrade MO, et al. A phosphorylation switch on Lon protease regulates bacterial type Ⅲ secretion system in host[J]. mBio, 2018, 9(1): e02146-17. DOI:10.1128/mBio.02146-17 |
| [13] |
Su SC, Stephens BB, Alexandre G, et al. Lon protease of the α-proteobacterium Agrobacterium tumefaciens is required for normal growth, cellular morphology and full virulence[J]. Microbiology, 2006, 152(4): 1197-1207. DOI:10.1099/mic.0.28657-0 |
| [14] |
Tu IF, Liao JH, Yang FL, et al. Lon protease affects the RdxA nitroreductase activity and metronidazole susceptibility in Helicobacter pylori[J]. Helicobacter, 2014, 19(5): 356-366. DOI:10.1111/hel.2014.19.issue-5 |
| [15] |
Breidenstein EBM, Janot L, Strehmel J, et al. The Lon protease is essential for full virulence in Pseudomonas aeruginosa[J]. PLoS One, 2012, 7(11): e49123. DOI:10.1371/journal.pone.0049123 |
| [16] |
Xie F, Li G, Zhang YH, et al. The Lon protease homologue LonA, not LonC, contributes to the stress tolerance and biofilm formation of Actinobacillus pleuropneumoniae[J]. Microbial Pathogenesis, 2016, 93: 38-43. DOI:10.1016/j.micpath.2016.01.009 |
| [17] |
Pressler K, Vorkapic D, Lichtenegger S, et al. AAA+ proteases and their role in distinct stages along the Vibrio cholerae lifecycle[J]. International Journal of Medical Microbiology, 2016, 306(6): 452-462. DOI:10.1016/j.ijmm.2016.05.013 |
| [18] |
Fernández L, Breidenstein EB, Taylor PK, et al. Interconnection of post-transcriptional regulation: The RNA-binding protein Hfq is a novel target of the Lon protease in Pseudomonas aeruginosa[J]. Scientific Reports, 2016, 6: 26811. DOI:10.1038/srep26811 |
| [19] |
Mukherjee S, Bree AC, Liu J, et al. Adaptor-mediated Lon proteolysis restricts Bacillus subtilis hyperflagellation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(1): 250-255. DOI:10.1073/pnas.1417419112 |
| [20] |
Lalsiamthara J, Lee JH. Virulence associated genes-deleted Salmonella montevideo is attenuated, highly immunogenic and confers protection against virulent challenge in chickens[J]. Frontiers in Microbiology, 2016, 7: 1634. |
| [21] |
Rogers A, Townsley L, Gallego-Hernandez AL, et al. The LonA protease regulates biofilm formation, motility, virulence, and the type Ⅵ secretion system in Vibrio cholerae[J]. Journal of Bacteriology, 2016, 198(6): 973-985. DOI:10.1128/JB.00741-15 |
| [22] |
Bhaskarla C, Das M, Verma T, et al. Roles of Lon protease and its substrate MarA during sodium salicylate-mediated growth reduction and antibiotic resistance in Escherichia coli[J]. Microbiology, 2016, 162(5): 764-776. DOI:10.1099/mic.0.000271 |
| [23] |
Nishii W, Maruyama T, Matsuoka R, et al. The unique sites in SulA protein preferentially cleaved by ATP-dependent Lon protease from Escherichia coli[J]. European Journal of Biochemistry, 2002, 269(2): 451-457. DOI:10.1046/j.0014-2956.2001.02668.x |
| [24] |
Nazir A, Harinarayanan R. Inactivation of cell division protein FtsZ by SulA makes Lon indispensable for the viability of a ppGpp0 strain of Escherichia coli[J]. Journal of Bacteriology, 2015, 198(4): 688-700. |
| [25] |
Heuveling J, Possling A, Hengge R. A role for Lon protease in the control of the acid resistance genes of Escherichia coli[J]. Molecular Microbiology, 2008, 69(2): 534-547. DOI:10.1111/mmi.2008.69.issue-2 |
| [26] |
Kim H, Lee H, Shin D. Lon-mediated proteolysis of the FeoC protein prevents Salmonella enterica from accumulating the Fe(Ⅱ) transporter FeoB under high-oxygen conditions[J]. Journal of Bacteriology, 2015, 197(1): 92-98. DOI:10.1128/JB.01826-14 |
| [27] |
Jonas K, Liu J, Chien P, et al. Proteotoxic stress induces a cell-cycle arrest by stimulating Lon to degrade the replication initiator DnaA[J]. Cell, 2013, 154(3): 623-636. DOI:10.1016/j.cell.2013.06.034 |
| [28] |
Arends J, Griego M, Thomanek N, et al. An integrated proteomic approach uncovers novel substrates and functions of the Lon protease in Escherichia coli[J]. Proteomics, 2018, 18(13): 1800080. DOI:10.1002/pmic.v18.13 |
| [29] |
Riethdorf S, Völker U, Gerth U, et al. Cloning, nucleotide sequence, and expression of the Bacillus subtilis lon gene[J]. Journal of Bacteriology, 1994, 176(21): 6518-6527. DOI:10.1128/jb.176.21.6518-6527.1994 |
| [30] |
Sobczyk A, Bellier A, Viala J, et al. The lon gene, encoding an ATP-dependent protease, is a novel member of the HAIR/HspR stress-response regulon in actinomycetes[J]. Microbiology, 2002, 148(6): 1931-1937. DOI:10.1099/00221287-148-6-1931 |
| [31] |
Robertson GT, Kovach ME, Allen CA, et al. The Brucella abortus Lon functions as a generalized stress response protease and is required for wild-type virulence in BALB/c mice[J]. Molecular Microbiology, 2000, 35(3): 577-588. |
| [32] |
Tojo N, Inouye S, Komano T. Cloning and nucleotide sequence of the Myxococcus xanthus lon gene: indispensability of lon for vegetative growth[J]. Journal of Bacteriology, 1993, 175(8): 2271-2277. DOI:10.1128/jb.175.8.2271-2277.1993 |
| [33] |
Schumann W. Regulation of bacterial heat shock stimulons[J]. Cell Stress and Chaperones, 2016, 21(6): 959-968. DOI:10.1007/s12192-016-0727-z |
| [34] |
Liu YF, Dong H, Peng XW, et al. RNA-seq reveals the critical role of Lon protease in stress response and Brucella virulence[J]. Microbial Pathogenesis, 2019, 130: 112-119. DOI:10.1016/j.micpath.2019.01.010 |