扩展功能
文章信息
- 毛雅慧, 李峰
- MAO Ya-Hui, LI Feng
- 细菌3-酮脂酰ACP还原酶研究进展
- Research advances on bacterial 3-oxoacyl-acyl carrier protein reductase
- 微生物学通报, 2019, 46(4): 931-939
- Microbiology China, 2019, 46(4): 931-939
- DOI: 10.13344/j.microbiol.china.180319
-
文章历史
- 收稿日期: 2018-04-22
- 接受日期: 2018-06-11
- 网络首发日期: 2018-06-22
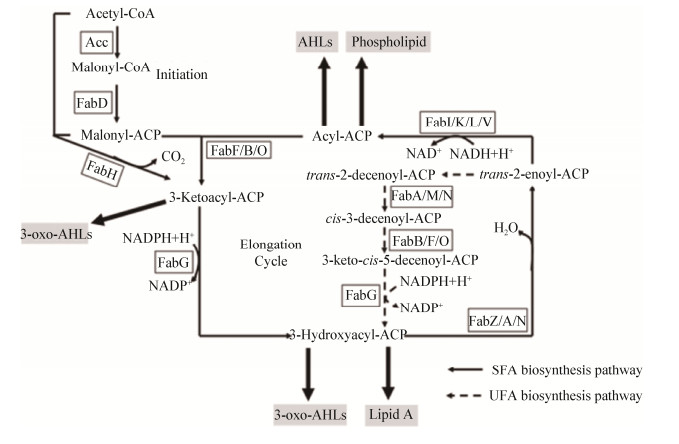
生物体能以乙酰辅酶A (CoA)作为前体物质,通过聚合、还原、脱水和再还原四步循环反应合成脂肪酸[1]。细菌采用II型脂肪酸合成系统从头合成脂肪酸,其特点是每步生化反应都由独立的酶催化完成,整个合成过程分起始和循环两个阶段,最终形成含16或18个碳原子的长链脂酰ACP[2](图 1)。以大肠杆菌(Escherichi coli)作为模式菌株,对Ⅱ型脂肪酸合成系统已进行了深入的研究。虽然细菌脂肪酸合成机制相对保守,但不同的细菌间也存在差异,表现出多样性[2-3]。目前,在脂肪酸合成酶系中已发现具有多样性特点的有:3-酮脂酰ACP合成酶[包括3-酮脂酰ACP合成酶III (FabH)[4-6]、3-酮脂酰ACP合成酶I (FabB)[7-8]和3-酮脂酰ACP合成酶II (FabF)[8-11]],四大类烯脂酰ACP还原酶FabI[12-16]、FabK[17-19]、FabL[20]和FabV[21-22],不同类型其酶学特征存在差异。然而3-酮脂酰ACP还原酶(FabG)的多样性报道较少,本文将针对3-酮脂酰ACP还原酶的功能、结构及其抑制剂等方面的研究进展进行综述,旨在为细菌脂肪酸合成途径及3-酮脂酰ACP还原酶的深入研究提供理论材料。
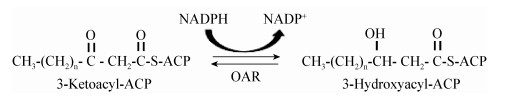
1 3-酮脂酰ACP还原酶简介3-酮脂酰ACP还原酶(OAR)在细菌中广泛存在并且十分保守,目前研究较为深入的是大肠杆菌的FabG,已知大肠杆菌有一个3-酮脂酰ACP还原酶(FabG),由fabG基因编码[23]。大肠杆菌FabG催化Ⅱ型脂肪酸合成循环反应中第一步还原反应,将3-酮脂酰ACP还原为3-羟脂酰ACP,以NADPH作为辅酶[24](图 2)。已经发现的所有FabG及其同系物都具有很高的相似性,并且具有类似的催化活性中心序列,它们都属于短链醇脱氢酶/还原酶(SDRs)超家族成员[25]。对细菌脂肪酸合成系统的研究至今,发现FabG是唯一能催化3-酮脂酰ACP这一中间底物还原的酶,也是限速酶,并早有研究报道fabG是大肠杆菌体内唯一编码3-酮脂酰ACP还原酶的基因,已有间接的转录分析数据证明了FabG对大肠杆菌生长的重要性。另外,Lai和Cronan于2003年分离鉴定了大肠杆菌和沙门氏菌(Salmonella enterica)的温度敏感fabG突变菌株也证明fabG基因对于这两种细菌的生长是必需的[26]。
|
| 图 2 3-酮脂酰ACP还原酶催化的反应 Figure 2 Reaction catalyzed by 3-Oxoacyl-ACP reductase |
|
|
除了大肠杆菌,在其他一些细菌中发现了3-酮脂酰ACP还原酶的同源蛋白,且不同细菌间3-酮脂酰ACP还原酶的功能具有差异性。由于FabG蛋白是SDR超家族的成员,这一家族为构成最大的蛋白质超家族之一,拥有大量细菌成员,使得对FabG蛋白的注释非常困难。目前有关3-酮脂酰ACP还原酶的研究多数集中于其在脂肪酸合成途径中的作用,参与细菌脂肪酸合成是该酶的典型特征,也是研究其生物学特性首先需要考虑的方面。然而3-酮脂酰ACP还原酶的功能又不仅仅局限于此,其功能还表现在其他诸多方面。
2.1 3-酮脂酰ACP还原酶FabG在脂肪酸合成中的功能对于常规的典型的FabG蛋白,主要是在细菌脂肪酸合成中发挥作用,因此,如果编码3-酮脂酰-ACP还原酶候选基因位于脂肪酸合成基因簇内,则对其功能的注释将更加准确。在乳酸乳球菌(Lactococcus lactis)中有2个注释为编码3-酮基脂酰ACP还原酶的基因fabG1和fabG2,它们在染色体上位置相隔较远,但能够与其邻近的其他脂肪酸合成酶基因一同转录。实验证明LlfabG1基因能编码有活性的3-酮基脂酰ACP还原酶,而LlfabG2基因编码的蛋白则不表现出3-酮基脂酰ACP还原酶的活性,但在体外实验中可以还原底物乙酰乙酰辅酶A (Acetoacetyl-CoA),所以乳酸乳球菌合成脂肪酸过程中行使3-酮基脂酰ACP还原酶活性的是LlFabG1,而目前LlFabG2具体功能尚不清楚[27]。在大多数情况下,细菌中只有一种OAR同系物被鉴定为在脂肪酸合成中起作用。不过近来有文献首次报道了同一细菌体内编码2个具有功能活性的3-酮脂酰ACP还原酶蛋白,在茄科雷尔氏菌(Ralstonia solanacearum)基因组中RSc1052 (fabG1)和RSp0359 (fabG2)被注释为fabG同源基因,研究发现FabG1和FabG2都具有3-酮脂酰ACP还原酶活性。不同的是,位于茄科雷尔氏菌染色体上的fabG1基因与大肠杆菌fabG一样,对细菌生长至关重要;而位于大质粒上的fabG2基因虽然可完全敲除,但敲除突变株具有与野生型不同的脂肪酸组成,说明FabG2也参与脂肪酸的合成;另外,FabG2还与逆境耐受性及植物致病性相关[28]。
2.2 3-酮脂酰ACP还原酶FabG在聚羟基脂肪酸合成途径中的作用光合蓝细菌Synechocystis sp. PCC6803的SpFabG的蛋白结构与乙酰乙酰CoA还原酶SpPhaB相似,PhaB是合成聚-3-羟基丁酸酯(PHB)所必需的。有研究证明SpFabG对蓝细菌脂肪酸合成是必需的,但非限速酶,通过体内实验分析表明SpFabG能够在缺氮的条件下部分补充SpPhaB,并且fabG的过表达将导致部分碳通量从脂肪酸合成途径向PHB的合成转移[29]。2000年Ren等发现铜绿假单胞菌的一个PafabG基因产物还表现出了3-酮脂酰CoA还原酶活性,并且能够使大肠杆菌FadA突变株像FadB突变一样积累中链长的多聚3-羟基饱和脂肪酸[30]。
2.3 FabG以脂肪酸为前体参与其他生物活性物质的合成在结核分枝杆菌(Mycobacterium tuberculosis)中发现有5个EcFabG的同源物MtFabG1– MtFabG5,其中MtFabG1/Rv1483具有较强的3-酮脂酰ACP还原酶活性,对于分枝杆菌的生长非常重要[31]。MtfabG1的功能可以由耻垢分枝杆菌(Mycobacterium smegmatis)的MsfabG互补,但不能被EcfabG互补,这可能是由于两者的蛋白结构不同所导致的。已知MtFabG1参与分枝菌酸的合成,而分枝菌酸是分枝杆菌细胞壁的主要成分,因此对MtFabG1理化性质的研究有助于开发新的抗结核病药物。Gurvitz于2009年报道了FabG1和FabG4能在酵母的FASII系统中行使功能[32]。尽管MtFabG4/Rv0242c像MtFabG1一样也是必需的,具有3-酮脂酰-硫酯还原酶活性,但其在分枝杆菌FASII中的功能仍有待探索。目前对MtFabG2–MtFabG5的生理功能还不清楚。天蓝色链霉菌(Streptomyces coelicolor)所产脂肪酸用于初级代谢和次级代谢物十二烷基二苷酸的生物合成,细菌基因组上有3个fabG (SCO1815、SCO1345、SCO1346),动力学分析显示,3种FabG都能够使用直链和支链β-酮脂酰-NAC,仅SCO1345能区分来自两种生物合成途径(脂肪酸合成和十一烷基纤维素生物合成)的ACP[33],其中SCO1815是脂肪酸合成的关键酶,但SCO1345和SCO1346的生物学功能还不清楚。
2.4 3-酮脂酰ACP还原酶FabG参与鞭毛的组装与调节经研究发现OAR还有参与细菌鞭毛的组装与调节的功能。巴西固氮螺菌(Azospirillum brasilense)由于具有组成型极性鞭毛(Fla)和诱导型侧鞭毛(Laf)的活性,使其可以进行游泳(Swim)和群泳(Swarm)。3-酮脂酰ACP还原酶同源基因fabG1 (AZOBR-p1160043)对这两种类型鞭毛的构成都是必需的[34-35]。fabG1对鞭毛在细胞膜上的正确定位和/或鞭毛的修饰起作用,这也是构建侧鞭毛Laf可诱导性所必需的,并且对于巴西固氮螺菌Sp245的适当游泳和群集运动也是必需的。
2.5 FabG同系物的功能在一些细菌中,除了具有FabG外,还存在其他一些具有3-酮脂酰ACP还原酶活性的酶,说明同一细菌中多拷贝3-酮脂酰ACP还原酶基因的存在并非冗余,它们具有不同的生物学功能,对细菌生理生化活动都具有十分重要的意义。例如,在铜绿假单胞菌(Pseudomonas aeruginosa)中至少有4个酮脂酰ACP还原酶同源基因,其中rhlG基因编码NADPH依赖的3-酮脂酰ACP还原酶RhlG不参与脂肪酸合成,但直接参与鼠李糖脂中脂链结构的合成,同时在聚羟基脂肪酸酯(PHA)的合成中发挥作用[36]。
毛雅慧等报道了在苜蓿中华根瘤菌(Sinorhizobium meliloti) SymA大质粒上位于nod基因簇中的nodG基因与染色体上传统的fabG在脂肪酸合成过程中所起的作用基本是一致的。但两者分别所编码的蛋白NodG和FabG在功能上又存在差异:首先,fabG是苜蓿中华根瘤菌生长所必需基因,而nodG突变不影响生长;其次,在体内过量表达NodG能够增强该菌抗逆性;再次,nodG突变会轻微影响其结瘤能力,验证了nodG不是苜蓿结瘤必需的[37],与先前的文献报道结论一致,但过量表达NodG在一定程度上增加结瘤效率[38]。尽管有文献报道过苜蓿中华根瘤菌中参与该细菌脂肪酸合成的另一限速酶烯脂酰ACP还原酶的同系物(SmFabI1和SmFabI2),并对fabI1的生理功能做了诠释,同样证明了fabI1的缺失对紫花苜蓿结瘤的影响[39]。然而却罕有文献报道研究其他共生固氮微生物中3-酮脂酰ACP还原酶的功能。
综合以上对3-酮脂酰ACP还原酶功能多样性的概述,可归纳总结为表 1。
| 功能 Function |
示例细菌 Bacterium |
FabG同系物 FabG Homologues |
参考文献 Reference |
| 参与脂肪酸合成 Involved in fatty acid synthesis |
Escherichi coli Lactococcus lactis Ralstonia solanacearum Sinorhizobium meliloti | EcFabG LlFabG1 RsFabG1, RsFabG2 SmFabG | [4] [8] [9] [18] |
| 参与聚羟基脂肪酸合成 Involved in polyhydroxyalkanoate synthesis |
Synechocystis sp. PCC6803 Pseudomonas aeruginosa | SpFabG PaFabG | [10] [11] |
| 参与其他生物活性物质的合成 Involved in biologically active substances synthesis |
Mycobacterium tuberculosis Streptomyces coelicolor Pseudomonas aeruginosa | MtFabG1(Rv1483) ScFabG(SCO1815) PaRhlG | [12-13] [14] [17] |
| 参与鞭毛的组装与调节 Involved in assembly and work of flagellar |
Azospirillum brasilense | AbFabG1 | [15-16] |
| 参与植物结瘤作用 Involved in alfalfa nodulation |
Sinorhizobium meliloti | SmNodG | [18] |
| 与环境耐受性相关 Related to environmental tolerance |
Ralstonia solanacearum Sinorhizobium meliloti | RsFabG2 SmNodG | [9] [18] |
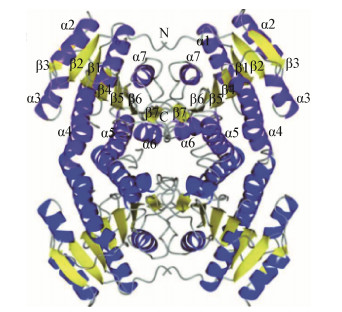
3-酮脂酰ACP还原酶多样性还表现在酶的结构及对底物专一性差异上。来自不同生物体的FabG晶体结构,不论是活性状态还是非活性状态已有相应报道。蛋白质数据库(Protein data bank,PDB)中报道的几种apo-FabG,包括大肠杆菌EcFabG[23]、结核杆菌MabA、立克次氏体RpFabG以及天蓝色链霉菌ScFabG,其蛋白构象为保守的SYK催化三联体(催化残基丝氨酸、酪氨酸和赖氨酸)的侧链彼此远离,NADP(H)结合口袋的入口被部分无序的β4-α4和β5-α5环遮蔽,这种构象被称为“无活性”形式(图 3)[40]。相反,在PDB中报道的EcFabG/NADP(H)和MabA/NADP+的复合构象则被称为“活性”形式的有序催化三联体构象。
E. coli FabG单体的分子质量为25.5 kD,在溶液中以四聚体的形式存在,其结构在2001年被确定。E. coli FabG晶体结构的研究结果表明,FabG结合其辅酶NADP(H)后,使3个与其催化活性有关的氨基酸(Ser138、Tyrl51、Lys155)进入活性口袋,引起FabG构象的变化,从而行使催化功能[41]。FabG与NADPH的结合为负合作,并且这种效应因酰基载体蛋白(ACP)的存在而增强。前文所述的结核分枝杆菌MtFabG1的分子结构模型表明,在其催化三分子附近有一个大的保守的底物结合口袋,适宜结合由FASII合成的长酰基链,与其它同源蛋白不同的是,MtFabG1优先作用于C8–C20的长链底物,而对C4底物亲和性较差。结核分枝杆菌MtFabG4的结构也有研究报道。通过模拟延伸的脂肪酰基链与CoA的共价连接,首次揭示了FabG4如何与其辅酶NADH和己酰基-CoA相互作用[42]。结构分析表明,FabG4的活性位点与己酰-CoA的结合显著区别于与分枝杆菌烯脂酰ACP还原酶FabI/InhA与C16脂酰底物的结合。ACP抑制实验表明FabG4能以脂酰-CoA或脂酰-ACP为催化底物。研究还表明,在FabG4二聚体中,一个单体的Arg146和Arg445与第二单体的C端相互作用,在底物缔合和催化中发挥关键作用[43]。对铜绿假单胞菌中3-酮脂酰ACP还原酶的晶体学分析表明,尽管RhlG.NADP+和FabG.NADP+复合物的整体结构确实相似,但在功能相关性结构上存在很大差异;此外,NADP(H)结合活性位点和底物结合亚结构域的结构不同[44]。RhlG虽然能催化β-酮癸酰-ACP向β-d-羟癸酰-ACP的还原,但比FabG催化相同反应时的活性低2 000倍[36]。
与EcFabG不同的是,霍乱弧菌VcFabG在溶液中主要以二聚体形式存在,并且能够自缔合成四聚体,这是晶体结构中的可见状态,辅因子NADP(H)可以促进四聚体的形成。VcFabG二聚体和四聚体状态之间的转变与二聚体-二聚体界面上5个螺旋构象的变化有关[45]。二聚体相邻界面的两个甘氨酸残基(G92和G141)为构象变化的铰链,而具有催化活性的酪氨酸(Y155)和一个谷氨酰胺残基与环β4-α4、环β5-α5间形成的氢键(Q152)起到了稳定活性构象的作用。
4 3-酮脂酰ACP还原酶的抑制剂近几年来,病原微生物导致的传染病的暴发和许多病原菌耐药性的增强,使得新型抗菌药物的研究开发工作变得越来越迫切,病原微生物全基因组测序的完成使许多抗菌药物筛选的靶点得以确定和深入研究。细菌脂肪酸合成酶系与真核生物不同,这为抗菌药物的设计和筛选提供了重要的信息[46-47]。以脂肪酸合成途径中关键酶作为潜在的药物作用靶点,筛选出克服耐药性、选择性高、毒性低的抑制剂,已成为抗菌药物研发的热点领域[48]。3-酮脂酰ACP还原酶(FabG)催化脂肪酸合成的关键还原反应对许多细菌的存活至关重要,且细菌中FabG高度保守,FabG抑制剂具有作为广谱抗生素的潜力,在分子水平上研究FabG的反应机制将有助于药物的合理设计[49]。目前已发现植物多酚类物质,包括黄酮类化合物对FabG具有抑制效果,例如绿茶中主要成分表焙儿茶素(EGCG)是一种广为人知的抑菌剂,EGCG和相关植物多酚的抗菌作用早已得到验证,且是以II型脂肪酸合成系统作为抑制靶点,EGCG及儿茶素有效地抑制了脂肪酸延伸过程中FabG和FabI所催化的还原反应[50]。有研究利用来源于5种不同品种枫叶的提取物及单宁酸进行抑菌实验,研究发现这些提取物对革兰氏阳性菌的抑制效果优于革兰氏阴性菌,其抑菌机制不同于标准抗生素左氧氟沙星(LFX),这些提取物能作用于3-酮脂酰ACP还原酶,且单宁酸对FabG的抑制效果最强[51]。2008年有研究提出,高良姜根茎提取物的抗菌作用可能与其能抑制3-酮脂酰ACP还原酶活性有关,并提出这种抑制机制是以对抗NADPH的竞争性模式抑制FabG[52];截至目前,少见研究报道抑制剂对FabG的不可逆抑制,而高良姜乙醇提取物可以不可逆地抑制FabG。反式肉桂酸衍生物作为大肠杆菌FabG的新型抑菌剂被开发,它可与辅因子NADPH竞争FabG活性位点的底物结合区,从而抑制酶的催化活性[53]。由此可见,对酶蛋白结构特点的研究对于开发针对细菌FabG的特异性抗生素是必不可少的。
5 展望细菌基因组中被注释为3-酮脂酰ACP还原酶的同源基因经常有多个拷贝,这一现象是生物进化的必然结果,同时也是细菌脂肪酸II型合成系统多样性特点的补充,同一细菌中不同同源蛋白的生物学功能存在差异,它们分工合作,共同维持细菌的生命活动。
聚酮体合成途径与脂肪酸合成途径相似性很高,尽管两者的催化酶系不同,但有着密切的联系[29]。细菌脂肪酸代谢过程中产生大量的R-3-羟基脂肪酸中间产物,以胞外分泌物组分(如鼠李糖脂)和胞内碳源(能量)的贮藏物质(如聚羟基脂肪酸酯,简称PHA)等形式存在。通过基因重组技术利用大肠埃希菌生产PHA等已有报道,在早期的研究中显示,PHA合酶基因和3-酮脂酰ACP还原酶基因(fabG)在重组大肠杆菌中的共表达可增强来自相关碳源,如脂肪酸的PHA的产生[54]。而后一种来自假单胞菌61-3菌株的新fabG基因被克隆,其基因表达产物也得到了鉴定,这一研究提供了第一个证据,即来自大肠杆菌(或具有fabH (F87T)的假单胞菌)的fabG基因和PHA合酶基因的共表达可以增强由非相关碳源产生的短链-中链长PHA共聚物[55]。这项研究确定了从脂肪酸生物合成产生短链-中链长PHA共聚物的途径,其可能影响重组生物体中PHA的产生。
群体感应是细菌间交流的一种重要方式。研究表明,革兰氏阴性细菌的脂肪酸合成代谢为群体感应信号分子的合成提供前体[56],但群体感应信号分子的合成机制,以及与脂肪酸合成代谢之间的调控等问题,还有待进一步探索。野油菜黄单胞菌(Xanthomnas campestris pv. campestris) Xcc8004基因组编码4个大肠杆菌FabG的同源蛋白,其中FabG1位于可能的脂肪酸合成酶基因簇中,推测FabG1是Xcc脂肪酸合成的关键酶,具有3-酮脂酰ACP还原酶活性。当细胞内3-酮脂酰ACP还原酶(FabG)活性增加时,其催化产物3-羟脂酰ACP的浓度也相应升高,其中部分3-羟脂酰ACP可作为RpfF的底物,用于DSF信号分子的合成,因此在Xcc中过量表达fabG1可显著升高DSF浓度[57]王海洪教授课题组在对野油菜黄单胞菌脂肪酸合酶的研究中发现FabG2和该菌群体感应信号分子DSF (Diffusible signal factor)合成直接相关,DSF是2004年首先在野油菜黄单胞菌中鉴定的一种新型长链不饱和脂肪酸群体感应信号[58],结构为顺式-11-甲基-2-十二碳烯酸,DSF信号分子的直接前体物质为顺式-11-甲基-2-十二碳羟脂酰ACP,即3-酮脂酰ACP还原酶的直接代谢产物,研究发现FabG2表达水平的高低会影响DSF信号分子的产量,进而影响该菌的致病性(数据待发表)。由此可见,FabG及其同系物除了参与脂肪酸的合成外,还参与其他生物活性物质的合成与代谢,这些生物活性物质包括细菌所产生的色素、生物素、与运动性相关的鼠李糖脂、对细胞生存和表面性质具有重要意义的类脂A以及群体感应中必需的酰基高丝氨酸内酯等,它们以脂肪酸合成途径所产生的中间产物为前体进行合成,3-酮脂酰ACP还原酶作为脂肪酸合成途径中的限速及关键酶,与这些生物活性物质的合成紧密相关。
细菌中同时具有多个OAR同源蛋白的现象普遍存在,当前国内外对3-酮脂酰ACP还原酶的研究主要针对其蛋白结构特征分析、酶学特点、抑制剂的开发等方面,而3-酮脂酰ACP还原酶其他方面的功能还有待探索和深入研究;由于缺乏理论材料和研究依据,3-酮脂酰ACP还原酶功能的开发仍然存在一定局限性。丰富细菌3-酮脂酰ACP还原酶多样性,有利于发现细菌未知的代谢途径。
另外,是否可根据FabG蛋白的结构特点和酶学特征将FabG分为不同的类型,作为细菌类别划分的依据也是值得思考的问题之一。FabG蛋白的多样性和对酶催化机制的结构特征的研究,对于合理设计特定针对FabG的抑制剂是十分重要的;以FabG作为靶点,进行抗菌药物的筛选和研发仍然是今后研究的热门方向。
| [1] |
Rawlings BJ. Biosynthesis of fatty acids and related metabolites[J]. Natural Product Reports, 1998, 15(3): 275-308. DOI:10.1039/a815275y |
| [2] |
Marrakchi H, Zhang YM, Rock CO. Mechanistic diversity and regulation of Type II fatty acid synthesis[J]. Biochemical Society Transactions, 2002, 30(6): 1050-1055. DOI:10.1042/bst0301050 |
| [3] |
Yu YH, Ma JR, Wang HH. Advances in fatty acid biosynthetic diversity in bacteria[J]. Journal of Microbiology, 2016, 36(4): 76-83. (in Chinese) 余永红, 马建荣, 王海洪. 细菌脂肪酸合成多样性的研究进展[J]. 微生物学杂志, 2016, 36(4): 76-83. DOI:10.3969/j.issn.1005-7021.2016.04.014 |
| [4] |
Campbell JW, Cronan JE Jr. Escherichia coli FadR positively regulates transcription of the fabB fatty acid biosynthetic gene[J]. Journal of Bacteriology, 2001, 183(20): 5982-5990. DOI:10.1128/JB.183.20.5982-5990.2001 |
| [5] |
White SW, Zheng J, Zhang YM, et al. The structural biology of type II fatty acid biosynthesis[J]. Annual Review of Biochemistry, 2005, 74: 791-831. DOI:10.1146/annurev.biochem.74.082803.133524 |
| [6] |
Choi KH, Heath RJ, Rock CO. β-ketoacyl-acyl carrier protein synthase III (FabH) is a determining factor in branched-chain fatty acid biosynthesis[J]. Journal of Bacteriology, 2000, 182(2): 365-370. DOI:10.1128/JB.182.2.365-370.2000 |
| [7] |
de Mendoza D, Klages Ulrich A, Cronan JE Jr. Thermal regulation of membrane fluidity in Escherichia coli. Effects of overproduction of beta-ketoacyl-acyl carrier protein synthase[J]. The Journal of Biological Chemistry, 1983, 258(4): 2098-2101. |
| [8] |
Garwin JL, Klages AL, Cronan JE Jr. Beta-ketoacyl-acyl carrier protein synthase II of Escherichia coli. Evidence for function in the thermal regulation of fatty acid synthesis[J]. The Journal of Biological Chemistry, 1980, 255(8): 3263-3265. |
| [9] |
Cheng JL, Ma JC, Lin JS, et al. Only one of the five Ralstonia solanacearum long-chain 3-ketoacyl-acyl carrier protein synthase homologues functions in fatty acid synthesis[J]. Applied and Environmental Microbiology, 2012, 78(5): 1563-1573. DOI:10.1128/AEM.07335-11 |
| [10] |
Zhu L, Cheng JL, Luo B, et al. Functions of the Clostridium acetobutylicium FabF and FabZ proteins in unsaturated fatty acid biosynthesis[J]. BMC Microbiology, 2009, 9: 119. DOI:10.1186/1471-2180-9-119 |
| [11] |
Ma JC, Deng LT, Tong WH, et al. Identification and function reasearch of five 3-ketoacyl-ACP synthase homologues[J]. Progress in Biochemistry and Biophysics, 2014, 41(9): 887-895. |
| [12] |
Massengo-Tiassé RP, Cronan JE. Diversity in enoyl-acyl carrier protein reductases[J]. Cellular and Molecular Life Sciences, 2009, 66(9): 1507-1517. DOI:10.1007/s00018-009-8704-7 |
| [13] |
Heath RJ, Rock CO. Enoyl-acyl carrier protein reductase (fabI) plays a determinant role in completing cycles of fatty acid elongation in Escherichia coli[J]. The Journal of Biological Chemistry, 1995, 270(44): 26538-26542. DOI:10.1074/jbc.270.44.26538 |
| [14] |
Heath RJ, White SW, Rock CO. Inhibitors of fatty acid synthesis as antimicrobial chemotherapeutics[J]. Applied Microbiology and Biotechnology, 2002, 58(6): 695-703. DOI:10.1007/s00253-001-0918-z |
| [15] |
Lei M, Ma JC, Wang HH. Identification of two enoyl-ACP reductase FabI1 and FabI2 in Brucella abortus[J]. Progress in Biochemistry and Biophysics, 2012, 39(5): 464-471. DOI:10.3724/SP.J.1206.2011.00463 |
| [16] |
Cummings JE, Kingry LC, Rholl DA, et al. The Burkholderia pseudomallei enoyl-acyl carrier protein reductase FabI1 is essential for in vivo growth and is the target of a novel chemotherapeutic with efficacy[J]. Antimicrobial Agents and Chemotherapy, 2014, 58(2): 931-935. DOI:10.1128/AAC.00176-13 |
| [17] |
Marrakchi H, Dewolf WE Jr, Quinn C, et al. Characterization of Streptococcus pneumoniae enoyl-(acyl-carrier protein) reductase (FabK)[J]. The Biochemical Journal, 2003, 370(3): 1055-1062. DOI:10.1042/bj20021699 |
| [18] |
Zhu L, Bi HK, Ma JC, et al. The two functional enoyl-acyl carrier protein reductases of Enterococcus faecalis do not mediate triclosan resistance[J]. mBio, 2013, 4(5): e00613-13. |
| [19] |
Bi HK, Zhu L, Wang HH, et al. Inefficient translation renders the Enterococcus faecalis fabK enoyl-acyl carrier protein reductase phenotypically cryptic[J]. Journal of Bacteriology, 2014, 196(1): 170-179. DOI:10.1128/JB.01148-13 |
| [20] |
Heath RJ, Su N, Murphy CK, et al. The Enoyl-[J]. Journal of Biological Chemistry, 2000, 275(51): 40128-40133. DOI:10.1074/jbc.M005611200 |
| [21] |
Zhu L, Lin JS, Ma JC, et al. The triclosan resistance of Pseudomonas aeruginosa PAO1 is due to FabV, a triclosan-resistant enoyl-acyl carrier protein reductase[J]. Antimicrobial Agents and Chemotherapy, 2010, 54(2): 689-698. DOI:10.1128/AAC.01152-09 |
| [22] |
Massengo-Tiasse RP, Cronan JE. Vibrio cholerae FabV defines a new class of enoyl-acyl carrier protein reductase[J]. The Journal of Biological Chemistry, 2008, 283(3): 1308-1316. DOI:10.1074/jbc.M708171200 |
| [23] |
Price AC, Zhang YM, Rock CO, et al. Structure of β-ketoacyl-[J]. Biochemistry, 2001, 40(43): 12772-12781. DOI:10.1021/bi010737g |
| [24] |
Hoang TT, Sullivan SA, Cusick JK, et al. β-ketoacyl acyl carrier protein reductase (FabG) activity of the fatty acid biosynthetic pathway is a determining factor of 3-oxo-homoserine lactone acyl chain lengths[J]. Microbiology, 2002, 148(12): 3849-3856. DOI:10.1099/00221287-148-12-3849 |
| [25] |
Oppermann U, Filling C, Hult M, et al. Short-chain dehydrogenases/reductases (SDR): the 2002 update[J]. Chemico-Biological Interactions, 2003, 143: 247-253. |
| [26] |
Lai CY, Cronan JE. Isolation and characterization of β-ketoacyl-acyl carrier protein reductase (fabG) mutants of Escherichia coli and Salmonella enterica serovar typhimurium[J]. Journal of Bacteriology, 2004, 186(6): 1869-1878. DOI:10.1128/JB.186.6.1869-1878.2004 |
| [27] |
Wang HH, Cronan JE. Only one of the two annotated Lactococcus lactis fabG genes encodes a functional β-ketoacyl-acyl carrier protein reductase[J]. Biochemistry, 2004, 43(37): 11782-11789. DOI:10.1021/bi0487600 |
| [28] |
Feng SX, Ma JC, Yang J, et al. Ralstonia solanacearum fatty acid composition is determined by interaction of two 3-ketoacyl-acyl carrier protein reductases encoded on separate replicons[J]. BMC Microbiology, 2015, 15: 223-237. DOI:10.1186/s12866-015-0554-x |
| [29] |
Zhang HW, Liu YH, Yao CH, et al. FabG can function as PhaB for poly-3-hydroxybutyrate biosynthesis in photosynthetic cyanobacteria Synechocystis sp. PCC 6803[J]. Bioengineered, 2017, 8(6): 707-715. DOI:10.1080/21655979.2017.1317574 |
| [30] |
Ren Q, Sierro N, Kellerhals M, et al. Properties of engineered poly-3-hydroxyalkanoates produced in recombinant Escherichia coli strains[J]. Applied and Environmental Microbiology, 2000, 66(4): 1311-1320. DOI:10.1128/AEM.66.4.1311-1320.2000 |
| [31] |
Parish T, Roberts G, Laval F, et al. Functional complementation of the essential gene fabG1 of Mycobacterium tuberculosis by Mycobacterium smegmatis fabG but not Escherichia coli fabG[J]. Journal of Bacteriology, 2007, 189(10): 3721-3728. DOI:10.1128/JB.01740-06 |
| [32] |
Gurvitz A. The essential mycobacterial genes, fabG1and fabG4, encode 3-oxoacyl-thioester reductases that are functional in yeast mitochondrial fatty acid synthase type 2[J]. Molecular Genetics and Genomics, 2009, 282(4): 407-416. DOI:10.1007/s00438-009-0474-2 |
| [33] |
Singh R, Reynolds KA. Characterization of FabG and FabI of the Streptomyces coelicolor dissociated fatty acid synthase[J]. ChemBioChem, 2015, 16(4): 631-640. DOI:10.1002/cbic.201402670 |
| [34] |
Filip'echeva YA, Shelud'ko AV, Prilipov AG, et al. Plasmid AZOBR_p1-borne fabG gene for putative 3-oxoacyl-[J]. Canadian Journal of Microbiology, 2018, 64(2): 107-118. DOI:10.1139/cjm-2017-0561 |
| [35] |
Kovtunov EA, Shelud'ko AV, Chernyshova MP, et al. Mutants of bacterium Azospirillum brasilense Sp245 with Omegon insertion in mmsB or fabG genes of lipid metabolism are defective in motility and flagellation[J]. Genetika, 2013, 49(11): 1270-1275. |
| [36] |
Miller DJ, Zhang YM, Rock CO, et al. Structure of RhlG, an essential β-ketoacyl reductase in the rhamnolipid biosynthetic pathway of Pseudomonas aeruginosa[J]. The Journal of Biological Chemistry, 2006, 281(26): 18025-18032. DOI:10.1074/jbc.M601687200 |
| [37] |
López-Lara IM, Geiger O. The nodulation protein NodG shows the enzymatic activity of an 3-oxoacyl-acyl carrier protein reductase[J]. Molecular Plant-Microbe Interactions, 2001, 14(3): 349-357. DOI:10.1094/MPMI.2001.14.3.349 |
| [38] |
Mao YH, Li F, Ma JC, et al. Sinorhizobium meliloti functionally replaces 3-oxoacyl-acyl carrier protein reductase (FabG) by overexpressing NodG during fatty acid synthesis[J]. Molecular Plant-Microbe Interactions, 2016, 29(6): 458-467. DOI:10.1094/MPMI-07-15-0148-R |
| [39] |
Liu Y, Zhu JB, Yu GQ, et al. The enoyl-ACP reductase gene, fabI1, of Sinorhizobium meliloti is involved in salt tolerance, swarming mobility and nodulation efficiency[J]. Chinese Science Bulletin, 2010, 55(3): 259-262. |
| [40] |
Pillai S, Rajagopal C, Kapoor M, et al. Functional characterization of β-ketoacyl-ACP reductase (FabG) from Plasmodium falciparum[J]. Biochemical and Biophysical Research Communications, 2003, 303(1): 387-392. DOI:10.1016/S0006-291X(03)00321-8 |
| [41] |
Srinivas S, Cronan JE. An eightresidue deletion in Escherichia coli FabG causes temperature-sensitive growth and lipid synthesis plus resistance to the calmodulin inhibitor trifluoperazine[J]. Journal of Bacteriology, 2017, 199(10): e00074-17. |
| [42] |
Dutta D, Bhattacharyya S, Roychowdhury A, et al. Crystal structure of hexanoyl-CoA bound to β-ketoacyl reductase FabG4 of Mycobacterium tuberculosis[J]. The Biochemical Journal, 2013, 450(1): 127-139. DOI:10.1042/BJ20121107 |
| [43] |
Blaise M, van Wyk N, Banères-Roquet F, et al. Binding of NADP+ triggers an open-to-closed transition in a mycobacterial FabG β-ketoacyl-ACP reductase[J]. The Biochemical Journal, 2017, 474(6): 907-921. DOI:10.1042/BCJ20161052 |
| [44] |
Cukier CD, Hope AG, Elamin AA, et al. Discovery of an allosteric inhibitor binding site in 3-oxo-acyl-ACP reductase from Pseudomonas aeruginosa[J]. ACS Chemical Biology, 2013, 8(11): 2518-2527. DOI:10.1021/cb4005063 |
| [45] |
Hou J, Zheng HP, Chruszcz M, et al. Dissecting the structural elements for the activation of β-ketoacyl-(acyl carrier protein) reductase from Vibrio cholerae[J]. Journal of Bacteriology, 2016, 198(3): 463-476. DOI:10.1128/JB.00360-15 |
| [46] |
Heath RJ, Rock CO. Fatty acid biosynthesis as a target for novel antibacterials[J]. Current Opinion in Investigational Drugs, 2004, 5(2): 146-153. |
| [47] |
Wallace J, Bowlin NO, Mills DM, et al. Discovery of bacterial fatty acid synthase type II inhibitors using a novel cellular bioluminescent reporter assay[J]. Antimicrobial Agents and Chemotherapy, 2015, 59(9): 5775-5787. DOI:10.1128/AAC.00686-15 |
| [48] |
Jin LM, Liu P, Tian WJ. Bacterial fatty acid biosynthesis enzymes-drug targets for antibacterial agent screen[J]. Journal of Shenyang Pharmaceutical University, 2006, 23(12): 814-818. (in Chinese) 金黎明, 刘鹏, 田文杰. 细菌脂肪酸合成酶--抗菌药物的筛选靶点[J]. 沈阳药科大学学报, 2006, 23(12): 814-818. DOI:10.3969/j.issn.1006-2858.2006.12.018 |
| [49] |
Nanson JD, Forwood JK. Structural characterisation of FabG from Yersinia pestis, a key component of bacterial fatty acid synthesis[J]. PLoS One, 2015, 10(11): e0141543. DOI:10.1371/journal.pone.0141543 |
| [50] |
Zhang YM, Rock CO. Evaluation of epigallocatechin gallate and related plant polyphenols as inhibitors of the FabG and FabI reductases of bacterial type II fatty-acid synthase[J]. The Journal of Biological Chemistry, 2004, 279(30): 30994-31001. DOI:10.1074/jbc.M403697200 |
| [51] |
Wu D, Wu XD, You XF, et al. Inhibitory effects on bacterial growth and β-ketoacyl-ACP reductase by different species of maple leaf extracts and tannic acid[J]. Phytotherapy Research, 2010, 24(Suppl 1): S35-S41. |
| [52] |
Huang H, Wu D, Tian WX, et al. Antimicrobial effect by extracts of rhizome of Alpinia officinarum Hance may relate to its inhibition of β-ketoacyl-ACP reductase[J]. Journal of Enzyme Inhibition and Medicinal Chemistry, 2008, 23(3): 362-368. DOI:10.1080/14756360701622099 |
| [53] |
Kristan K, Bratkovič T, Sova M, et al. Novel inhibitors of β-ketoacyl-ACP reductase from Escherichia coli[J]. Chemico-Biological Interactions, 2009, 178(1/3): 310-316. |
| [54] |
Nomura CT, Taguchi K, Gan ZH, et al. Expression of 3-ketoacyl-acyl carrier protein reductase (fabG) genes enhances production of polyhydroxyalkanoate copolymer from glucose in recombinant Escherichia coli JM109[J]. Applied and Environmental Microbiology, 2005, 71(8): 4297-4306. DOI:10.1128/AEM.71.8.4297-4306.2005 |
| [55] |
Vo MT, Lee KW, Jung YM, et al. Comparative effect of overexpressed phaJ and fabG genes supplementing (R)-3-hydroxyalkanoate monomer units on biosynthesis of mcl-polyhydroxyalkanoate in Pseudomonas putida KCTC1639[J]. Journal of Bioscience and Bioengineering, 2008, 106(1): 95-98. DOI:10.1263/jbb.106.95 |
| [56] |
Wang MZ, Schaefer AL, Dandekar AA, et al. Quorum sensing and policing of Pseudomonas aeruginosa social cheaters[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(7): 2187-2191. DOI:10.1073/pnas.1500704112 |
| [57] |
Zhou L, Yu YH, Chen XP, et al. The multiple DSF-family QS signals are synthesized from carbohydrate and branched-chain amino acids via the FAS elongation cycle[J]. Scientific Reports, 2015, 5: 13294. DOI:10.1038/srep13294 |
| [58] |
Wang LH, He YW, Gao YF, et al. A bacterial cell-cell communication signal with cross-kingdom structural analogues[J]. Molecular Microbiology, 2004, 51(3): 903-912. |
 2019, Vol. 46
2019, Vol. 46