
扩展功能
文章信息
- 侯佳林, 聂唱, Venki Perumal, 肖湘, 王风平
- HOU Jia-Lin, NIE Chang, Venki Perumal, XIAO Xiang, WANG Feng-Ping
- 基于宏基因组学重建来自深海热液喷口的Epsilonproteobacteria基因组及其代谢分析
- Metabolic analysis of Epsilonproteobacteria genomes reconstructed from the deep sea hydrothermal vent chimney based on metagenomic technology
- 微生物学通报, 2018, 45(9): 1843-1852
- Microbiology China, 2018, 45(9): 1843-1852
- DOI: 10.13344/j.microbiol.china.180100
-
文章历史
- 收稿日期: 2018-01-31
- 接受日期: 2018-03-29
- 网络首发日期(www.cnki.net): 2018-05-15
2. 上海交通大学 微生物代谢国家重点实验室 上海 200240;
3. 上海交通大学 海洋工程国家重点实验室 上海 200240
2. State Key Laboratory of Microbial Metabolism, Shanghai Jiao Tong University, Shanghai 200240, China;
3. State Key Laboratory of Ocean Engineering, Ocean and Civil Engineering, Shanghai Jiao Tong University, Shanghai 200240, China
Epsilonproteobacteria纲是变形菌门中一支独特的细菌类群,最新的研究提出将所有Epsilonproteobacteria和部分的Deltaproteobacteria重新归为一个单独的门水平的分类单元Epsilonbacteraeota[1]。Epsilonproteobacteria最初是作为一种致病菌被发现的,并且在临床医疗领域得到了长期的重点关注[2],如导致胃溃疡的幽门螺旋杆菌(Helicobacter pylori),据估计全球至少有50%的人群已被其感染。近些年随着分子测序技术的不断发展,越来越多的非致病性Epsilonproteobacteria在各种类型的极端环境中被发现,比如深海热液口、泥火山、陆地热泉和硫化洞穴等[3-10]。除了可以营自由生活以外,部分Epsilonproteobacteria还可以寄生于各种无脊椎动物(如热液管状蠕虫)的体内或者体表[11-12]。在深海热液微生物群落中,Epsilonproteobacteria有时会占到全部物种的90%以上并形成大规模的菌席[10, 13]。基于为数不多的可培养代表种的研究结果,绝大部分非致病性Epsilonproteobacteria都为无机化能自养营养型,如Nitratifractor salsuginis、Nautilia nitratireducens、Hydrogenimonas thermophila和Caminibacter profundus可以利用氢气作为电子供体[3-5, 7],而Sulfurovum lithotrophicum和Sulfurimonas paralvinellae则能够通过氧化单质硫和硫代硫酸盐提供能量[4, 14]。另外,也有个别类群如Nautilia nitratireducens为混合营养或者化能异养类型,能够利用甲酸、乙酸等简单小分子有机物[5, 15]。所有的非致病性Epsilonproteobacteria都可以利用硝酸盐作为电子受体,此外它们中的部分菌也能够将产生的电子传递至氧气和单质硫。因此,非致病性Epsilonproteobacteria被认为在其所栖息微生物群落中很可能会作为初级生产者而发挥着重要的生态功能[16-17]。有研究表明,来自于深海热液环境的化能自养Epsilonproteobacteria很可能是其现代致病性类群的祖先[2, 15]。
由于现代微生物培养条件和技术的限制,大部分极端自然环境中的Epsilonproteobacteria都基于16S rRNA基因被发现,只有少数的代表种已经被分离培养,而在自然环境中超过99%的微生物尚不可培养,这极大地限制了对其生理代谢特点进行深入研究。现代的宏基因组学Binning技术可以直接从环境基因组中分装并重新构建出高质量的微生物基因组,这为进一步研究这些非培养微生物的进化分类地位和代谢潜能提供了新的方法和途径,也为更加深入地理解和评估其生态功能以及在全球化学元素循环中所发挥的作用提供了研究基础。
本研究利用宏基因组学Binning方法对来自于东太平洋海隆深海热液硫化物烟囱体的样本进行深入分析,重新构建获得4个高质量的Epsilonproteobacteria基因组,并对其系统发育地位和代谢潜能进行全面而深入的研究。
1 材料与方法 1.1 主要试剂和仪器所有试剂均购自生工生物工程(上海)股份有限公司。DNA提取缓冲液(均pH 8.0的100 mmol/L Tri-HCl、EDTA钠、磷酸钠,1.5 mmol/L NaCl,1%十六甲基三甲基铵溴化物),溶解酶(20 g/L),蛋白酶K (20 g/L),SDS溶液(20%),醋酸钠(pH 5.2)。Illumina HiSeq 2000平台,深圳华大基因公司。
1.2 样品信息及DNA提取测序研究样本采集于2013年12月至2014年1月的AT26-10航次,采集地点为东太平洋海隆北纬9°-10° L-vent深海热液区域中一个处于活跃喷发状态的硫化物烟囱体,经测定其喷发的热液温度为231 ℃,富含大量的硫化氢和氢气。样本采集后立即储存于-70 ℃条件下,随后使用干冰转运至实验室进行后续处理。样本DNA提取采用基于SDS的方法[18]:300 mg烟囱体样本研磨后混合等量0.1 mm的玻璃珠并加入670 µL DNA提取缓冲液,3 000×g离心5 min后加入组织裂解液并混匀。随后再加入50 µL溶解酶和蛋白酶K混合液,37 ℃静置30 min。加入70 µL SDS后,65 ℃静置2 h。经1 000×g离心10 min后收集上清液,剩余样品使用同样方法再提取一遍。上清液混合等量的苯酚:氯仿:异戊基乙醇溶液(24:25:1,体积比),离心后收集上清液,加入冰丙基乙醇和0.3 mol/L醋酸钠4 ℃沉淀过夜。经16 000×g离心20 min后去除上清液收集底部DNA,经70%乙醇冲洗后加入蒸馏水使其重新悬浮。同一样本分3份独立提取出DNA后混合进行下游分析。
1.3 测序数据组装和基因组Binning宏基因组由深圳华大基因公司使用Illumina HiSeq 2000平台进行测序,原始读长为2×100 bp。数据由公司返回后,使用Sickle软件(https://github.com/najoshi/sickle)对其进行质量控制,对测序质量低的碱基使用参数--se进行剪切和删除。随后使用IDBA-UD[19]进行拼接,参数为--mink 52、--maxk 92、--step 8。对于拼接后得到的长度大于3 kb的拼接片段,通过比较四核苷酸频率特征,使用ESOM[20]进行Binning,并使用Bowtie2[21]将原始的DNA短读段比对到拼接片段上,计算每条拼接片段的测序深度。随后使用mmgenome[22]对这些初步分装获得的基因组进行基于四核苷酸频率、G+C%含量和测序深度的整合分析,手动检查并进行更加精细的基因组片段reBinning,最后使用checkM[23]对其进行初步的物种鉴定以及完整度和污染度的评估。
1.4 功能基因的预测和注释对于上述拼接和分装得到的高质量基因组,使用Prodigal[24]进行蛋白编码序列(CDS)的预测,参数为-meta,同时分别使用RNAmmer[25]和checkM[23]进行16S rRNA基因的预测。对于蛋白质编码序列,将其比对已知的注释信息数据库NCBI NR、KEGG[26]和COG[27]得到功能注释,并手动检测注释的准确性,使用阈值为E-values < 10–5。
1.5 系统发育树的构建为了确定分装得到的基因组的物种分类信息,选取了NCBI物种数据库已发表的78个Epsilonproteobacteria代表种作为参考基因组(包括所有的非致病种,以及Helicobacter、Campylobacter和Arcobacter 3个致病属的代表种各5个),并选取Aquifex aeolicus VF5作为外群用于构建系统发育树。通过文献调研,使用38个古菌和细菌共有的保守标记基因构建系统发育树以确定各基因组的进化地位[28]。对于上述的每一个保守单拷贝基因的蛋白序列分别进行相同的操作:合并目标基因组和参考基因组中对应的蛋白序列,使用MAFFT[29]进行序列比对,参数默认。随后根据其所属的基因组,将比对好的每一组序列按照相同顺序重新首位连接,用空位补齐缺失的蛋白。将上述连接后得到的序列使用trimal进行degap,使用-automated1参数,适合后续极大似然法系统发育树的分析。手动检查并删除比对时引入的大量空位。使用FastTree2[30]基于Whelan-And-Goldman (WAG)模型和CAT预测法构建最大似然系统发育树,自展值通过1000次重复计算得到。
2 结果与分析 2.1 重构Epsilonproteobacteria基因组的基本信息经过精细的拼装和Binning,从研究的深海热液烟囱体样本中重新构建了4个高质量的基因组。经checkM检测,它们的完整度都在90%以上且污染度在1%以下,基因组大小在1.32 Mb-1.57 Mb (表 1),且checkM内置系统发育分析结果表明这4个基因组都属于Epsilonproteobacteria。4个重构基因组都已上传DDBJ/ENA/GenBank数据库,BioProject ID为PRJNA430147,GenBank登录号为PUXW00000000-PUXZ00000000。
| Bin ID | Bin225 | Bin51 | Bin54 | Bin189 |
| Genome size (bp) | 1 578 483 | 1 440 596 | 1 391 252 | 1 321 064 |
| Contigs | 59 | 20 | 94 | 156 |
| N50 | 45 720 | 120 868 | 22 545 | 10 677 |
| Mean contig length (bp) | 26 753 | 72 029 | 14 800 | 8 468 |
| Completeness | 98.58 | 97.97 | 94.11 | 90.11 |
| Contamination | 0.81 | 0.41 | 0 | 0.41 |
| Strain heterogeneity | 0 | 0 | 0 | 0 |
| GC | 53.98 | 56.1 | 48.96 | 43.83 |
| Predicted genes | 1 639 | 1 522 | 1 564 | 1 347 |
| Metabolism | 688 | 663 | 666 | 587 |
| Genetic information processing | 172 | 169 | 176 | 153 |
| Environmental information processing | 68 | 66 | 57 | 35 |
| Others | 76 | 53 | 18 | 7 |
| Unknown | 635 | 571 | 647 | 565 |
在宏基因组中正确地组装高度相似的16S rRNA编码基因一直是宏基因组学研究中亟待解决的问题之一,而Epsilonproteobacteria在本研究的原始样本中为优势类群,必然存在大量近源类群,这可能是造成4个重建的基因组中均未检测到16S rRNA编码基因的重要原因。因此,我们通过38个串联的保守基因蛋白质序列对其进行系统发育树的构建,从而确定这些基因组在Epsilonproteobacteria类群中的具体进化地位(图 1)。结果显示,Bin225、Bin51和Bin54之间具有相对密切的进化关系,而且它们最近的亲缘物种都为Nitratiruptor sp. SB155-2;而Bin189与其他3个基因组具有较远的进化距离,并且与Caminibacter mediatianticus、Nautilia profundicola以及Lebetimonas属中的类群具有相对较近的系统发育关系,但其具体的分类和进化地位仍不能确定。
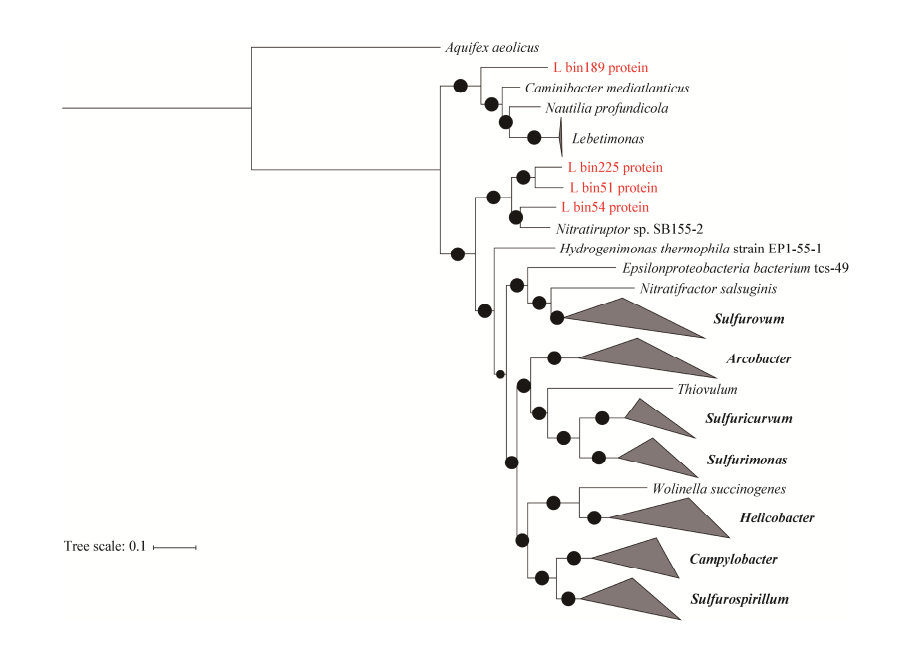
|
| 图 1 基于38个串联保守蛋白序列对4个重构Epsilonproteobacteria基因组的系统发育分析 Figure 1 Phylogenetic analysis of 4 reconstructed Epsilonproteobacteria genomes based on 38 concatenated conversed marker protein sequences 注:自展值> 75的显示为黑色圆点,其直径与自展的大小值成正比. Note: Bootstrap larger than 75 were presented and in direct proportion to the diameter of the black dots on the brunches. |
|
|

|
| 图 2 重构4个Epsilonproteobacteria的关键代谢途径 Figure 2 Key metabolic pathways of four reconstructed Epsilonproteobacteria 注:黑色表示存在于所有基因组的代谢途径,蓝色表示Bin189特有代谢途径,而红色表示在其余的3个基因组中至少存在2个及以上的代谢途径. The black pathways represent those are present in all genomes and the blue ones represent pathways that only are detected in Bin189. The red pathways represent those exist at least 2 of the other 3 genomes. |
|
|
在重建的4个来自深海热液烟囱体的Epsilonproteobacteria基因组中都检测到了几乎完整的Reductive TCA碳固定途径,其中包括与CO2固定直接相关的关键酶,如por (编码丙酮酸铁氧还蛋白氧化还原酶)、korA/B/C/D (编码2-酮戊二酸铁氧还蛋白氧化还原酶)和sdh/frdA/B/C/D (编码琥珀酸脱氢酶/延胡索酸还原酶)。完整的aclA/B (编码ATP柠檬酸裂解酶)也在Bin189和Bin54中都被发现,而在Bin225和Bin51中仅发现了aclB。除此之外,在4个Epsilonproteobacteria基因组中也都发现了完整的非氧化磷酸戊糖途径和缺失己糖激酶的糖酵解途径,而pyk (编码丙酮酸激酶)只在Bin189中被发现。
在硫代谢方面,所有的基因组都检测到了sqr (编码硫化物:醌氧化还原酶),其中Bin225和Bin54具有3个相同的复制基因,而Bin189和Bin51分别具有2个和1个。与Sox系统(硫代硫酸盐氧化系统)相关的编码基因soxA/B/X/Y/Z只在Bin51和Bin54中被发现。虽然sat (编码硫酸盐腺苷转移酶)同样也在4个基因组中都被发现,而与异化硫酸盐还原相关其他功能基因apr (编码腺苷酰硫酸还原酶)和dsr (编码亚硫酸盐还原酶)并未在任何基因组中被检测到。
在氮代谢方面,除了Bin189以外,在其他的3个基因组中都发现了完整的反硝化途径相关的功能基因,包括napA/B (编码周质硝酸盐还原酶)、nirS (编码亚硝酸还原酶)、norB/C (编码一氧化氮还原酶)和nosZ (编码一氧化二氮还原酶)。而除了nap以外,其他与异化硝酸盐还原相关的功能基因在所有的基因组中都未被检测到。
对于氢化酶,在Bin54和Bin51中都发现了可以利用醌体作为电子供体的1型Ni-Fe氢化酶基因hydA/B和2型氢化酶小亚基hybO,而Bin189和Bin225中未发现任何与氢化酶相关的基因。
与金属离子、无机矿物相关的转运子以及假定的ABC转运子基因在4个Epsilonproteobacteria基因组中被大量检测到,如modA/B/C (编码钼酸盐转运系统)、malE/F/D (编码磷脂转运子)和znuA/B/C(编码锌离子转运子)在所有的基因组中都被发现。同时,与次级代谢产物输出相关的转运子基因lptA/B/D/G (编码脂多糖输出转运系统)也被发现存在于所有的4个基因组中。此外,Bin189还具有PstA/B/C/S (编码磷酸盐/氨基酸转运子)、livK/G/F (编码支链氨基酸转运子)、dctP/Q/M (编码C4-二羧酸阴离子转运子)和cbiN/Q/M/O (编码镍/钴转运子)。
在4个重构的基因组中都检测到了多种与分泌系统相关的完整功能基因,包括gsp (普通分泌系统)、sec (Sec-SRP分泌系统)和tat (Tat分泌系统)。除此之外,在Bin255和Bin51中还发现了flh和fli (Ⅲ型鞭毛相关分泌系统)。
对于细胞的移动能力,结果显示Bin255和Bin51中具有完整的趋化性相关的功能基因cheA/W/R/Y/V,与鞭毛移动、分泌、粘附和调控相关的基因如flg、mot、fli等,还有与纤毛移动相关的基因pilT/B/C/D。而Bin189和Bin54中仅发现纤毛振颤移动蛋白的编码基因pliT和前体肽酶pliD。此外,aer (趋氧性受体)在所有的4个基因组中都被发现。
3 讨论化能自养Epsilonproteobacteria经常在深海热液环境中的微生物群落中作为优势物种被发现,有时甚至会形成大规模的微生物菌席覆盖在热液烟囱体表面[10, 13]。作为深海热液微生物群落的初级生产者之一,这些化能自养微生物也被认为是整个深海活跃热液生态系统得以存在和发展的重要基础[16-17]。但是由于现阶段对原位深海热液环境模拟技术的困难和其本身嗜热、对氧气敏感、化能自养等不易分离培养的生理生化特点,我们对深海热液Epsilonproteobacteria分类进化地位和代谢途径的认识还十分有限,目前绝大多数研究都仅仅是基于16S rRNA基因的检测。随着日益发展的宏基因组技术和生物信息学方法,研究者们有机会可以在基因组层面更加深入地探究这些热液未培养微生物的代谢潜能及其在原位环境中可能发挥的生态功能。虽然一些来自热液可培养Epsilonproteobacteria的预测基因的生化功能已经在之前的研究中得到了实验验证[31-32],但是对于更多新发现的来自未培养物种的功能基因仍需实验证明,而本文所预测的功能基因以及重建的基因组为进一步研究其生理生化性质提供了材料和基础。当然,现阶段所有功能基因预测的方法都是基于保守结构域的序列相似性,因此不能排除与已知基因具有完全不同或未知结构域但具有相同生化功能的同工酶基因存在的可能性,而对于这些同工酶的发现可能更加依赖未来原位活性检测技术、蛋白质功能检测技术以及测序技术的发展和进步。
在本研究中,我们利用Binning技术从来自东太平洋海隆深海热液烟囱体的样本中重构了4个高质量的Epsilonproteobacteria基因组,系统发育分析结果表明其中Bin54很可能属于Nitratiruptor属,而Bin51和Bin225虽然也与该属具有较近的进化关系,但其确切的分类地位仍需要更多的参考基因组来支持。Bin189在系统发育树的位置相对靠近根部,与已发表的任何Epsilonproteobacteria基因组没有较近的进化关系,因此可能属于一支相对古老的新发现的深海热液Epsilonproteobacteria类群。
所有分离自深海热液环境的Epsilonproteobacteria都可以氧化还原性硫化物和(或)氢气,并且利用rTCA固碳途径进行化能自养生长,如Nitratiruptor、Hydrogenimonas、Sulfurovum属等[15, 17]。类似地,在本研究中重构的4个基因组都具有碳固定rTCA途径,说明深海热液环境中大量未知的、未得到纯培养的Epsilonproteobacteria很可能也同样可以进行化能自养的固碳过程,可能在深海生物地球化学碳元素循环以及维持整个热液生态系统的稳定中发挥重要作用。此外,这些基因组也都具有糖酵解和非氧化磷酸戊糖的代谢潜能,表明这些来自热液的Epsilonproteobacteria可以合成生命所必需的核苷酸和各种氨基酸,同时也暗示它们具有异养代谢的可能性。
所有重构的Epsilonproteobacteria都具有利用sqr氧化硫化物的潜能,但除Bin189之外,其他3个基因组都具有完整的反硝化途径,其中Bin51和Bin54还具有完整的Sox系统和氢化酶,这些也都与Nitratiruptor sp. SB155-2的代谢特征一致[2],说明它们还可以利用硫代硫酸盐、单质硫和氢气作为能源,并且将产生的电子通过反硝化传递至硝酸盐生成氮气。同时这也支持了系统发育分析的结果,Bin51很可能属于Nitratiruptor属,而Bin54和Bin225也与Nitratiruptor属在进化上具有较近的亲缘关系。
除了大量的金属和无机离子转运通道及ABC转运子以外,所有重构的基因组中都含有次级代谢产物转运系统,比如lpt脂多糖转出系统。另外,它们还都具有强大的细菌分泌系统。由此说明这些热液Epsilonproteobacteria很可能会在生长的不同阶段向胞外环境分泌次级代谢产物,使其能够形成具有一定机械强度的三级结构,以抵抗超高温羽流的直接冲击或者波动影响。同时,这些次级代谢产物也可以为其他微生物提供相对稳定的栖息环境和能源物质,比如之前在Loki castle热液区域烟囱体微生物群落的研究中观察到Bacteroidetes附着生长于Epsilonproteobacteria产生的纤维状多糖细丝上[10],这可能也是深海热液环境大规模微生物席形成的重要原因之一。与其他基因组不同的是Bin189还具有转运氨基酸和C4-二羧酸的潜能[33-34],Bin189很可能会利用这些胞外有机物作为碳源和能源物质进行化能异养代谢。
鉴于完整趋化性和鞭毛移动相关的功能基因,Bin256和Bin51被推测很可能具有自由移动的能力,这一点不同于其他2个基因组。当然,这也有可能是由于基因组的非完整性所造成的。此外,趋氧性受体说明这些深海热液Epsilonproteobacteria类似于其已分离的近源物种,对氧气都具有较高的敏感性,很可能为厌氧或者微耐氧微生物。
4 结论非致病性Epsilonproteobacteria广泛分布于不同的自然环境中,在一些极端生境中常常作为优势物种被发现,比如陆地热泉和深海热液喷口。它们当中的绝大多数为化能自养类型且具有多样的代谢潜能,在碳、氮、硫等地球化学元素循环中发挥着重要的作用。本研究对东太平洋海隆深海热液烟囱体宏基因组样本进行拼接和Binning,从中重构出4个高质量的Epsilonproteobacteria基因组Bin225、Bin51、Bin54和Bin189并进行了详细的系统发育和代谢途径分析。结果显示,Bin189可能为一种新型深海热液兼性化能营养型Epsilonproteobacteria类群,除了可以利用完整的rTCA碳固定途径和sqr硫氧化进行化能自养生长以外,同时也具有转运和利用胞外氨基酸和有机物进行异养代谢的潜力;而其他3个基因组与Nitratiruptor属具有相对较近的进化关系和相似的代谢特征,它们都具有完整的反硝化途径,其中2个基因组还包括Sox系统和氢化酶,具有氧化多种硫化物和氢气的潜能。此外,所有重构的Epsilonproteobacteria基因组中都具有脂多糖输出系统和强大的分泌系统,说明其很可能在深海热液微生物群落的形成和发展中发挥着重要作用。本研究对4个重构自深海热液系统的Epsilonproteobacteria基因组的系统发育地位和代谢潜能进行了深入的探索研究,这不仅有助于更加深入理解Epsilonproteobacteria在深海热液生态系统和地球化学元素循环中所发挥的作用,同时也为今后研究非培养Epsilonproteobacteria的代谢特征以及分离培养条件的优化提供了更加丰富的数据支持和理论基础。
| [1] |
Waite DW, Vanwonterghem I, Rinke C, et al. Comparative genomic analysis of the class Epsilonproteobacteria and proposed reclassification to epsilonbacteraeota (phyl. nov.)[J]. Frontiers in Microbiology, 2017, 8: 682. DOI:10.3389/fmicb.2017.00682 |
| [2] |
Nakagawa S, Takaki Y, Shimamura S, et al. Deep-sea vent ε-proteobacterial genomes provide insights into emergence of pathogens[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(29): 12146-12150. DOI:10.1073/pnas.0700687104 |
| [3] |
Nakagawa S, Takai K, Inagaki F, et al. Nitratiruptor tergarcus gen. nov., sp. nov. and Nitratifractor salsuginis gen. nov., sp. nov., nitrate-reducing chemolithoautotrophs of the ε-Proteobacteria isolated from a deep-sea hydrothermal system in the Mid-Okinawa Trough[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(2): 925-933. DOI:10.1099/ijs.0.63480-0 |
| [4] |
Inagaki F, Takai K, Nealson KH, et al. Sulfurovum lithotrophicum gen. nov., sp. nov., a novel sulfur-oxidizing chemolithoautotroph within the ε-Proteobacteria isolated from Okinawa Trough hydrothermal sediments[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(5): 1477-1482. DOI:10.1099/ijs.0.03042-0 |
| [5] |
Pérez-Rodríguez I, Ricci J, Voordeckers JW, et al. Nautilia nitratireducens sp. nov., a thermophilic, anaerobic, chemosynthetic, nitrate-ammonifying bacterium isolated from a deep-sea hydrothermal vent[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(5): 1182-1186. DOI:10.1099/ijs.0.013904-0 |
| [6] |
Keller AH, Schleinitz KM, Starke R, et al. Metagenome-based metabolic reconstruction reveals the ecophysiological function of Epsilonproteobacteria in a hydrocarbon-contaminated sulfidic aquifer[J]. Frontiers in Microbiology, 2015, 6: 1396. |
| [7] |
Voordeckers JW, Starovoytov V, Vetriani C. Caminibacter mediatlanticus sp. nov., a thermophilic, chemolithoautotrophic, nitrate-ammonifying bacterium isolated from a deep-sea hydrothermal vent on the Mid-Atlantic Ridge[J[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(2): 773-779. DOI:10.1099/ijs.0.63430-0 |
| [8] |
Meyer JL, Huber JA. Strain-level genomic variation in natural populations of Lebetimonas from an erupting deep-sea volcano[J]. The ISME Jouanal, 2014, 8(4): 867-880. DOI:10.1038/ismej.2013.206 |
| [9] |
Goris T, Schubert T, Gadkari J, et al. Insights into organohalide respiration and the versatile catabolism of Sulfurospirillum multivorans gained from comparative genomics and physiological studies[J]. Environmental Microbiology, 2014, 16(11): 3562-3580. DOI:10.1111/emi.2014.16.issue-11 |
| [10] |
Stokke R, Dahle H, Roalkvam I, et al. Functional interactions among filamentous Epsilonproteobacteria and Bacteroidetes in a deep-sea hydrothermal vent biofilm[J]. Environmental Microbiology, 2015, 17(10): 4063-4077. DOI:10.1111/1462-2920.12970 |
| [11] |
Grzymski JJ, Murray AE, Campbell BJ, et al. Metagenome analysis of an extreme microbial symbiosis reveals eurythermal adaptation and metabolic flexibility[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(45): 17516-17521. DOI:10.1073/pnas.0802782105 |
| [12] |
Zbinden M, Marqué L, Gaudron SM, et al. Epsilonproteobacteria as gill epibionts of the hydrothermal vent gastropod Cyathermia naticoides (North East-Pacific Rise)[J]. Marine Biology, 2015, 162(2): 435-448. DOI:10.1007/s00227-014-2591-7 |
| [13] |
Steen IH, Dahle H, Stokke R, et al. Novel barite chimneys at the Loki's Castle Vent Field shed light on key factors shaping microbial communities and functions in hydrothermal systems[J]. Frontiers in Microbiology, 2015, 6: 1510. |
| [14] |
Takai K, Suzuki M, Nakagawa S, et al. Sulfurimonas paralvinellae sp. nov., a novel mesophilic, hydrogen- and sulfur-oxidizing chemolithoautotroph within the Epsilonproteobacteria isolated from a deep-sea hydrothermal vent polychaete nest, reclassification of Thiomicrospira denitrificans as Sulfurimonas denitrificans comb. nov. and emended description of the genus Sulfurimonas[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(8): 1725-1733. DOI:10.1099/ijs.0.64255-0 |
| [15] |
Nakagawa S, Takaki Y. Nonpathogenic Epsilonproteobacteria[A]//Encyclopedia of Life Sciences[M]. New York: John Wiley & Sons, 2009 http://www.researchgate.net/publication/230239809_Nonpathogenic_Epsilonproteobacteria
|
| [16] |
Campbell BJ, Engel AS, Porter ML, et al. The versatile ε-proteobacteria: key players in sulphidic habitats[J]. Nature Reviews Microbiology, 2006, 4(6): 458-468. DOI:10.1038/nrmicro1414 |
| [17] |
Sievert SM, Vetriani C. Chemoautotrophy at deep-sea vents: past, present, and future[J]. Oceanography, 2012, 25(1): 218-233. DOI:10.5670/oceanog |
| [18] |
He Y, Li M, Perumal V, et al. Genomic and enzymatic evidence for acetogenesis among multiple lineages of the archaeal phylum Bathyarchaeota widespread in marine sediments[J]. Nature Microbiology, 2016, 1(6): 16035. DOI:10.1038/nmicrobiol.2016.35 |
| [19] |
Peng Y, Leung HCM, Yiu SM, et al. IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth[J]. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174 |
| [20] |
Dick GJ, Andersson AF, Baker BJ, et al. Community-wide analysis of microbial genome sequence signatures[J]. Genome Biology, 2009, 10(8): R85. DOI:10.1186/gb-2009-10-8-r85 |
| [21] |
Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [22] |
Albertsen M, Hugenholtz P, Skarshewski A, et al. Genome sequences of rare, uncultured bacteria obtained by differential coverage binning of multiple metagenomes[J]. Nature Biotechnology, 2013, 31(6): 533-538. DOI:10.1038/nbt.2579 |
| [23] |
Parks DH, Imelfort M, Skennerton CT, et al. CheckM: assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes[J]. Genome Research, 2015, 25(7): 1043-1055. DOI:10.1101/gr.186072.114 |
| [24] |
Hyatt D, Chen GL, Locascio PF, et al. Prodigal: prokaryotic gene recognition and translation initiation site identification[J]. BMC Bioinformatics, 2010, 11: 119. DOI:10.1186/1471-2105-11-119 |
| [25] |
Lagesen K, Hallin P, Rødland EA, et al. RNAmmer: consistent and rapid annotation of ribosomal RNA genes[J]. Nucleic Acids Research, 2007, 35(9): 3100-3108. DOI:10.1093/nar/gkm160 |
| [26] |
Kanehisa M, Goto S. KEGG: kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27 |
| [27] |
Tatusov RL, Fedorova ND, Jackson JD, et al. The COG database: an updated version includes eukaryotes[J]. BMC Bioinformatics, 2003, 4: 41. DOI:10.1186/1471-2105-4-41 |
| [28] |
Rinke C, Schwientek P, Sczyrba A, et al. Insights into the phylogeny and coding potential of microbial dark matter[J]. Nature, 2013, 499(7459): 431-437. DOI:10.1038/nature12352 |
| [29] |
Katoh K, Misawa K, Kuma K, et al. MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform[J]. Nucleic Acids Research, 2002, 30(14): 3059-3066. DOI:10.1093/nar/gkf436 |
| [30] |
Price MN, Dehal PS, Arkin AP. FastTree 2-approximately maximum-likelihood trees for large alignments[J]. PLoS One, 2010, 5(3): e9490. DOI:10.1371/journal.pone.0009490 |
| [31] |
Yamamoto M, Nakagawa S, Shimamura S, et al. Molecular characterization of inorganic sulfur-compound metabolism in the deep-sea epsilonproteobacterium Sulfurovum sp. NBC37-1[J]. Environmental Microbiology, 2010, 12(5): 1144-1153. DOI:10.1111/emi.2010.12.issue-5 |
| [32] |
Ray S, Banerjee A, Bagchi A. Molecular level insight into the interactions of SoxC and SoxD from Epsilonproteobacteria Sulfurimonas denitrificans: a biomolecular computational approach[J]. New Delhi: Springer, 2016, 379: 401-410. |
| [33] |
Eick-Helmerich K, Braun V. Import of biopolymers into Escherichia coli: nucleotide sequences of the exbB and exbD genes are homologous to those of the tolQ and tolR genes, respectively[J]. Journal of Bacteriology, 1989, 171(9): 5117-5126. DOI:10.1128/jb.171.9.5117-5126.1989 |
| [34] |
Forward JA, Behrendt MC, Wyborn NR, et al. TRAP transporters: a new family of periplasmic solute transport systems encoded by the dctPQM genes of Rhodobacter capsulatus and by homologs in diverse gram-negative bacteria[J]. Journal of Bacteriology, 1997, 179(17): 5482-5493. DOI:10.1128/jb.179.17.5482-5493.1997 |
 2018, Vol. 45
2018, Vol. 45




