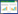扩展功能
文章信息
- 张琪, 杨思琪, 何书奋, 罗金环, 任丹丹, 丁琼
- ZHANG Qi, YANG Si-Qi, HE Shu-Fen, LUO Jin-Huan, REN Dan-Dan, DING Qiong
- 海南濒危树种保育圃白木香与降香黄檀幼苗根部真菌谱系与生态型多样性比较
- Comparison of phylogenetic diversity and ecological types of root-associated fungi on Aquilaria sinensis and Dalbergia odorifera seedlings in an endangered plant restoration nursery of Hainan Island
- 微生物学通报, 2018, 45(5): 1035-1047
- Microbiology China, 2018, 45(5): 1035-1047
- DOI: 10.13344/j.microbiol.china.170541
-
文章历史
- 收稿日期: 2017-07-17
- 接受日期: 2017-11-07
- 网络首发日期(www.cnki.net): 2017-12-27
2. 三亚市林业科学研究院 海南 三亚 572023
2. Sanya Forestry Institute, Sanya, Hainan 572023, China
根部真菌(Root-associated fungi,RAF)是指生长在植物根系组织内部、根表、根外的真菌总称,该类真菌能直接或间接对植物产生中性、有益或有害的效应[1]。RAF按其营养特性可分为共生型、腐生型和病原型真菌。共生型RAF主要包括菌根真菌和内生真菌。丛枝菌根真菌(Arbuscular mycorrhiza fungi,AMF)是最普遍的共生型RAF,最新分类系统将其分类地位由原来的球囊菌门(Glomeromycota)降为球囊菌亚门(Glomeromycotina),并划归到毛霉菌门(Mucoromycota)[2]。AMF能与90%以上的有花植物共生,促进植物对土壤养分尤其是磷的吸收,进而调节植物生长及抗逆性[3]。内生真菌是共生型RAF的又一大类群,生活在植物组织内,不引起植物组织产生明显病症,能产生具有生理活性的物质,促进或抑制植物生长[4]。腐生真菌也是RAF中的重要类群,它能分解根系的老废及死亡组织供植物利用,并对幼苗期的病原型真菌有拮抗作用[5]。病原型RAF在苗圃幼苗根部被频繁检测到,它不仅影响幼苗在苗圃中的死亡率,也极大地影响幼苗出圃后的存活率[6]。真菌是异养生物,依赖植物的光合产物提供营养。因此,真菌的生长繁殖和植物之间关系非常密切[7],尤其是对共生型的菌根真菌而言,宿主植物物种是真菌群落构建的主要驱动因子[8]。
真菌核糖体内转录间隔区(Internal transcribed spacer,ITS)是综合性能较为优越的真菌DNA条形码序列,适合真菌群落物种多样性研究[9]。真菌通用引物ITS1F/ITS4及其改良的系列引物能扩增多类真菌,如子囊菌、担子菌以及AMF,被广泛应用于环境真菌检测。然而,采用真菌通用引物检测到的RAF群落中,AMF的物种数偏低,如蔷薇属植物群落检测到的31种RAF中,AMF仅有5种[10];日本温带森林31种非外生菌根植物检测到的268种RAF中,仅有10种为AMF[11]。采用AMF特异性引物的研究却显示,根部AMF多样性极高,如泰国热带橡胶园中仅从橡胶树和木薯根系上检测到的AMF分别可达95种和87种,单株上检测到的AMF平均物种数分别为18种和23种[12]。因此,通用引物虽然能更全面地揭示根部真菌的谱系组成,但却可能会低估RAF群落中的AMF多样性。
瑞香科植物白木香(Aquilaria sinensis)和豆科植物降香黄檀(Dalbergia odorifera)均为海南珍贵乡土树种,为满足市场对珍贵乡土树种的需求,海南大力开展珍贵乡土树种的人工育苗工作,每年培育的白木香和降香黄檀幼苗分别约1 000万株和1 500万株。苗期是植物生长最脆弱的阶段,有研究者发现接种菌根真菌能有效促进幼苗的生长[13]。然而有研究表明,幼苗出圃移栽后,在新环境因子的驱动下,人工接种的真菌会随时间迁移逐渐被本土性真菌取代[14]。因此,本土性真菌繁殖体是苗圃幼苗可持续的侵染源。当前尚不清楚苗圃条件下,未接种真菌的土壤介质中是否有真菌繁殖体侵染幼苗根系,以及不同宿主植物幼苗的根部真菌群落物种组成是否存在差异。本研究采用真菌通用引物与AMF特异性引物进行研究来揭示三亚苗圃白木香和降香黄檀幼苗RAF谱系多样性,并应用基于Metadata关联的方法研究RAF的生态学特性,以及非公制多维尺度分析(Non-metric multidimensional scaling,NMDS)研究宿主植物对RAF群落物种组成的影响。此外还探讨引物选择、测序深度对揭示RAF群落物种多样性的潜在影响。研究结果对热带地区珍贵乡土树种育苗、种群恢复、资源保护与利用有重要的理论与实践意义。
1 材料与方法 1.1 试验地及基本情况海南濒危树种保育圃是三亚林业科学研究院的科研苗圃,位于海南省三亚市南部的天涯区凤凰片区内(E113°02′,N28°06′),占地约46 hm2,为海南岛最大的热带珍稀濒危树种种质资源库。本苗圃地处热带海洋季风性气候区,年平均气温为25.4 ℃,气温最高月为7月,平均气温为28.4 ℃;气温最低月为1月,平均气温为21.0 ℃。极端气温最高35.9 ℃,最低5.1 ℃。全年日照时数达2 563 h,光照充足,全年有300 d以上艳阳高照。年降雨量在600-2 000 mm,6-10月为雨季,降水量约占全年的90%,11月至翌年4月为旱季。本区母岩为花岗岩,土壤属沙壤性砖红壤,土层深厚。苗圃内地势平坦,平均海拔5-8 m。本研究中白木香与降香黄檀的育苗介质为排水良好的砂质土壤,其土壤pH为7.2-7.4,养分含量为:铵态氮16.33±6.30 mg/kg,速效磷19.85±2.38 mg/kg,速效钾19.53±4.05 mg/kg,有机质10.50±0.40 g/kg。
1.2 主要试剂和仪器Taq聚合酶、DNA纯化试剂盒,北京艾德莱生物科技有限公司;Escherichia coli DH5α感受态细胞、pMD19-T载体,宝生物工程(大连)有限公司。高通量多样品组织研磨仪,南京先欧仪器制造有限公司;PCR扩增仪,力康生物医疗科技控股有限公司;电泳仪,北京君意东方电泳仪器设备有限公司;紫外凝胶成像系统,北京五洲东方科技发展有限公司。
CTAB缓冲液配方:0.1 mol/L Tris-HCl,pH 8.0;0.02 mol/L EDTA,pH 8.0;1.4 mol/L NaCl,2% CTAB (质量体积比)。
1.3 样品采集与处理试验所用白木香与降香黄檀幼苗为2015年播种于育苗钵的,苗龄1.5年,外观健康,叶色浓绿、无明显病虫害。幼苗株高、地径、叶面积等形态学指标见表 1。沿苗圃东西方向以200 m以上间隔距离取白木香与降香黄檀幼苗各3株,去除根围育苗介质,取出完整的根系,用流水洗净根系表面附着的土壤等杂质,再以无菌水清洗2遍。以每株幼苗根颈部为中心,沿根系的10个不同方向各剪下2段长约1 cm的最末一级侧根的根段,再以无菌水清洗2遍,置于灭菌的滤纸上吸干多余的水分,将每个方向的根尖转移到一个2 mL离心管中,向管中加入50 μL CTAB提取缓冲液,放置于-80 ℃保存。
| 形态指标 Morphological indices |
白木香 Aquilaria sinensis |
降香黄檀 Dalbergia odorifera |
| 地径 Ground diameter (cm) |
0.90±0.26 | 0.53±0.08 |
| 株高 Plant height (cm) |
64.00±14.42 | 48.00±3.46 |
| 叶面积 Leaf area (cm2) |
19.69±3.69 | 12.84±1.47 |
取出冷冻的60份根尖样品(单份根尖样品重量为6.25±0.85 mg),参考黄彩微等的方法提取根系总DNA[15]。具体步骤如下:用高通量组织研磨仪研磨根尖组织至匀浆状,向离心管中加入650 μL CTAB提取缓冲液,置于65 ℃水浴1 h (每15 min振摇一次)。取出离心管,加入等体积的氯仿:异戊醇(24:1,体积比)摇匀,室温下12 000 r/min离心8 min,吸取上清液,重复操作1次。将上清液转移到1.5 mL离心管中,加入2倍体积的冰无水乙醇,置于-20 ℃沉淀过夜。将过夜的离心管取出,室温下12 000 r/min离心8 min,弃上清液,加75%乙醇500 μL润洗2次,将沉淀物放入60 ℃烘箱烘干,最后向DNA沉淀中加20 μL无菌双蒸水溶解,置-20 ℃保存备用。
1.4.2 ITS扩增以真菌通用引物ITS1F (5′-CTTGGTCATTTA GAGGAAGTAA-3′)和ITS4 (5′-TCCTCCGCTTATT GATATGC-3′)扩增真菌的ITS区。PCR反应体系:模板DNA (15–110 ng/μL) 1 μL,dNTPs (2.5 mmol/L) 1 μL,10×PCR缓冲液2.5 μL,ITS1F与ITS4引物(10 μmol/L)各1 μL,1.5 U Taq DNA聚合酶,ddH2O补足25 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 40 s,55 ℃ 40 s,72 ℃ 55 s,30个循环;72 ℃ 10 min。对于AMF,采用Nested PCR法扩增AMF的ITS区,即第一轮PCR采用AMF特异性引物SSUmAf (5′-TGGGTAATCTTTTGAAACTTYA-3′;5′-TGGGTAATCTTRTGAAACTTCA-3′)和LSUmAr (5′-TCGCTCTTCAACGAGGAATC-3′;5′-TATTGT TCTTCAACGAGGAATC-3′;5′-TATTGCTCTTNA ACGAGGAATC-3′),扩增区域涵盖28S大亚基(LSU) rDNA部分区域、完整的ITS区以及18S小亚基(SSU) rDNA的部分区域。这套引物可以扩增当前已知的AMF所有类群[16]。PCR反应体系:模板DNA (15–110 ng/μL) 1 μL,dNTPs (2.5 mmol/L) 1 μL,10×PCR缓冲液2.5 μL,SSUmAf与LSUmAr引物(10 μmol/L)各1 μL,1.5 U Taq DNA聚合酶,ddH2O补足25 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 40 s,60 ℃ 45 s,72 ℃ 2 min,30个循环;72 ℃ 15 min。再以本轮PCR的扩增子为模板,用真菌通用引物对TIS1F/ITS4进行第二轮扩增,得到AMF的ITS区扩增产物。最后分别将PCR产物在1.5%的琼脂糖凝胶上电泳,用凝胶成像仪观察、拍照。
1.4.3 PCR产物克隆及测序每株幼苗独立扩增10份样品,所获PCR产物混合后切胶纯化。将回收的PCR产物作为外源DNA克隆到pMD19-T载体,在16 ℃连接过夜,用E. coli DH5α感受态细胞进行转化。6株幼苗、2类引物共构建12个克隆文库,从每个真菌克隆文库中随机挑选48个呈白斑的克隆,用引物对ITS1F/ITS4扩增;PCR产物在1.5%的琼脂糖凝胶上电泳检测。电泳检测呈阳性的PCR产物委托生工生物工程(上海)股份有限公司测序。
1.5 真菌ITS序列分析在Bioedit (Version 7.2.5)中对真菌的ITS1-5.8S-ITS2全序列进行编辑,使用ClustalX (Version 2.1.0)进行比对,用Mothur (Version 1.0.0)划分真菌OTU。AMF的ITS序列一致性(Identity)≥92%的真菌被视为同一个分类单元(Operational taxonomical unit,OTU)[17-18],AMF以外的真菌ITS序列一致性≥97%视为同一OTU[19-20],在NCBI数据库中检索与OTU代表序列亲缘关系最近的真菌序列,推断真菌的分类地位。所有OTU代表序列提交NCBI,已添加至GenBank数据库中,序列号为KY965393–KY965443。
1.6 数据分析采用基于核酸与Metadata数据关联分析的FUNGuild软件[21],将2种植物的RAF划到不同的营养型(Trophic mode)和共位群(Guild)。营养型包括共生型(Symbiotroph)、腐生型(Saprotroph)、病原型(Pathotroph)以及介于2种或几种营养型的过渡型;共位群包括杜鹃花菌根真菌、外生菌根真菌、丛枝菌根真菌、兰科植物菌根真菌、内生真菌、病原真菌、腐生真菌[22]。构建真菌-植物相互作用的0, 1矩阵,对2种宿主植物与2种引物组合的克隆文库中真菌的谱系进行聚类以比较其谱系组成差异。采用NMDS的方法研究幼苗RAF群落物种组成差异(Bray-Curtis distance)与宿主植物物种、形态学指标(株高、地径、叶面积)的关系。
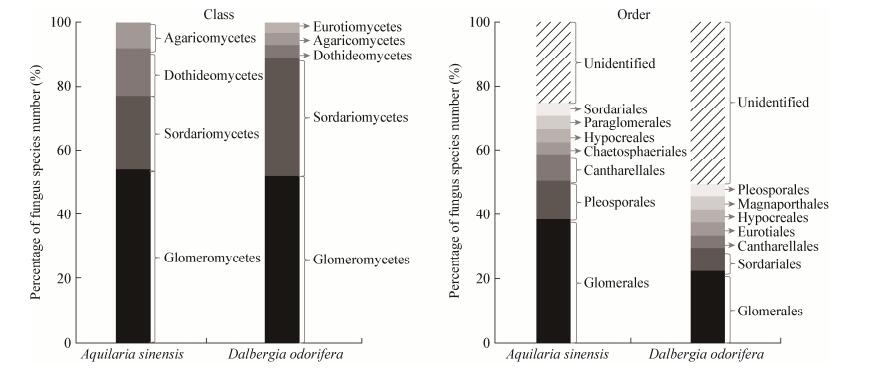
2 结果与分析 2.1 白木香与降香黄檀幼苗根部真菌的谱系与生态类型2种引物的扩增子构建的RAF克隆文库共12个,从中筛选出576个克隆用于测序。其中,真菌通用引物对应的克隆文库获得79条有效真菌序列和165条植物序列,划分为28个真菌OTU;AMF特异性引物对应的克隆文库获得256条有效真菌序列,划分为24个OTU (表 2)。从白木香幼苗检测到的RAF有4类,分别为球囊霉纲(Glomeromycetes)、粪壳菌纲(Sordariomycetes)、座囊菌纲(Dothideomycetes)和伞菌纲(Agaricomycetes)。物种最丰富的是球囊霉纲,共14种(54%);其次为粪壳菌纲6种(23%)。在目水平上,球囊霉目(Glomerales)物种最丰富,占38%;其次为格孢腔菌目(Pleosporales),占12% (图 1)。降香黄檀幼苗RAF有5类,物种数由高到低依次为球囊霉纲14种(52%)、粪壳菌纲10种(37%)、座囊菌纲1种(4%)、伞菌纲1种(4%)及散囊菌纲(Eurotiomycetes) 1种(4%);在目水平上,球囊霉目(Glomerales)的真菌物种最丰富,占22%;其次为格孢腔菌目(Pleosporales),占7% (图 1)。白木香与降香黄檀幼苗的共有真菌仅2种,Glomeromycetes sp. 2和Rhizophagus irregularis。其中,Glomeromycetes sp. 2被检测到的频度最高,在83%幼苗根部均被检测到;R. irregularis在33%幼苗根部被检测到。仅在白木香或降香黄檀幼苗根部检测到的真菌分别为24与25种(图 2)。

|
|
| 图 1 白木香与降香黄檀幼苗根部真菌类群在纲、目水平上的组成比较 Figure 1 Compositional differences of root-associated fungi grouped by Class or Order between Aquilaria sinensis and Dalbergia odorifera seedlings |
|
|
|
| 图 2 丛枝菌根真菌专性与真菌通用引物扩增的白木香与降香黄檀幼苗根部真菌谱系组成聚类图 Figure 2 Phylogenetic clustering of root-associated fungi of Aquilaria sinensis and Dalbergia odorifera seedlings revealed by AMF specific and fungal universal primer pairs 注:灰色与天蓝色分别对应相互作用的有/无(1/0). Note:Grey and sky blue represent presence and absence (1/0) of interaction between plant and fungus. |
|
|
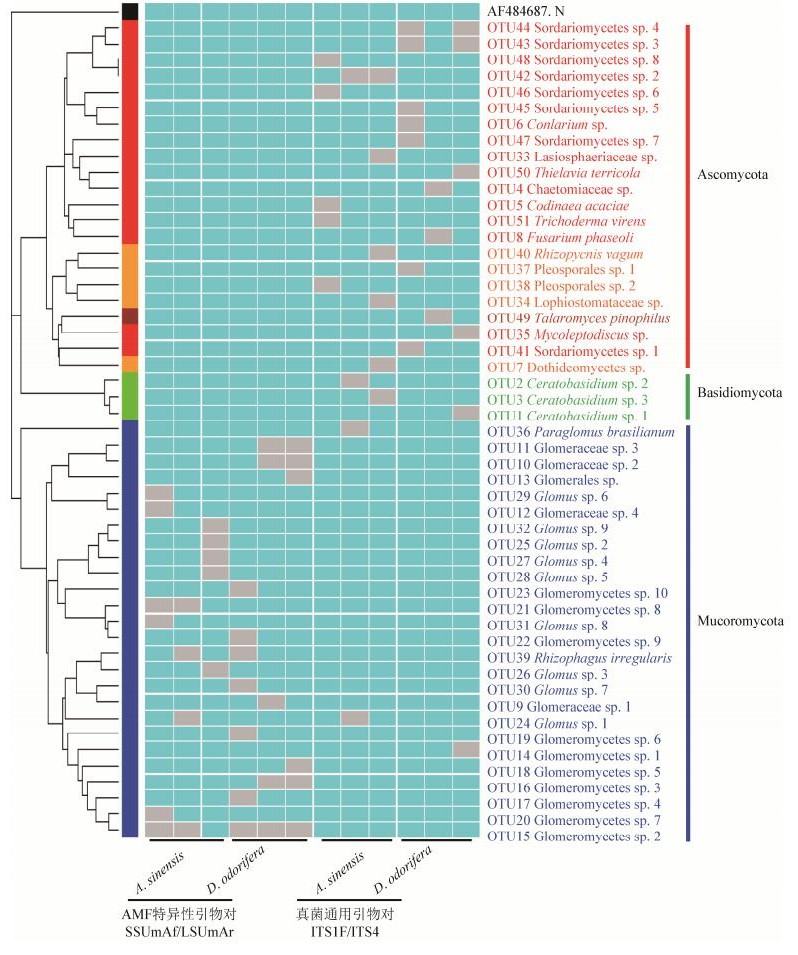
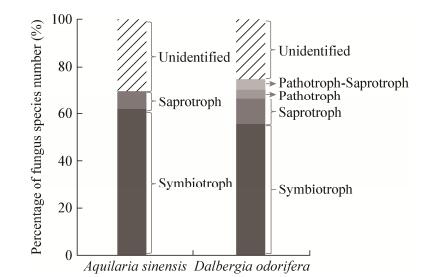
采用FUNGuild软件分析RAF的生态类型,结果表明,2种植物幼苗的RAF群落中,物种最丰富的是共生型真菌,主要包括球囊菌纲的26种真菌以及伞菌纲的3种真菌,前者是公认的AMF,后3种真菌(Ceratobasidium sp. 1、Ceratobasidium sp. 2和Ceratobasidium sp. 3)被确定为外生菌根真菌;腐生真菌是2种幼苗RAF群落中第二大类群,共5种,分别为Conlarium sp.、Rhizopycnis vagum、Talaromyces pinophilus、Thielavia terricola、Trichoderma virens;此外,还有2种真菌分别被确定为病原型真菌(Mycoleptodiscus sp.)和病原-腐生过渡型真菌(Fusarium phaseoli) (表 2)。白木香幼苗RAF至少包括2种生态类型:共生型和腐生型,各占62%和8%;降香黄檀幼苗RAF至少包括4种生态类型:共生型、腐生型、病原型、病原-腐生过渡型,各占56%、11%、4%和4% (图 3)。
|
| 图 3 白木香与降香黄檀幼苗根部真菌生态类型组成比较 Figure 3 Comparison of ecological types of root-associated fungi associated with Aquilaria sinensis and Dalbergia odorifera |
|
|
采用NMDS分析的结果表明,对于整个RAF群落而言,宿主植物的物种、叶面积、株高、地径均对真菌群落的物种组成没有明显的影响(表 3)。对于AMF群落而言,虽然宿主植物的物种、叶面积、地径对AMF群落的物种组成也没有明显的影响,但是群落的物种组成沿NMDS1与NMDS2轴的变异量中,有79%的变异量可以通过幼苗株高的差异解释,只是这一效应相对较弱(NMDS,P=0.082) (表 3,图 4)。
| 形态指标 Morphological characters |
Community by fungal universal primer | Community by AMF specific primer | |||||||
| NMDS1 | NMDS2 | R2 | P | NMDS1 | NMDS2 | R2 | P | ||
| 地径Ground diameter | -0.516 | 0.856 | 0.52 | 0.385 | -0.747 | -0.664 | 0.56 | 0.254 | |
| 株高Plant height | -0.842 | 0.539 | 0.10 | 0.857 | -0.464 | -0.885 | 0.79 | 0.082 | |
| 叶面积Leaf area | -0.653 | -0.757 | 0.42 | 0.571 | -0.095 | -0.995 | 0.39 | 0.491 | |
| 植物物种Plant identity | -0.985 | -0.171 | 0.66 | 0.613 | 0.182 | -0.983 | 0.14 | 0.805 | |

|
| 图 4 植物物种、地径、株高、叶面积对丛枝苗根真菌群落影响的非公制多维尺度分析排序图 Figure 4 Non-metric multidimensional scaling (NMDS) ordination biplot based on the occurrences of AMF, plant species, basal diameter, height and leaf area |
|
|
真菌通用引物既能检测到子囊菌门真菌(22种),又能检测到担子菌门真菌和AMF (各3种),而AMF特异性引物仅能检测到毛霉菌门真菌,但物种数多达24种,并且真菌通用引物与AMF特异性引物检测到的共有AMF仅有1种(Glomus sp. 1)相同(图 5)。
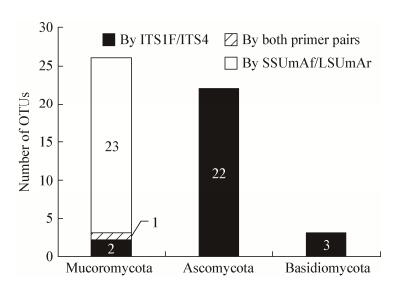
|
| 图 5 真菌通用引物与丛枝菌根真菌特异性引物检测到的各类真菌物种丰富度比较 Figure 5 Comparison of species richness of different fungal groups detected by using universal and AMF specific primer pairs |
|
|
基于克隆数的RAF物种累积曲线显示,采用真菌通用引物在单株幼苗根部检测到的真菌物种数介于3-7种,当检测到的RAF序列克隆数达到40个以上时,真菌物种数增加的趋势变平缓;而AMF特异性引物在单株幼苗根部检测到的真菌物种数介于4-7种,当检测到的RAF测序克隆数达到30个以上时,真菌物种数增加的趋势即变平缓(图 6)。
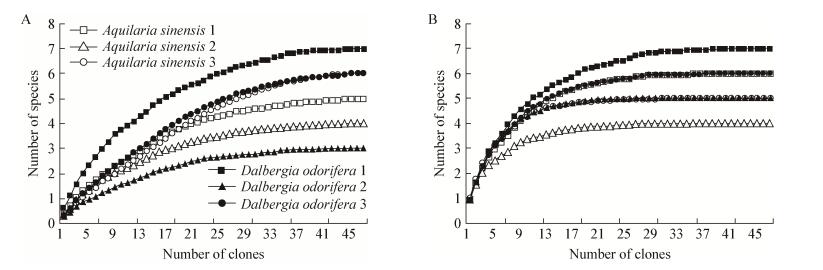
|
| 图 6 基于克隆数的单株幼苗根部真菌物种累积曲线 Figure 6 Accumulation curves of fungal species based on the number of clones of root-associated fungi of individual seedling 注:A:真菌通用引物扩增;B:丛枝菌根真菌特异性引物扩增. Note: A: Amplified by fungal universal primers; B: Amplified by AMF specific primers. |
|
|
根部真菌是影响植物幼苗存活、定植和生长的重要因子之一[23]。因此,揭示苗圃育苗的土壤介质中本土性的RAF谱系与功能多样性,对热带地区珍贵乡土树种育苗、种群恢复、资源保护与利用有重要的理论和实践意义。
3.1 苗圃白木香与降香黄檀幼苗RAF谱系和功能多样性从2种植物幼苗根部检测到的RAF谱系多样性高,包括担子菌门、子囊菌门、毛霉菌门真菌,并且这些真菌的生态类型较丰富,有腐生型也有共生型,这一结果表明,海南濒危树种保育圃育苗中普遍采用的土壤介质中存在较多的本土性真菌接种源。以AMF为代表的共生型真菌是白木香与降香黄檀幼苗RAF群落中物种最丰富的类群,
如Glomus sp. 1和Rhizophagus irregularis等,这类菌根真菌可能促进这2种植物幼苗的养分吸收与环境适应性[3]。以子囊菌门为主的腐生型真菌在白木香与降香黄檀幼苗RAF群落中也较常见,如Trichoderma virens与Talaromyces pinophilus。这2种真菌分别在瑞典森林苗圃与埃及农田中被检测到,并且前者对病害可能有拮抗作用[5],后者能产生β-葡萄糖苷水解酶促进有机质分解[24]。本研究中,83%以上的幼苗根部同时检测到共生型与腐生型真菌。有研究表明人工接种腐生真菌有效地促进了Pinus radiata幼苗生长,而且同时接种腐生真菌与菌根真菌效果更好[25]。因此,本研究中同株幼苗根部2种生态类型的真菌共存可能对幼苗养分吸收有积极的作用。本研究从降香黄檀幼苗根系检测到少量的病原型真菌,如子囊菌门的Mycoleptodiscus sp.和Fusarium phaseoli。虽然在热带森林中病原真菌对植物群落的密度制约是维持热带森林生态系统物种多样性的重要机制之一[26],但是对于苗圃育苗而言,病原型的RAF具有致幼苗生长不良或死亡的风险,并且会影响树苗的移栽成活率[6]。热带地区雨季高温高湿,容易导致病害盛行,因此有必要在幼苗移栽前对根部病原真菌进行监测。
当土壤中真菌繁殖体不足时,人工接种根部真菌成为培育健康优质苗木的技术措施之一[13, 27],然而人工接种的真菌会随着时间迁移逐渐被本土性真菌取代,如将担子菌门真菌接种到幼苗,后期生长中检测到的真菌却以子囊菌为主[14]。因此,苗圃土壤介质中本土性真菌繁殖体才是幼苗的可持续侵染源[3]。在幼苗出圃前对其根部真菌状况进行全面监测,将有助于形成完善的育苗技术体系,并采取合理的育苗管理措施。
3.2 宿主植物对RAF群落物种组成的影响有较多的研究表明,宿主植物物种与环境因子共同驱动RAF群落的物种组成[7-8],但是本研究NMDS分析的结果表明,宿主植物物种、株高、地径、叶面积对RAF群落物种组成的影响不显著,真菌群落物种组成的变异(Variance)在宿主种间与种内均较大。这一结果可能是由于本苗圃条件下,真菌繁殖体分布的空间异质性造成。值得注意的是,当仅考虑AMF群落时,宿主植物幼苗的株高对AMF群落的物种组成有轻度影响。可能是由于AMF为专性共生,对宿主植物的依赖相对于其它类群的真菌更大[8]。真菌群落的物种组成是一系列环境因子驱动的结果,气候[22]、土壤有机质和养分含量[28-29]均会影响真菌群落的组配。因此,幼苗移栽后,在新环境因子的驱动下RAF群落将如何变化,值得进一步研究。
3.3 引物和测序深度对揭示RAF群落多样性的影响自然生态系统中,AMF与90%以上的有花植物共生,采用AMF专性引物的研究发现,仅单株植物根系上就能检测到20种以上的AMF[12, 30],然而使用真菌通用引物检测植物RAF群落组成时,AMF物种数所占比例极低[10-11]。本研究通过对AMF专性引物与真菌通用引物的比较研究证实,真菌通用引物虽然能更全面地揭示根部真菌的谱系组成,却可能低估RAF群落中的AMF多样性。这一结果的可能原因是:(1) AMF的DNA模板与真菌通用引物序列的匹配性差。然而,本研究中以AMF特异性引物的第一轮扩增子为模板,真菌通用引物扩增到的AMF种类极为丰富。因此,模板与引物的匹配性不是导致这一现象的原因。(2)特定真菌的基因组DNA在总DNA中的相对浓度,决定其竞争性地与引物结合的能力[15]。从根系提取的总DNA中,AMF基因组DNA相对浓度较低,即使采用AMF专性引物,第一轮的扩增子量不足以在电泳中被检测到[16]。本研究采用Nested PCR法,将AMF特异性引物与通用引物相结合进行扩增,获得了多样性的AMF、担子菌和子囊菌。因此,根部真菌研究中,若同时采用真菌通用引物与AMF特异性引物扩增RAF,能更全面地揭示RAF群落物种组成。
克隆文库中,被测序的克隆数是影响真菌群落物种组成能否被完全反映的重要因素之一[31]。本研究中,根部真菌的物种累积曲线显示,检测48个真菌克隆即能较全面地反映2种植物的单株幼苗RAF克隆文库的物种组成。
| [1] |
Vandenkoornhuyse P, Baldauf SL, Leyval C, et al. Extensive fungal diversity in plant roots[J]. Science, 2002, 295(5562): 2051. DOI:10.1126/science.295.5562.2051 |
| [2] |
Spatafora JW, Chang Y, Benny GL, et al. A phylum-level phylogenetic classification of zygomycete fungi based on genome-scale data[J]. Mycologia, 2016, 108(5): 1028-1046. DOI:10.3852/16-042 |
| [3] |
Smith SE, Read DJ. Mycorrhizal Symbiosis[M]. 3rd ed. London: Academic Press, 2008, 558-581.
|
| [4] |
Stone JK, Bacon CW, White JF. An overview of endophytic microbes: endophytism defined[A]//Bacon CW, White Jr JF. Microbial Endophytes[M]. New York: Marcel Dekker, 2000: 29-33
|
| [5] |
Hohmann P, Jones EE, Hill RA, et al. Understanding Trichoderma in the root system of Pinus radiata: associations between rhizosphere colonisation and growth promotion for commercially grown seedlings[J]. Fungal Biology, 2011, 115(8): 759-767. DOI:10.1016/j.funbio.2011.05.010 |
| [6] |
Filion M, Hamelin RC, Bernier L, et al. Molecular profiling of rhizosphere microbial communities associated with healthy and diseased black spruce (Picea mariana) seedlings grown in a nursery[J]. Applied and Environmental Microbiology, 2004, 70(6): 3541-3551. DOI:10.1128/AEM.70.6.3541-3551.2004 |
| [7] |
Fujimura KE, Egger KN. Host plant and environment influence community assembly of high Arctic root-associated fungal communities[J]. Fungal Ecology, 2012, 5(4): 409-418. DOI:10.1016/j.funeco.2011.12.010 |
| [8] |
Martínez-García LB, Richardson SJ, Tylianakis JM, et al. Host identity is a dominant driver of mycorrhizal fungal community composition during ecosystem development[J]. New Phytologist, 2015, 205(4): 1565-1576. DOI:10.1111/nph.13226 |
| [9] |
Schoch CL, Seifert KA, Huhndorf S, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for fungi[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(16): 6241-6246. DOI:10.1073/pnas.1117018109 |
| [10] |
Menkis A, Ihrmark K, Stenlid J, et al. Root-associated fungi of Rosa rugosa grown on the frontal dunes of the Baltic Sea Coast in Lithuania[J]. Microbial Ecology, 2014, 67(4): 769-774. DOI:10.1007/s00248-013-0351-8 |
| [11] |
Toju H, Sato H, Yamamoto S, et al. How are plant and fungal communities linked to each other in belowground ecosystems? A massively parallel pyrosequencing analysis of the association specificity of root-associated fungi and their host plants[J]. Ecology and Evolution, 2013, 3(9): 3112-3124. DOI:10.1002/ece3.706 |
| [12] |
Herrmann L, Lesueur D, Bräu L, et al. Diversity of root-associated arbuscular mycorrhizal fungal communities in a rubber tree plantation chronosequence in Northeast Thailand[J]. Mycorrhiza, 2016, 26(8): 863-877. DOI:10.1007/s00572-016-0720-5 |
| [13] |
He YJ, Zhong ZC, Liu JM, et al. Growth response of Broussonetia papyrifera seedlings to VA mycorrhizal fungi inoculation[J]. Chinese Journal of Applied Ecology, 2007, 18(10): 2209-2213. 何跃军, 钟章成, 刘济明, 等. 构树幼苗对接种丛枝菌根真菌的生长响应[J]. 应用生态学报, 2007, 18(10): 2209-2213. |
| [14] |
Muthukumar T, Udaiyan K. Growth of nursery-grown bamboo inoculated with arbuscular mycorrhizal fungi and plant growth promoting rhizobacteria in two tropical soil typeswith and without fertilizer application[J]. New Forests, 2006, 31(3): 469-485. DOI:10.1007/s11056-005-1380-z |
| [15] |
Huang CW, Liao YH, Ding Q. Two sample pooling strategies revealed different root-associated fungal diversity of Rhododendron species[J]. Acta Microbiologica Sinica, 2017, 57(4): 571-581. 黄彩微, 廖映辉, 丁琼. 两种混合样品策略对揭示杜鹃花根部真菌多样性的影响[J]. 微生物学报, 2017, 57(4): 571-581. |
| [16] |
Krüger M, Stockinger H, Krüger C, et al. DNA-based species level detection of Glomeromycota: one PCR primer set for all arbuscular mycorrhizal fungi[J]. New Phytologist, 2009, 183(1): 212-223. DOI:10.1111/nph.2009.183.issue-1 |
| [17] |
Stockinger H, Krüger M, Schüßler A. DNA barcoding of arbuscular mycorrhizal fungi[J]. New Phytologist, 2010, 187(2): 461-474. DOI:10.1111/j.1469-8137.2010.03262.x |
| [18] |
Börstler B, Renker C, Kahmen A, et al. Species composition of arbuscular mycorrhizal fungi in two mountain meadows with differing management types and levels of plant biodiversity[J]. Biology and Fertility of Soils, 2006, 42(4): 286-298. DOI:10.1007/s00374-005-0026-9 |
| [19] |
O'Brien HE, Parrent JL, Jackson JA, et al. Fungal community analysis by large-scale sequencing of environmental samples[J]. Applied and Environmental Microbiology, 2005, 71(9): 5544-5550. DOI:10.1128/AEM.71.9.5544-5550.2005 |
| [20] |
Smith ME, Douhan GW, Rizzo DM. Intra-specific and intra-sporocarp ITS variation of ectomycorrhizal fungi as assessed by rDNA sequencing of sporocarps and pooled ectomycorrhizal roots from a Quercus woodland[J]. Mycorrhiza, 2007, 18(1): 15-22. DOI:10.1007/s00572-007-0148-z |
| [21] |
Nguyen NH, Song ZW, Bates ST, et al. FUNGuild: an open annotation tool for parsing fungal community datasets by ecological guild[J]. Fungal Ecology, 2016, 20: 241-248. DOI:10.1016/j.funeco.2015.06.006 |
| [22] |
Tedersoo L, Bahram M, Põlme S, et al. Global diversity and geography of soil fungi[J]. Science, 2014, 346(6213): 1256688. DOI:10.1126/science.1256688 |
| [23] |
Devine WD, Harrington CA, Leonard LP. Post-planting treatments increase growth of Oregon white oak (Quercus garryana Dougl. ex Hook.) seedlings[J]. Restoration Ecology, 2007, 15(2): 212-222. DOI:10.1111/rec.2007.15.issue-2 |
| [24] |
El-Naggar NEA, Haroun SA, Oweis EA, et al. Identification of newly isolated Talaromyces pinophilus and statistical optimization of β-Glucosidase production under solid-state fermentation[J]. Preparative Biochemistry and Biotechnology, 2015, 45(7): 712-729. DOI:10.1080/10826068.2014.943375 |
| [25] |
Chávez D, Pereira G, Machuca Á. Stimulation of Pinus radiata seedling growth using ectomycorrhizal and saprophytic fungi as biofertilizers[J]. Bosque, 2014, 35(1): 57-63. |
| [26] |
van der Putten WH. Pathogen-driven forest diversity[J]. Nature, 2000, 404(6775): 232-233. DOI:10.1038/35005188 |
| [27] |
Southworth D, Carrington EM, Frank JL, et al. Mycorrhizas on nursery and field seedlings of Quercus garryana[J]. Mycorrhiza, 2009, 19(3): 149-158. DOI:10.1007/s00572-008-0222-1 |
| [28] |
Aguilar R, Carreón-Abud Y, López-Carmona D, et al. Organic fertilizers alter the composition of pathogens and arbuscular mycorrhizal fungi in maize roots[J]. Journal of Phytopathology, 2017, 165(7/8): 448-454. |
| [29] |
Grant C, Bittman S, Montreal M, et al. Soil and fertilizer phosphorus: effects on plant P supply and mycorrhizal development[J]. Canadian Journal of Plant Science, 2005, 85(1): 3-14. DOI:10.4141/P03-182 |
| [30] |
Li LF, Li T, Zhang Y, et al. Molecular diversity of arbuscular mycorrhizal fungi and their distribution patterns related to host-plants and habitats in a hot and arid ecosystem, southwest China[J]. FEMS Microbiology Ecology, 2010, 71(3): 418-427. DOI:10.1111/fem.2010.71.issue-3 |
| [31] |
Taylor DL, Herriott IC, Long J, et al. TOPO TA is A-OK: a test of phylogenetic bias in fungal environmental clone library construction[J]. Environmental Microbiology, 2007, 9(5): 1329-1334. DOI:10.1111/emi.2007.9.issue-5 |
 2018, Vol. 45
2018, Vol. 45