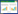扩展功能
文章信息
- 姚义勇, 江莉莎, 张莉, 郭述良, 柳岩
- YAO Yi-Yong, JIANG Li-Sha, ZHANG Li, GUO Shu-Liang, LIU Yan
- 分枝杆菌噬菌体Guo1环状基因组特征及生物学特性分析
- Circular genetic information and biologic characteristics of mycobacteriophage Guo1
- 微生物学通报, 2015, 42(8): 1529-1538
- Microbiology China, 2015, 42(8): 1529-1538
- 10.13344/j.microbiol.china.140872
-
文章历史
- 收稿日期: 2014-11-04
- 接受日期: 2015-02-05
- 优先数字出版日期(www.cnki.net): 2015-03-04
2012年WHO公布的疫情显示:2011年全球新增结核病人900万,值得注意的是印度和中国的耐药结核占到全世界的60%[1],我国也是全球第二大结核病高负担国家,发病人数一直位于甲、乙类传染病前列[2]。结核病治疗面临着易复发和病程迁延两大难题,临床上常采用长疗程化疗来彻底清除结核菌,实际上在化疗初期2−3周就清除了90%以上的结核菌,其余8−9个月的疗程主要是清除潜伏感染的结核菌,长疗程显著增加了病人的经济负担和药物不良反应,导致病人治疗依从性显著下降,不规则用药和不足疗程用药增多。目前认为潜伏感染是由持留结核菌导致[3],结核菌在面临不利条件(药物、免疫、缺氧等)会发生代谢降低、胞壁增厚等变化,进入持留状态对结核药产生耐受。
在藤黄微球菌中鉴定出的复苏因子(Resuscitationpromoting factor,Rpf)能够使持留状态的结核菌恢复正常生长[4],重新对药物产生敏感性,一般认为Rpf主要通过水解持留结核菌胞壁的肽聚糖来解除休眠菌增厚的细胞壁或通过水解细胞壁肽聚糖启动一系列信号通路来恢复生长[5,6]。分枝杆菌噬菌体具有能够裂解结核分枝杆菌的特性,使其成为新型抗结核病药物的研究热点,近年来关于利用噬菌体诊断和治疗结核病的各项研究已经取得了重大进展[7,8,9],在对分枝杆菌噬菌体TM4基因组研究中发现含有Motif3基序的噬菌体具有Rpf样水解肽聚糖的作用,且可以更容易的将DNA注入休眠宿主菌[10,11],本课题组通过体外实验确证了噬菌体TM4能够复苏休眠菌[12]。在完成了分枝杆菌噬菌体Guo1基因组测序和生物信息学初步分析之后,发现其具有与噬菌体TM4相同的Motif3基序,具有复苏持留结核菌的潜力,本研究初步探索噬菌体Guo1基因组学特征和生物学特性,为下一步开发利用Guo1奠定基础。
1 材料与方法 1.1 材料 1.1.1 宿主菌及噬菌体来源:宿主菌:耻垢分枝杆菌(CMCC93202)来自中国药品生物制品检定所;分枝杆菌噬菌体Guo1为本课题组自主分离鉴定株。 1.1.2 主要试剂及仪器:λ噬菌体DNA提取试剂盒,艾比根生物技术有限公司;限制性内切酶Bgl II,Fermentas公司;DNaseⅠ、RNase A,Sigma公司;双层琼脂固体培养基,底层7H10培养基,上层7H9固体培养基(含0.4%琼脂),BD公司;噬菌体缓冲液(Phage buffer)用于稀释噬菌体,配方:Tris-base 1.211 g,MgSO4 1.204 g,NaCl 4 g,ddH2O 980 mL,pH 7.5。CO2恒温培养箱,Thermo Forma公司;低温超速离心机,Sigma公司;数字图像分析系统,Bio-Rad公司;pH计,Mettler Toledo公司。
1.2 方法 1.2.1 纯化噬菌体颗粒:采用双层琼脂平板法扩增噬菌体,24 h后刮取上层培养基,4°C、10 000×g离心10 min,收集上清用0.22 μm滤器过滤除菌,加入DNaseⅠ(终浓度40 mg/L)和RNase A (终浓度10 mg/L),37°C孵育30 min,加入NaCl(终浓度58.5 g/L),混匀后冰浴1 h,10 000×g离心10 min后收集上清液,在上清液中加入10%聚乙二醇8000,冰浴1 h,11 000×g离心10 min,弃上清,倒置离心管,待管底白色沉淀干燥后,加入1 mL Phage buffer缓冲液反复吹打混匀,吹打动作要轻柔,避免破坏噬菌体尾端,4°C静置过夜。次日10 000×g离心10 min收集上清液,用0.22 μm滤器过滤除菌后即得噬菌体粗制颗粒。 1.2.2 提取分枝杆菌噬菌体Guo1核酸:用λ噬菌体DNA提取试剂盒提取Guo1核酸,具体步骤如下:取纯化的噬菌体颗粒10 mL,加入2 mL冰预冷的噬菌体沉淀液,混匀后冰浴1 h,10 000×g、4°C离心10 min,弃上清,干燥后收集白色沉淀;加入500 μL裂解缓冲液,吹打重悬;加入100 μL 20% SDS,70°C温育10 min后置冰上冷却;加入100 μL杂质沉淀液,混匀后13 000×g、4°C离心10 min,收集上清液,加入350 μL结合液LB,将混合液加入吸附柱中12 000 r/min离心30 s,倒掉废液;向吸附柱中加入700 μL漂洗液,12 000 r/min离心1 min,弃废液;将吸附柱放回空收集管中,13 000 r/min离心2 min,除去漂洗液;离心后将吸附柱放入1.5 mL离心管中,加100 μL预热至50°C的洗脱缓冲液,室温放置1 min后超速离心机12 000 r/min离心1 min,收集离心管中的液体即得噬菌体基因组DNA,用紫外分光光度计检测核酸的浓度(A260)和纯度(A260/A280),并进行琼脂糖凝胶电泳和脉冲场电泳检测DNA质量。 1.2.3 Guo1全基因组序列测定:将提取的Guo1核酸送北京六合华大基因科技股份有限公司(北京,中国)按照鸟枪法策略进行测序,经Phred/PhraD/Consedh软件组装重叠群和PCR扩增连接重叠群缺口,完成全基因组测序,上传序列信息至GenBank,登录号为KJ725374。 1.2.4 噬菌体基因组一般特性及编码基因预测分析:使用DNAStar软件包中EditSeq软件分析基因组大小、基因组碱基组成、G+C含量;采用华盛顿大学LOW LAB实验室的tRNAscan-SE软件预测基因组上的tRNA基因;运用Promoter predictions软件预测可能的启动子区;通过TRF软件预测DNA序列中的串联重复序列;使用美国国立卫生研究院(National Institutes of Health,NIH)网站上Glimmer软件预测噬菌体Guo1基因组的编码序列,进一步对潜在编码序列进行同源性对比及基因功能注释。 1.2.5 系统进化树构建:使用BLAST软件下的核酸序列比对(Nucleotide blast)进行全基因组序列比对,设置参数为:Tree Method选项“Neighbor-Joining”,Max Seq Difference选项“0.05”,Database选项“nr”,根据相似性比对结果构建噬菌体系统进化树[13],初步判断确定噬菌体分群和生物进化关系。 1.3 噬菌体的生物学特性 1.3.1 最佳感染复数的测定:感染复数(Multiplicity of infection,MOI)是指初始感染时加入的噬菌体数量与宿主菌数量的比值。参照Lu的方法[14],采用梯度稀释和双层琼脂培养法测定噬菌体滴度。预实验分别测定宿主菌和噬菌体的浓度,按照MOI分别为10−3、10−2、10−1、1、101、102的比例加入噬菌体和宿主菌,各个MOI均采用双份复板培养取平均值,同时设置不加噬菌体的宿主菌和不加宿主菌的噬菌体为对照组,37°C双层琼脂培养24 h后收集噬菌体,采用梯度稀释测定噬菌体滴度,能产生最高滴度噬菌体的MOI即为最佳感染复数。 1.3.2 一步生长曲线的绘制:参照黄建军等[15]高感染复数测定噬菌体一步生长曲线的方法,略有修改。按照MOI=10的比例加入噬菌体和宿主菌,37°C温育15 min,13 000×g离心1 min,弃上清,7H9液体培养基洗涤2次后,重悬于5 mL的7H9液体培养基,迅速置于37°C摇床培养(160 r/min),300 min,每30 min取样100 μL,13 000×g离心1 min,吸取上清,采用梯度稀释和双层琼脂培养法测定噬菌体滴度,各时间点做双份复管培养取平均值,实验重复3次。以感染时间为横坐标,实际测得的噬菌体滴度(×104 PFU/mL)为纵坐标,绘制一步生长曲线,得出裂解量、潜伏期和裂解期。由于采用的是高感染复数的方式,可以认为整个反应体系的宿主菌均被噬菌体感染,初始的宿主菌浓度即为初始感染的细菌数。根据:裂解量=裂解末期噬菌体数/初期感染噬菌体的宿主菌数,计算出噬菌体的裂解量。 1.3.3 噬菌体对紫外线照射的抵抗力:取噬菌体原液3 mL于直径6 cm的培养皿中,置于紫外灯(18 w,36 cm)下照射90 min,每15 min取样100 µL,暗处室温放置30 min,测定其效价,实验重复3次。存活率以0 min时取样为100%,计算出存活率。 1.3.4 噬菌体对氯仿的敏感性:750 µL噬菌体原液中加入等体积氯仿混匀,37°C静置10 min,4 000×g离心15 min,吸取上清,测定其效价,设置加入等体积ddH2O为对照组,实验重复3次,存活率以加入等体积ddH2O为100%,计算存活率。 1.3.5 噬菌体对酒精的抵抗力:320 µL噬菌体原液中加入95%酒精1 180 µL混匀(酒精终浓度为75%),37°C静置10 min,4 000×g离心15 min,吸取下层水相,测定其效价,设置加入同样体积ddH2O为对照组,实验重复3次,存活率以加入等体积ddH2O为100%,计算存活率。 1.3.6 温度对噬菌体的影响:取等量噬菌体原液于不同温度(25、37、50、60°C)分别水浴30 min及60 min,取出后迅速冰浴冷却,测定噬菌体滴度,以处理温度为横坐标,实际测得的噬菌体滴度(×105 PFU/mL)为纵坐标。 1.3.7 pH对噬菌体的影响:将噬菌体加入不同pH (3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0、12.0)的7H9液体培养基中,37°C水浴1 h,测定不同pH液体培养基中噬菌体的滴度。 2 结果与分析 2.1 基因组一般特性Guo1具有双链环状DNA基因组,长度为40 086 nt,碱基组成:A=5 789,C=13 375,G=13 420,T=7 502,G+C含量为66.84%,这与分枝杆菌噬菌体G群的G+C含量很相近;tRNAscan-SE软件未找到tRNA。
2.2 启动子序列和串联重复序列预测结果考虑到启动子位于结构基因的上游以及基因的分布,对Promoter predictions软件预测出的具有启动子结构特征的区域中,推测出Guo1基因组可能的7个启动子区,见表1;TRF软件预测出3个串联重复序列,见表2。
| 起始位点 Start site |
终止位点 End site |
评分 Score |
启动子区 Promoter sequence |
| 1 954 | 1 999 | 0.95 | AACGGGTTGAGTCCGAGCTCGGATCCGTAGATCATCGCCGCGGCAGCCTC |
| 5 212 | 5 257 | 0.83 | GTCTTGAGCAGAATCGGTTTCCAGCCGGCGATGATGGTCGCGGCGATGTT |
| 15 331 | 15 376 | 0.82 | TCGGGTTGTCGAGCATCTGGTCGATGAGATAGATCGCTTCCTGCGCCGGC |
| 17 020 | 17 065 | 0.90 | GAATGACTTGTGGGTGTGGACGGTGCCGATGTAGTTCATCCGCTTGTCCC |
| 24 014 | 24 059 | 0.91 | TCGGTTTGAGTGCGACCGTCCAGAAGACGGAAATTGCTTGGCCGAACAGT |
| 24 726 | 24 771 | 0.86 | GCCAGGTTCGAAGTCGAACGGGCCCAGGTTGTAATCCCTCACGGGCCCGT |
| 38 359 | 38 404 | 0.82 | GAGGTCAGTGACGGGTCGATTCCGACGGCGATCATGCGCGGTGTCCCTTG |
Note: The −35 region and −10 region shown in underlined type; The transcription start shown in bold type.
| 起始位点 Start site | 终止位点 End site | 长度 Length (nt) | 重复序列 Tandem repeat sequences | 评分 Score |
| 20 791 | 20 831 | 21 | GGTTCATCGCCGCCGCCTGCT | 55 |
| 30 456 | 30 484 | 15 | CCAGCGTCGGCCGGC | 51 |
| 34 509 | 34 564 | 20 | CCGCAGTTCGGGTGCTGCTA | 50 |
Glimmer软件预测出59个编码基因(表3),共有3种不同的起始密码子,其中51个ATG,7个GTG和1个TTG;Guo1基因组的推定基因中,最长的编码基因是基因28 (4 014 nt),编码卷尺蛋白,最小的编码基因是基因4 (153 nt),编码的蛋白质功能不明,除了5个编码基因位于正链外,其余的编码基因都位于负链;结构基因:基因41和42编码入门蛋白,基因21、23、24、25、26和27编码尾部蛋白,基因28编码卷尺蛋白,基因37编码衣壳蛋白;裂解酶系统:基因16和基因17分别编码裂解酶B和裂解酶A;整合酶系统:基因12编码整合酶,基因10编码阻遏酶;基因43编码末端酶;其中11个推定基因未匹配到相应的基因功能。
| 基因编号 Number |
起始位点 Start site |
终止位点 end site |
基因长度 length |
核酸链 strand |
起始密码子 Start codon |
NCBI注释 NCBI annotation |
功能预测 Putative function |
| 1 | 277 | 810 | 534 | − | ATG | Leo 0043 | Hypothetical protein |
| 2 | 813 | 2 216 | 1 404 | − | ATG | Leo 0042 | Hypothetical protein |
| 3 | 2 234 | 3 319 | 1 086 | − | ATG | Leo 0041 | PDDEXK-like domain |
| 4 | 3 316 | 3 468 | 153 | − | ATG | Leo 0040 | Hypothetical protein |
| 5 | 3 452 | 3 802 | 351 | − | ATG | Leo 0039 | Hypothetical protein |
| 6 | 3 802 | 3 951 | 150 | − | ATG | Halo gp39 | − |
| 7 | 3 948 | 4 298 | 351 | − | ATG | Leo | ASC-1-like protein |
| 8 | 4 352 | 4 468 | 117 | + | GTG | Halo gp37 | Hypothetical protein |
| 9 | 4 477 | 5 013 | 537 | − | ATG | Leo 0035 | Hypothetical protein |
| 10 | 5 076 | 5 474 | 399 | − | ATG | Sedge | repressor |
| 11 | 5 471 | 5 716 | 246 | − | ATG | Angel gp34 | Helix-turn-helix domain |
| 12 | 5 747 | 6 388 | 642 | + | TTG | Leo | Integrase |
| 13 | 6 385 | 7 002 | 618 | − | GTG | Legendre 0028 | Hypothetical protein |
| 14 | 6 999 | 7 244 | 246 | − | GTG | DNAIII 0030 | Hypothetical protein |
| 15 | 7 324 | 7 674 | 351 | − | ATG | Halo gp29 | − |
| 16 | 7 700 | 8 908 | 1 209 | − | ATG | Halo gp28 | Lysin B |
| 17 | 8 908 | 10 224 | 1 317 | − | ATG | Halo gp27 | Lysin A |
| 18 | 10 285 | 10 479 | 195 | − | ATG | Halo gp26 | − |
| 19 | 10 479 | 10 754 | 276 | − | ATG | Halo gp2 | − |
| 20 | 10 754 | 10 936 | 183 | − | ATG | Halo gp23 | − |
| 21 | 10 939 | 11 259 | 321 | − | ATG | Leo | Minor tail protein |
| 22 | 11 274 | 13 409 | 2 136 | − | ATG | BPs 121 | − |
| 23 | 13 409 | 13 843 | 435 | − | ATG | Leo | Minor tail protein |
| 24 | 13 854 | 14 990 | 1 137 | − | ATG | Leo | Minor tail protein |
| 25 | 14 987 | 15 448 | 462 | − | ATG | Halo | Minor tail protein |
| 26 | 15 448 | 17 190 | 1 743 | − | ATG | Halo | Minor tail protein |
| 27 | 17 202 | 18 374 | 1 173 | − | ATG | Halo | Minor tail protein |
| 28 | 18 374 | 22 387 | 4 014 | − | ATG | Halo | Tape measure protein |
| 29 | 22 387 | 22 764 | 378 | − | GTG | Leo 0015 | Hypothetical protein |
| 30 | 22 773 | 23 282 | 510 | − | ATG | Bps | Potenial virion protein |
| 31 | 23 383 | 23 997 | 615 | − | ATG | Bps | Major tail subunit |
| 32 | 23 994 | 24 422 | 429 | − | ATG | BPs 112 | Hypothetical protein |
| 33 | 24 434 | 24 697 | 264 | − | ATG | BPs 111 | Hypothetical protein |
| 34 | 24 684 | 25 040 | 357 | − | ATG | BPs 110 | Hypothetical protein |
| 35 | 25 040 | 25 537 | 498 | − | ATG | BPs 19 | Hypothetical protein |
| 36 | 25 549 | 25 776 | 228 | − | ATG | BPs 18 | Hypothetical protein |
| 37 | 25 813 | 26 748 | 936 | − | ATG | Chy2 006 | Capsid protein |
| 38 | 26 794 | 27 336 | 543 | − | ATG | BPs 16 | Hypothetical protein |
| 39 | 27 455 | 27 661 | 207 | − | ATG | Halo gp5 | Hypothetical protein |
| 40 | 27 658 | 30 447 | 2 790 | − | ATG | BPs 14 | Major surface protein1B |
| 41 | 30 448 | 30 696 | 249 | − | ATG | Angel gp3 | Portal protein |
| 42 | 30 797 | 31 963 | 1 167 | − | GTG | Sedge 003 | Portal protein |
| 43 | 31 960 | 33 396 | 1 437 | − | ATG | Leo | Terminase |
| 44 | 33 464 | 33 805 | 342 | − | ATG | Halo gp1 | − |
| 45 | 33 851 | 34 672 | 822 | + | ATG | Chy2 0058 | Hypothetical protein |
| 46 | 34 651 | 34 893 | 243 | − | ATG | Halo gp63 | − |
| 47 | 34 992 | 35 327 | 336 | − | ATG | Leo 0056 | Pfam05305 |
| 48 | 35 359 | 35 541 | 183 | − | ATG | Halo gp61 | − |
| 49 | 35 541 | 35 756 | 216 | − | ATG | DNAIII 0054 | Hypothetical protein |
| 50 | 35 802 | 36 290 | 489 | + | GTG | DNAIII 0055 | Hypothetical protein |
| 51 | 36 335 | 36 571 | 237 | − | ATG | Leo 0053 | Hypothetical protein |
| 52 | 36 676 | 37 101 | 426 | − | ATG | Legendre 006 | Hypothetical protein |
| 53 | 37 101 | 37 259 | 159 | − | ATG | Halo gp55 | − |
| 54 | 37 316 | 37 591 | 276 | + | GTG | Legendre 007 | Hypothetical protein |
| 55 | 37 803 | 38 393 | 591 | − | ATG | Leo | RuvC |
| 56 | 38 390 | 38 830 | 441 | − | ATG | Halo gp51 | − |
| 57 | 38 827 | 39 171 | 345 | − | ATG | Leo 0047 | Hypothetical protein |
| 58 | 39 168 | 39 488 | 321 | − | ATG | Leo 0046 | Hypothetical protein |
| 59 | 39 687 | 40 058 | 372 | − | ATG | Leo 0044 | Hypothetical protein |
Guo1全基因组序列BLAST比对发现:Guo1与分枝杆菌噬菌体G群具有明显的同源性,基因组序列覆盖率大于95%,一致性大于98%,E值为0的噬菌体有14株,对14株相似性高的噬菌体构建系统进化树分析(图1),Guo1亲缘较近的是Leo、BO4和DNAⅢ,这3株噬菌体均由本课题组完成测序和基因初步分析,已证实为G群噬菌体[16,17,18],且噬菌体Liefie、Angel、Halo、Hope、BPs、Avrafan均为G群噬菌体,表明噬菌体Guo1应该归为分枝杆菌噬菌体G群。
|
|
图 1
噬菌体Guo1的系统进化树
Figure 1
Phylogenetic tree of mycobacteriophage Guo1
注:进化树由BLAST比对产生(Tree method:Neighbor-Joining,Max Seq Difference:0.05,Database:nr);括号内代表GenBank中序列登录号;标尺代表进化距离. Note: phylogenetic tree was constructed by BLAST (Tree method: Neighbor-Joining,Max Seq Difference: 0.05,Database: nr); the numbers in the bracket denote GenBank accession number,the scale plate denote evolutionary distance. |
| 感染复数 MOI |
噬菌体浓度 Number of bacterium phage (PFU/mL) |
细菌浓度 Number of bacteria (CFU/mL) |
24 h滴度 Titer at 24 h (PFU/mL) |
| 10−3 | 2×109 | 1×107 | 2.9×107 |
| 10−2 | 2×108 | 1×107 | 6.4×107 |
| 10−1 | 2×107 | 1×107 | 2.1×107 |
| 100 | 2×106 | 1×107 | 0.7×107 |
| 101 | 2×105 | 1×107 | 3.9×107 |
| 102 | 2×104 | 1×107 | 5.3×107 |
|
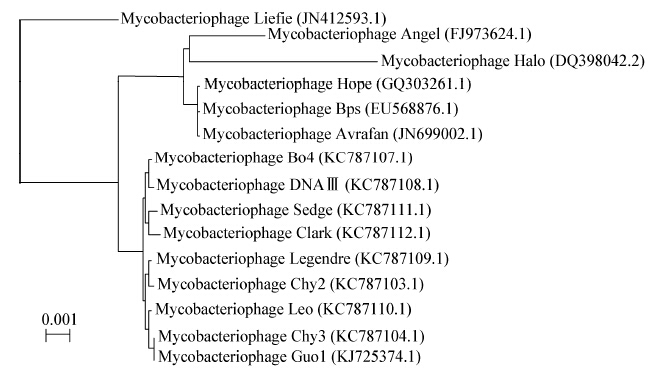
| 图 2 噬菌体Guo1一步生长曲线 Figure 2 One step growth curve of Guo1 |
|
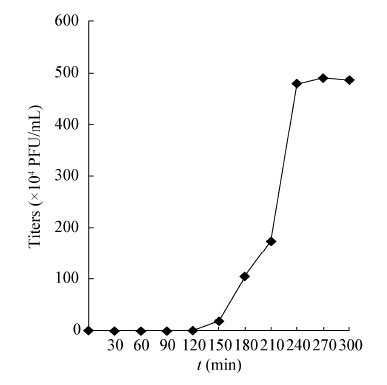
| 图 3 噬菌体Guo1对紫外线的耐受性 Figure 3 Ultraviolet resistibility of Guo1 |
|
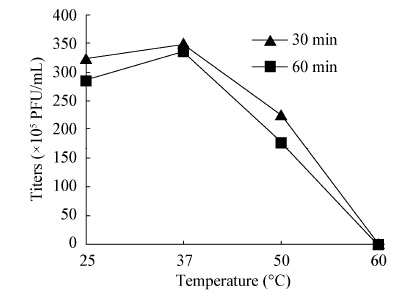
| 图 4 温度对噬菌体Guo1的影响 Figure 4 Influence of temperature on Guo1 |
|
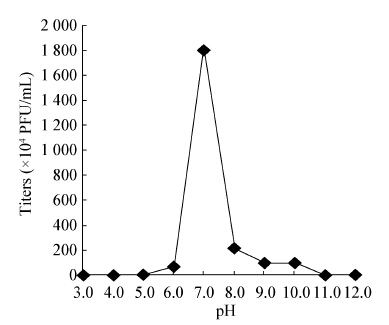
| 图 5 pH对噬菌体Guo1活性的影响 Figure 5 Influence of pH on the activity of Guo1 |
多数噬菌体基因组为线性双链DNA,少数为环形DNA,也有部分噬菌体的遗传物质为RNA,而噬菌体Guo1是目前所发现的分枝杆菌G群噬菌体中唯一具有环状基因组结构的[19,20],这值得我们进一步研究G群噬菌体之间的进化关系。重复序列的含量在不同的生物种类之间差异很大,如智人基因组中重复序列占50%,而病毒基因组的重复序列只占1%[21]。重复序列中含有丰富的遗传信息,参与基因表达的调控,在噬菌体Guo1中发现了3个串联重复序列,它们在基因表达中所起的作用尚不清楚。
国外的报道指出[22,23]:烈性噬菌体需要比溶源性噬菌体更多的tRNA用于转录,推测可能是溶源性噬菌体可以利用宿主体内的tRNA而不需要自身提供tRNA所致,在Guo1的基因组中未发现一个tRNA,提示Guo1属于溶源性噬菌体;基因12的推定基因与本实验室前期测序的噬菌体Leo整合酶基因[18]相似,推断为整合酶基因,且基因10的推定基因与噬菌体Sedge阻遏酶基因相似,一般认为同时具有这两种酶才能保证噬菌体的溶源性[24],结合以上两种证据支持噬菌体Guo1属于溶源性噬菌体。
采用高感染复数的方法测定噬菌体一步生长曲线,可以认为初始加入的宿主菌全部被感染;未能吸附宿主菌的噬菌体通过高倍稀释避免二次吸附,造成对后续时间点噬菌体滴度的影响。一步生长曲线测得Guo1潜伏期120 min,明显小于课题组前期研究的分枝杆菌噬菌体Legendre[25](潜伏期180 min)和Chy2[26](潜伏期210 min),通常认为潜伏期短的噬菌体具有更好的临床应用前景,而裂解量大小对噬菌体的裂解能力的影响存在争议[27,28]。Guo1对酒精、高热、氯仿敏感,对紫外线表现出抵抗性,在实验操作中可以选择酒精和高压灭菌来进行消毒;目前大多数实验室都使用紫外灯照射消毒,该噬菌体对紫外线抵抗性强,应该避免单用紫外线消毒。Guo1的最适pH在7.0左右,人体正常的血液pH在7.35−7.45之间,推测Guo1可以在血液pH的环境下用于结核病的治疗,此外结核菌为胞内寄生菌,造成耐药的结核菌常存在于巨噬细胞溶酶体内,溶酶体的pH为5.0,此时Guo1仍可以生存,推测噬菌体具有杀灭胞内结核菌的潜力。
随着抗生素的广泛使用,耐药现象不仅发生在其他细菌性疾病,在结核分枝杆菌中也同样变得日益严峻,第一代抗结核药(异烟肼,利福平,吡嗪酰胺和乙胺丁醇)使用近四十年后,目前还没有一种新药投入市场,能够杀灭耐多药结核菌和超级耐药结核菌[29]。由于噬菌体杀灭细菌具有专一性强,不会破坏正常菌群[30,31],且用量少具有放大效应的特性[32],逐渐成为研究的重点,并取得了一定进展,Wilson等就利用结核分枝杆菌噬菌体D29能够裂解结核菌的特性建立了一种快速检验结核菌的新技术PhaB[33]。噬菌体生物学特性和遗传背景的研究是利用噬菌体的基础。
Piuri等[10]发现噬菌体TM4野生株与噬菌体TM4 motif 3缺陷株相比:将基因组DNA注入耻垢分枝杆菌休眠菌的能力更强,而将基因组DNA注入对数生长期耻垢分枝杆菌的能力无差别,这可能提示复苏休眠菌是具有Motif3基序的噬菌体的特有作用。我课题组前期实验验证噬菌体TM4[12]能促使体外休眠菌复苏,初步研究发现噬菌体Guo1 (数据未列出)同样可以复苏休眠菌,生物信息学分析显示TM4和Guo1的尾部蛋白均包含Motif 3基序,具有与Rpf相似的水解肽聚糖的能力,然而Rpf在体外通过水解细胞壁肽聚糖促使休眠菌复苏的机制尚不清楚。在目前已经完成测序的分枝杆菌噬菌体G群中Guo1是唯一具有环状基因组的,在明确了噬菌体Guo1的生物学特性和基因组结构后,本课题组正在进一步研究Motif 3是否参与甚至主导了体内结核休眠菌的复苏以及G群噬菌体之间的进化关系。
| [1] | World Health Organization. Global tuberculosis report 2012[M]. Geneva: WHO Library, 2012: 89 |
| [2] | Technical Guidance Group of the Fifth National TB Epidemiological Survey, The Office of the Fifth National TB Epidemiological Survey. The fifth national tuberculosis epidemiological survey in 2010[J]. Chinese Journal of Antituberculosis, 2012, 34(8): 485-486 (in Chinese) 全国第五次结核病流行病学抽样调查技术指导组, 全国第五次结核病流行病学抽样调查办公室. 2010年全国第五次结核病流行病学抽样调查报告[J]. 中国防痨杂志, 2012, 34(8): 485-486 |
| [3] |
Gomez JE, McKinney JD. |
| [4] | Er J, Zhu XW, Liu ZQ. Resuscitating effect for dormant tuberculosis by recombinant resuscitation promoting factor (Rpf) proteins[J]. China Medical Engineering, 2008, 16(2): 92-95 (in Chinese) 佴静, 朱雪薇, 刘忠泉. 复苏因子促进休眠结核杆菌复苏作用的研究[J]. 中国医学工程, 2008, 16(2): 92-95 |
| [5] | Keep NH, Ward JM, Cohen-Gonsaud M, et al. Wake up! Peptidoglycan lysis and bacterial non-growth states[J]. Trends in Microbiology, 2006, 14(6): 271-276 |
| [6] | Telkov MV, Demina GR, Voloshin SA, et al. Proteins of the Rpf (resuscitation promoting factor) family are peptidoglycan hydrolases[J]. Biochemistry Biokhimiia, 2006, 71(4): 414-422 |
| [7] |
McNerney R, Kambashi BS, Kinkese J, et al. Development of a bacteriophage phage replication assay for diagnosis of |
| [8] | Kumar V, Loganathan P, Sivaramakrishnan G, et al. Characterization of temperate phage Che12 and construction of a new tool for diagnosis of tuberculosis[J]. Tuberculosis (Edinburgh, Scotland), 2008, 88(6): 616-623 |
| [9] |
Trigo G, Martins TG, Fraga AG, et al. Phage therapy is effective against infection by |
| [10] | Piuri M, Hatfull GF. A peptidoglycan hydrolase motif within the mycobacteriophage TM4 tape measure protein promotes efficient infection of stationary phase cells[J]. Molecular Microbiology, 2006, 62(6): 1569-1585 |
| [11] | Pedulla ML, Ford ME, Houtz JM, et al. Origins of highly mosaic Mycobacteriophage genomes[J]. Cell, 2003, 113(2): 171-182 |
| [12] |
Gan YL, Guo SL. Preliminary study on resuscitation of mycobacteriophage TM4 in dormant cells of |
| [13] | Zhang Z, Schwartz S, Wagner L, et al. A greedy algorithm for aligning DNA sequences[J]. Journal Computational Biology, 2000, 7(1/2): 203-214 |
| [14] |
Lu Z, Breidt F Jr, Fleming HP, et al. Isolation and characterization of a |
| [15] |
Huang JJ, Hu XM, Rao XC, et al. Biological characteristics of |
| [16] | Gan YL, Liu P, Wu TT, et al. Genetic information of mycobacteriophages DNAⅢ and its anti-tuberculosis potential[J]. Journal of Shanghai Jiao Tong University (Medical Science Edition), 2013, 33(10): 1323-1327 (in Chinese) 甘易玲, 刘平, 邬亭亭, 等. 分枝杆菌噬菌体DNAⅢ的遗传学信息及抗结核潜力初步研究[J]. 上海交通大学学报: 医学版, 2013, 33(10): 1323-1327 |
| [17] |
Gan YL, Wu TT, LIU P, et al. Characterization and classification of Bo4 as a cluster G mycobacteriophage that can infect and lyse |
| [18] | Jiang LS, Wu TT, Liu P, et al. Genetic information and biological characteristics of the mycobacteriophage Leo[J]. Acta Medicinae Universitatis Scientiae et Technologiae Huazhong, 2014, 43(5): 506-511 (in Chinese) 江莉莎, 邬亭亭, 刘平, 等. 分枝杆菌噬菌体Leo的生物学和基因组学特征[J]. 华中科技大学学报: 医学版, 2014, 43(5): 506-511 |
| [19] | Hatfull GF, Jacobs-Sera D, Lawrence JG, et al. Comparative genomic analysis of 60 Mycobacteriophage genomes: genome clustering, gene acquisition, and gene size[J]. Journal of Molecular Biology, 2010, 397(1): 119-143 |
| [20] | Hatfull GF. Complete genome sequences of 138 mycobacteriophages[J]. Journal of Virology, 2012, 86(4): 2382-2384 |
| [21] | Ai DY. The meanings of repeat sequences on genomes[J]. Chemistry of life, 2008, 28(3): 343-345 (in Chinese) 艾队元. 基因组中重复序列的意义[J]. 生命化学, 2008, 28(3): 343-345 |
| [22] | de Paepe M, Taddei F. Virusesʼ life history: towards a mechanistic basis of a trade-off between survival and reproduction among phages[J]. PLoS Biology, 2006, 4(7): e193 |
| [23] | Bailly-Bechet M, Vergassola M, Rocha E. Causes for the intriguing presence of tRNAs in phages[J]. Genome Research, 2007, 17(10): 1486-1495 |
| [24] | Broussard GW, Oldfield LM, Villanueva VM, et al. Integration-dependent bacteriophage immunity provides insights into the evolution of genetic switches[J]. Molecular Cell, 2013, 49(2): 237-248 |
| [25] | Liu P, Wu TT, Peng L, et al. Biological characteristics of mycobacteriophage Legendre and its potentials in drug resistant tuberculosis control[J]. Journal of third military medical university, 2012, 34(9): 821-825 (in Chinese) 刘平, 邬亭亭, 彭丽, 等. 噬菌体Legendre的生物学特性及抗耐药结核潜力的初步研究[J]. 第三军医大学学报, 2012, 34(9): 821-825 |
| [26] | Liu P, Wu TT, Peng L, et al. Isolation and biological characteristics of mycobacteriophage Chy2[J]. Journal of third military medical university, 2012, 34(12): 1176-1180 (in Chinese) 刘平, 邬亭亭, 彭丽,等. 分枝杆菌噬菌体Chy2的分离及生物学特性[J]. 第三军医大学学报, 2012, 34(12): 1176-1180 |
| [27] |
Niu DY. Use of bacteriophage to control |
| [28] |
Zhou YB, Shen XD, Li M, et al. The biological characterization of |
| [29] | Kaneko T, Cooper C, Mdluli K. Challenges and opportunities in developing novel drugs for TB[J]. Future Medicinal Chemistry, 2011, 3(11): 1373-1400 |
| [30] | Zhao QY, Zhu RL. Recent advances and prospects in bacteriophage therapy[J]. China Journal of Veterinary Medicine, 2010, 44(7): 40-43 (in Chinese) 赵庆友, 朱瑞良. 噬菌体制剂的研究现状及发展前景[J]. 中国兽药杂志, 2010, 44(7): 40-43 |
| [31] | Hu ZY, Cai LT. Research progress on phage therapy of bacterial plant disease[J]. Guizhou Agricultural Sciences, 2011, 39(3): 101-103 (in Chinese) 胡重怡, 蔡刘体. 噬菌体治疗作物细菌性病害的研究进展[J]. 贵州农业科学, 2011, 39(3): 101-103 |
| [32] |
Guenther S, Huwyler D, Richard S, et al.Virulent bacteriophage for efficient biocontrol of |
| [33] |
Wilson SM, Al-Suwaidi Z, McNerney R, et al. Evaluation of a new rapid bacteriophage-based method for the drug susceptibility testing of |
 2015, Vol. 42
2015, Vol. 42