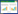中国科学院微生物研究所、中国微生物学会主办
文章信息
- 申玉玉, 陈忠秀, 陈杰, 赵宝顶, 吕佳, 桂玲, 路福平, 黎明
- SHEN Yuyu, CHEN Zhongxiu, CHEN Jie, ZHAO Baoding, LÜ Jia, GUI Ling, LU Fuping, LI Ming
- 一种高效无选择标记的黑曲霉基因组编辑方法
- An efficient marker-free genome editing method for Aspergillus niger
- 生物工程学报, 2022, 38(12): 4744-4755
- Chinese Journal of Biotechnology, 2022, 38(12): 4744-4755
- 10.13345/j.cjb.220162
-
文章历史
- Received: March 3, 2022
- Accepted: April 24, 2022
- Published: June 1, 2022
黑曲霉培养成本低,极端酸性环境中有强大的稳定性,具有较高的蛋白分泌能力和食品安全性,是一个不可多得的细胞工厂,是有机酸和酶制剂的重要工业生产菌株[1-4]。随着黑曲霉基因组信息的公布,越来越多的功能未知的DNA序列涌入视野,它们的功能鉴定及生物学特性研究成了后基因组时代的研究热点。不论是细胞工厂的构建还是基因功能的研究,往往都涉及基因组中不同基因的编辑。高效的黑曲霉基因编辑体系是进行黑曲霉基因功能研究和细胞工厂构建的关键技术之一[5]。
基于CRISPR/Cas9系统的新一代基因编辑技术已经成为生物学研究中重要的一部分。由于CRISPR/Cas9介导的基因编辑技术具有高度的灵活性、简单性和较高的编辑效率,已经被广泛应用于各个物种[6]。在利用CRISPR/Cas9技术进行基因编辑时,sgRNA的高效表达和成熟是进行高效基因编辑的关键。在真核生物中,sgRNA的表达主要依赖两类启动子:被RNA聚合酶Ⅱ识别的Ⅱ型启动子和被RNA聚合酶Ⅲ识别的Ⅲ型启动子。Ⅱ型启动子需要在sgRNA的5′端加上HH核酶、3′端加HDV核酶才能保证sgRNA的正常成熟,不仅编辑效率有待提高,而且sgRNA的构建相对烦琐[7]。与Ⅱ型启动子相比,Ⅲ型启动子不需要HH和HDV核酶,构建相对简单,但Ⅲ型启动子具有种属的特异性,使用也受到一些限制。Zheng等[8]使用Ⅲ型RNA聚合酶启动子——U6启动子启动sgRNA的表达,基因编辑效率最高达79%。为了提高基因编辑效率,Zheng等[9]创建了以5S rRNA启动子作为sgRNA表达启动子的CRISPR/Cas9系统,该系统基因编辑效率高达100%,说明5S rRNA启动子是一个非常好的启动sgRNA表达的启动子。
但是,Zheng等[9]建立的CRISPR/Cas9系统进行基因编辑时,需要在基因组中整合选择标记(潮霉素抗性基因表达盒) 帮助筛选。真菌的选择标记有限,标记的回收利用耗时较长或需要先构建缺陷型菌株;而且,在工程菌株的改造过程中应尽量避免选择标记的累积,避免抗性标记整合到基因组。随着黑曲霉的基因组编辑技术的发展,无痕编辑和多基因编辑成了重要的研究方向[5]。无痕编辑后的基因组中未插入选择标记,这样就避免了连续基因编辑时标记的回收利用,大大缩短了基因编辑的周期。CRISPR/Cas9基因编辑技术和具有复制起始功能的AMA1 (autonomously maintained in Aspergillus) 片段的发现,为建立丝状真菌的无痕编辑技术奠定了基础。1991年,Gems等[10]首次从构巢曲霉中分离出携带AMA1片段的质粒pARp1,发现AMA1片段在丝状真菌的质粒中具有复制起始功能。1994年,Verdoes等[11]发现含有AMA1片段的质粒在黑曲霉中转化效率提高10−100倍,之后AMA1片段被广泛用于黑曲霉的质粒构建。1997年,Aleksenko等[12]研究表明含有AMA1片段的质粒在没有选择压力下极易丢失。它的这一不稳定特性被用于丝状真菌CRISPR/Cas9的基因编辑,短暂地表达Cas9蛋白和(或) sgRNA,避免向基因组中整合Cas9蛋白基因和Cas9蛋白持续表达带来的脱靶效应;同时在基因编辑完成后很容易通过无抗平板传代丢失质粒,解决了抗性质粒存在于细胞内不易丢失的问题。2016年,Pohl等[13]首次在产黄青霉菌中利用携带AMA1片段的线性化质粒YN-2、hCas9-sgRNA片段和sgRNA- amdS片段共转化宿主细胞进行无选择标记的基因组编辑。但供体DNA需要1 kb左右的同源臂,且编辑载体构建比较复杂,编辑效率也有待提高。2019年,Van Leeuwe等[14]在黑曲霉中进行了无选择标记的基因组编辑研究,采用PtRNAPro1启动sgRNA的表达,利用携带AMA1片段的质粒pFC332-sgRNA共表达sgRNA和Cas9蛋白,结果一些基因的编辑效率仅为66.7%,且供体DNA同样需要较长的同源臂(800−1 000 bp)。编辑效率低的原因可能是PtRNAPro1启动sgRNA表达的效率不高。2020年,Vanegas等[15]在黑曲霉中实现了仅有60 bp同源臂的无选择标记供体DNA进行的无选择标记的基因编辑,但是他们使用的是CRISPR/ LbCpf1系统,而且编辑效率有待提高,原因可能与采用的是U3启动子启动sgRNA的表达效率不高有关。
自然条件下丝状真菌进行同源重组(homologous recombination, HR) 的概率很低,原因是在丝状真菌中双链DNA的断裂修复时,非同源末端连接(non-homologous end joining pathway, NHEJ) 优先于HR,所以很多研究通过阻断NHEJ修复来提高HR的效率[16-18]。Meyer等[16]研究表明,敲除kusA (Ku70蛋白编码基因) 可以阻断NHEJ修复,获得高效的基因靶向突变体,与野生型相比HR提高70%;而且kusA基因的缺失并不会给菌株生长造成明显影响。本研究利用5S rRNA启动子启动sgRNA的表达,构建了基于AMA1复制起始片段的sgRNA和Cas9共表达质粒;同时通过敲除kusA基因,获得一株NHEJ修复缺陷的高效同源重组菌株;最后利用含有AMA1片段质粒的不稳定特性通过无抗平板传代实现抗性质粒的丢失,从而建立了一种高效无选择标记的黑曲霉基因组编辑方法。该方法在采用20 bp同源臂的无选择标记供体DNA进行基因编辑时,基因编辑效率也达到100%。该方法为黑曲霉基因功能的研究和细胞工厂的构建奠定了基础。
1 材料与方法 1.1 材料 1.1.1 菌株和培养基本研究所用的出发菌株为果胶酶黑曲霉生产菌AnP1.0,大肠杆菌(Escherichia coli) DH5α用于构建质粒,所用菌株见表 1。本研究使用的培养基有PDA、CM、MM、MMS、MMS上层琼脂,所有培养基参照Arentshorst等[19]描述的配制,使用的MMS培养基未添加咖啡因,与转化液混合的MMS上层琼脂添加潮霉素(hygromycin B, hyg) 抗性。
制备原生质体所用试剂按照Arentshorst等[19]描述配制,PrimeSTAR Max DNA Polymerase、T4 DNA Ligase、Bgl Ⅱ、BamH Ⅰ、Nhe Ⅰ、Xho Ⅰ等限制性核酸内切酶购自宝生物工程(大连) 有限公司,Pac Ⅰ和Bbs Ⅰ为NEB公司生产,潮霉素B购自天津百奥泰科技有限公司。
1.1.3 引物本实验所用引物如表 2所示。
| Primer name | Primer sequences (5′→3′) | Size (bp) |
| sgRNA-albA1 F | CACCAGTGGGATCTCAAGAACTAC | 24 |
| sgRNA-albA1 R | AAACGTAGTTCTTGAGATCCCACT | 24 |
| sgRNA-prtT F | CACCAGATGTGATCTAGATCCGCG | 24 |
| sgRNA-prtT R | AAACCGCGGATCTAGATCACATCT | 24 |
| GHsgRNA-kusA1 F | CACCTGATGATAAGACCCTACGGT | 24 |
| GHsgRNA-kusA1 R | AAACACCGTAGGGTCTTATCATCA | 24 |
| GHsgRNA-kusA3 F | CACCAGAAAGGCATATAAGTTCGG | 24 |
| GHsgRNA-kusA3 R | AAACCCGAACTTATATGCCTTTCT | 24 |
| AlbA 100 F | GACGAGCTCCACGAAGTCT | 19 |
| AlbA 100 R | TGGCCTTGAATGACGCGG | 18 |
| DFkusA F | AACCAGATATTCACATCCAGAGCGC | 25 |
| DFkusA R | ccgcgcggatctagatcacatctACCGTAGGGTCTTATCATCACCATGG | 49 |
| DRkusA F | agatgtgatctagatccgcgcggAGACCAAGTAACCTTTTCGCCTGAT | 48 |
| DRkusA R | TGGAAGCGCAGTGATAGGCTTGA | 24 |
| sgRNA L F | CCGCTCGAGAACTGCTGAGATCTCACATACGACCACAGG | 39 |
| sgRNA L R | CGCGGATCCAAAAAAGCACCGACTCGG | 27 |
| D60kusA F | GGCGCTTGTTCATCATAACCGAC | 23 |
| D60kusA R | ACTGGCTCACCGAAGTCCC | 19 |
| D40kusA F | GACAATGACAACCCCCATGGTG | 22 |
| D40kusA R | GAGCGCCTTCTGTTCATCAGG | 21 |
| D20 kusAF | TGATGATAAGACCCTACGGT | 20 |
| D20 kusAR | GCGAAAAGGTTACTTGGTCT | 20 |
| lowercase letters – 23 bp prtT sequence. | ||
sgRNA以5S rRNA为启动子进行表达,启动子序列参照Zheng等[9]设计,在5′端添加Xho Ⅰ和Bgl Ⅱ的酶切位点,在3′端添加Pac Ⅰ的酶切位点,送华大基因合成。靶位点通过CRISPOR (http://crispor.tefor.net/) 或者CHOPCHOP (http://chopchop.cbu.uib.no/) 在线设计,通过Bbs Ⅰ酶切pUC57-sgRNA将合成的靶位点序列连接到质粒中,构建pUC57- sgRNA-target的靶向质粒(图 1A)。用Bgl Ⅱ/Pac Ⅰ酶切pUC57-sgRNA-target质粒得到线性的sgRNA-target表达盒片段,再用同样的酶酶切pFC332,将酶切后的pFC332载体与sgRNA-target表达盒片段连接,构建pFC332_target-sgRNA靶向质粒(图 1B)。
|
| 图 1 质粒及供体DNA的构建A:sgRNA表达盒的构建;B:pFC332_target-sgRNA的构建;C:sgRNA表达盒的串联;D:无选择标记供体DNA片段构建。图用BioRender.com在线制作 Fig. 1 Schematic representation of the plasmid and donor DNA construction. (A) Construction of sgRNA cassette. (B) Construction of pFC332_target-sgRNA. (C) Thread of the sgRNA cassettes. (D) Construction of marker-free donor DNA fragments. Created with BioRender.com. |
| |
进行多靶位点基因编辑时,需串联两个或两个以上的sgRNA表达盒。先分别构建单个靶位点的sgRNA表达盒质粒pUC57-sgRNA-target 1、pUC57-sgRNA-target 2等,然后用sgRNA表达盒的上游引物和添加BamH Ⅰ酶切位点的下游引物PCR扩增表达盒sgRNA-target 2,用Xho Ⅰ/BamH Ⅰ酶切sgRNA表达盒2片段;用Xho Ⅰ/Bgl Ⅱ酶切pUC57-sgRNA-target 1,将酶切后的载体和片段连接构建含有两个靶位点sgRNA表达盒质粒pUC57-sgRNA-target 2-sgRNA-target 1 (图 1C);同理可以构建出含有多个靶位点的sgRNA表达盒质粒。将构建的pUC57-sgRNA-target 2-sgRNA-target 1质粒用Bgl Ⅱ/ Pac Ⅰ酶切得到sgRNA-target 2-sgRNA-target 1片段,再将酶切后的sgRNA表达盒连接到pFC332载体上,构建pFC332-sgRNA-target 2-sgRNA-target 1靶向质粒(图 1B)。无选择标记的敲除供体DNA片段选取靶位点两端的左右各100 bp的同源臂,中间由一个20 bp的核苷酸和PAM序列连接,两对引物重叠PCR得到供体DNA片段(图 1D)。供体kusA片段由图 1D引物1和4 PCR扩增AnP1.1基因组得到,缩短的供体DNA片段由相应引物扩增供体kusA片段得到。
1.2.3 原生质体转化及转化子筛选原生质体的制备及转化参照Arentshorst等[19]描述,转化子平板培养5−7 d,挑取转化子于MM+200 µg/mL hyg平板,30 ℃培养2−3 d,再次传代并挑取适量菌丝于1.5 mL离心管中,加50 µL 10×TE和少量石英砂,振荡使菌丝分散,置于微波炉700 W加热2 min,然后立即冰浴2 min,13 000×g离心1 min,吸取10 µL上清作模板。用验证引物进行PCR扩增靶向基因,送华大基因测序验证或琼脂糖凝胶电泳验证。
1.2.4 靶向质粒的丢失携带AMA1片段的质粒极不稳定,在无选择压力下易丢失。挑取验证正确的转化子在MM无抗板上进行单孢子划线,以丢失Cas9-sgRNA质粒。再挑取单菌落于MM平板上划线,划出尽可能多的单菌落。选取多个单菌落分别在MM平板和MM+200 µg/mL hyg平板上划线作丢失质粒的对照,在MM平板上生长同时在抗性平板上不生长的为质粒丢失菌株,可用于下一次的基因编辑。大多数菌株经两次无抗平板传代后,会出现质粒丢失菌株。
2 结果与分析 2.1 CRISPR/Cas9系统构建及编辑效率的验证构建的CRISPR/Cas9系统以潮霉素为转化筛选标记的自主复制型质粒pFC332[20]来同时表达Cas9蛋白和sgRNA (图 1B)。Cas9基因序列来源于酿脓链球菌,按照黑曲霉密码子的偏好性优化并在其3′端添加了核定位信号SV40。Cas9由构巢曲霉的强组成型启动子tef1启动表达、终止子tef1终止表达。由5S rRNA启动子启动sgRNA的表达,5S rRNA在胞内被RNA聚合酶Ⅲ转录,为Cas9-sgRNA复合物的形成提供充足的sgRNA;5S rRNA具有明确的结构(5S rRNA结构由http://rna.tbi.univie.ac.at//cgi-bin/RNAWebSuite/RNAfold.cgi网站预测),既不妨碍暴露的spacer,也不干扰sgRNA骨架的结构,可以有效形成Cas9-sgRNA复合物并结合到靶位点进行基因编辑(图 3A)。为了测试CRISPR/Cas9系统的作用,我们选择一个假定聚酮合酶(PKS) 的基因albA为黑曲霉的靶基因。albA编码的PKS是合成黑色素的第一个关键酶,其突变体为可见的分生孢子白化表型。根据方法1.2.1构建靶向质粒pFC332_albA- sgRNA (表 3),转化黑曲霉菌株AnP1.0,通过孢子颜色的变化来检测CRISPR/Cas9系统的基因编辑效率。
| Plasmid name | Gene name | An# (gene) | Target sequences | PAM | References |
| pUC57_albA-sgRNA | albA | An09g05730 | AGTGGGATCTCAAGAACTAC | TGG | This study |
| pUC57_kusA3-sgRNA | kusA | An15g02700 | TGATGATAAGACCCTACGGT | CGG | This study |
| pUC57_kusA1-sgRNA | kusA | An15g02700 | AGAAAGGCATATAAGTTCGG | TGG | This study |
| pUC57_prtT-sgRNA | - | - | AGATGTGATCTAGATCCGCG | CGG | This study |
| pFC332_albA-sgRNA | albA | An09g05730 | AGTGGGATCTCAAGAACTAC | TGG | This study |
| pFC332_kusA3-1-sgRNA | kusA | An15g02700 | TGATGATAAGACCCTACGGT | CGG | This study |
| AGAAAGGCATATAAGTTCGG | TGG | ||||
| pFC332_prtT-sgRNA | - | - | AGATGTGATCTAGATCCGCG | CGG | This study |
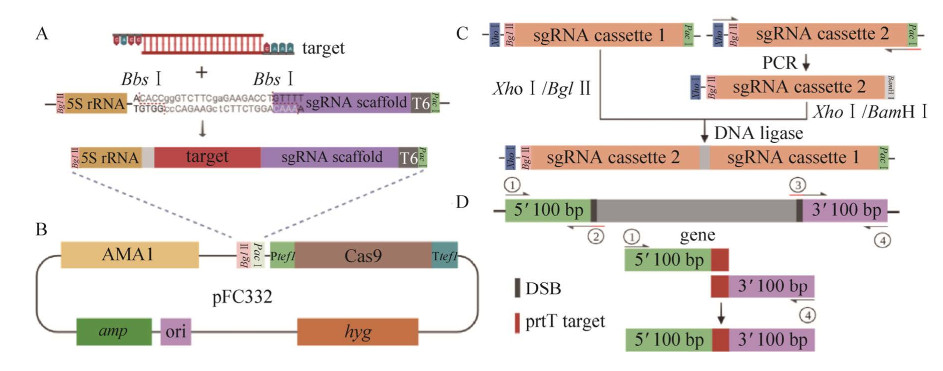
为了测试sgRNA表达盒是以片段形式还是以质粒形式存在基因编辑效率高,我们测试了两种方法:一种是用sgRNA-albA表达盒片段与pFC332质粒共转化,另一种将sgRNA- albA表达盒片段插入到pFC332中构建成pFC332_albA-sgRNA质粒进行转化。然后随机从潮霉素筛选平板上各随机挑取转化子接种到MM抗性平板上培养并观察孢子颜色(图 2B)。结果表明:当用sgRNA-albA表达盒片段与pFC332质粒共转化时,70个转化子中仅一个转化子的孢子颜色变成灰白色(图 2B红圈所示),albA的基因编辑效率仅为1.4%;而用pFC332_albA-sgRNA质粒进行转化时,59个转化子的孢子颜色全部变成了灰白色,albA的基因编辑效率达到了100%,说明将sgRNA表达盒克隆到pFC332质粒中进行转化的基因编辑方式明显优于线性片段sgRNA表达盒与pFC332共转化的基因编辑方式,这可能与黑曲霉转化困难和线性片段在胞内容易被降解有关[17-18]。当sgRNA-albA表达盒片段与pFC332质粒共转化时共有70个转化子,在潮霉素抗性平板上均能生长但69个转化子孢子颜色仍表现为黑色,说明质粒已经转入但未完成基因编辑。因此,在后续的基因编辑实验中,采用sgRNA表达盒克隆到pFC332质粒上进行。随机挑取部分转化子提取基因组,用引物AlbA 100 F和AlbA 100 R进行PCR扩增靶位点区域,并进行测序验证,结果如图 2A所示:靶位点区域都发生了基因突变,基因编辑方式是部分碱基插入或碱基缺失突变。其中21号和42号菌株基因组测序结果显示,靶位点处缺失7个碱基,30号菌株的靶基因测序靶位点处插入一个碱基,均发生了移码突变。挑选一个已测序的转化子在MM无抗平板传代丢失质粒,如方法1.2.4所示,经两次无抗平板传代后,约有85%的转化子丢失质粒(12/14),挑选一个质粒丢失的转化子保存为AnP1.1,用于后续的基因编辑。
|
| 图 2 albA基因的编辑A:转化子albA基因部分序列比对;B:转化子菌落颜色。左:用pFC332_albA-sgRNA质粒进行转化后转化子;右:pFC332和sgRNA表达盒片段共转化后的转化子 Fig. 2 Genome editing of A. niger at albA site. (A) Alignment of partial sequences of albA gene. (B) The colony color of transformants. Left: transformants for pFC332_albA-sgRNA; right: transformants for pFC332 and sgRNA cassette. |
| |
kusA编码的Ku70蛋白是DNA双链断裂修复NHEJ途径的关键蛋白,研究表明,kusA基因的敲除可造成NHEJ修复途径缺陷,有助于提高丝状真菌的同源重组效率[16]。本研究通过敲除kusA基因构建NHEJ缺陷型菌株,提高了菌株的同源重组效率。
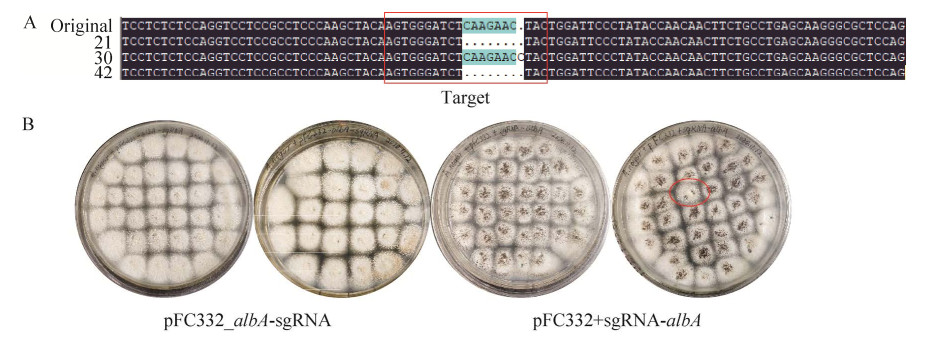
kusA基因敲除选择双靶点策略,断裂两个位点,产生两个双链断裂缺口,同时整合上无选择标记的供体DNA片段(图 3C)。供体DNA片段选取两断裂位点左右各100 bp同源臂,中间加上一个23 bp的靶点序列prtT (20 bp核苷酸序列和PAM序列),构建成同源重组片段DR-prtT-DR。将质粒pFC332_kusA3-1-sgRNA和同源重组片段DR-prtT-DR共同转化到AnP1.1菌株中,随机挑选6个转化子提取基因组,用引物DFkusA F和DRkusA R PCR扩增验证。若发生了同源重组,PCR后琼脂糖凝胶电泳条带为223 bp,否则条带为730 bp。电泳结果如图 3B所示,其中,3个转化子条带在250 bp左右,发生同源重组修复。表明在NHEJ非缺陷的条件下,该菌株发生同源重组的效率为50%。随机选取已同源重组的菌株测序验证,测序比对结果显示,prtT靶点序列已正确整合到基因组的kusA基因位点(图 3F),表明kusA基因被敲除,NHEJ修复缺陷型菌株构建成功。按方法1.2.4传代丢失质粒,获得NHEJ缺陷菌株AnP2.1。
|
| 图 3 基因编辑策略及验证A:albA基因编辑示意图;B:kusA基因敲除验证;C:kusA基因敲除策略;D:NHEJ缺陷型菌株同源重组示意图;E:kusA基因同源重组的PCR验证;F:prtT靶点测序比对图。X代表敲除的基因序列。图A、C和D用BioRender.com在线制作 Fig. 3 A. niger genome editing strategies and validation. (A) Schematic diagram of the albA gene editing. (B) Diagnostic PCR of the kusA locus. (C) The knockout strategy of kusA gene. (D) Schematic diagram of homologous recombination of NHEJ-deficient strain. (E) Diagnostic PCR of the kusA locus homologous recombination. (F) Alignment of partial sequences of prtT. X: knockout sequence. Figure A, C and D were created with BioRender.com. |
| |
为了检测AnP2.1菌株的同源重组效率,本研究在基因组的prtT位点整合一段无选择标记的kusA基因片段(图 3D)。将质粒pFC332_ prtT-sgRNA和供体kusA (730 bp) 转入菌株AnP2.1,挑取所有转化子提取基因组,用引物DFkusA F和DRkusA R PCR扩增验证。琼脂糖凝胶电泳结果如图 3E所示,所有转化子条带在730 bp左右,表明转化子均已整合上供体DNA,发生同源重组效率为100%。实验结果表明,NHEJ缺陷型菌株在使用本研究构建的高效的pFC332/5S rRNA-sgRNA基因编辑系统,同时提供730 bp的供体DNA片段的条件下,菌株发生同源重组的效率为100%。
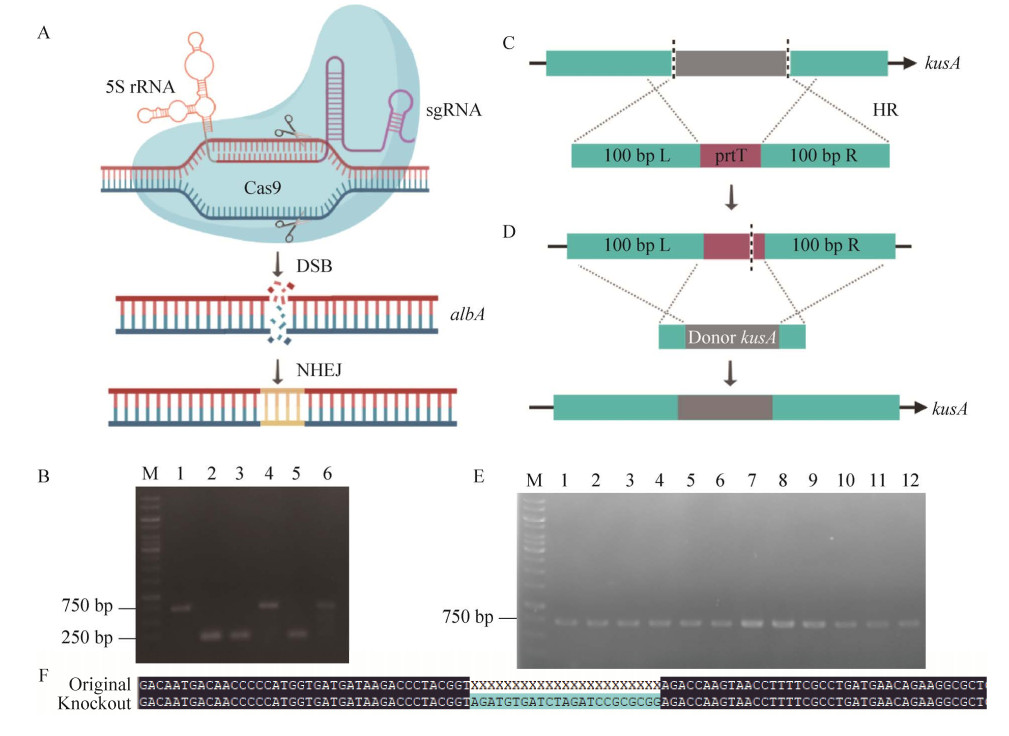
2.4 供体DNA同源臂长度的优化为探究不同长度同源臂的供体DNA对基因编辑的影响,本研究设计了不同长度同源臂的供体DNA进行转化实验。在上述2.3中的供体kusA的同源臂分别缩短至60、40和20 bp,再次转化AnP2.1。培养4 d后观察,共转化不同长度同源臂的供体片段。在供体片段浓度相同的条件下,获得的转化子数量不同,同源臂长度越长,转化子的数量越多。挑取不同长度同源臂的所有转化子提取基因组,分别用D60kusA F / D60kusA R、D40kusA F / D40kusA R和D20kusA F / D20kusA R进行PCR验证,若整合上供体DNA,正确条带大小分别为650、610、570 bp。琼脂糖凝胶电泳结果如图 4所示,所有转化子条带均显示已整合上供体DNA。在提供60、40、20 bp同源臂的无筛选标记的供体DNA片段条件下,菌株发生同源重组概率均为100%。表明本研究构建的CRISPR/Cas9系统,即使采用20 bp长度的同源臂供体DNA,也可以进行高效的无痕基因组编辑。

|
| 图 4 不同长度同源臂供体DNA的转化子PCR验证1−10:60 bp同源臂;11−17:40 bp同源臂;18−19:20 bp同源臂 Fig. 4 Diagnostic PCR of the transformants edited with homologous arms of different length. Lane 1−10: 60 bp homologous arms. Lane 11−17: 40 bp homologous arms. Lane 18−19: 20 bp homologous arms. |
| |
黑曲霉是一类重要的丝状真菌,在重组蛋白的分泌表达、次级代谢产物的生产和环境污染治理等方面都具有重要的应用价值。构建成熟高效的黑曲霉基因编辑系统对我们挖掘其潜在的应用价值具有重要意义。本研究利用5S rRNA启动子启动sgRNA的高效表达,构建了一个基于AMA1复制起始片段的sgRNA和Cas9共表达质粒,用该质粒转化黑曲霉菌株进行基因编辑时,基因编辑效率达到100%。但是,若要整合一个外源供体DNA片段到目的基因位点时,定点整合的基因编辑效率降低到了50%,这可能是由于黑曲霉中NHEJ修复优先于HR修复所致。为了提高同源重组效率,进一步敲除了黑曲霉基因组中的kusA基因,构建一株NHEJ修复缺陷型的高效同源重组菌株AnP2.1。当sgRNA和Cas9的共表达质粒与同源臂长度为20 bp的无选择标记供体DNA共转化NHEJ修复缺陷菌株AnP2.1时,外源供体DNA可以高效地整合到目的基因位点,而且定点整合的基因编辑效率也达到了100%。表明本研究建立了一种高效无选择标记的黑曲霉基因组编辑方法。该方法不仅实现了高效无选择标记的基因组编辑,还极大地缩短了供体DNA片段同源臂的长度,利用一对引物即可构建出含有上下游同源臂的供体DNA片段,简化了供体DNA片段的构建过程。需要注意的是,在加入等量供体DNA片段的条件下,同源臂长度越短,转化子数量越少。当使用20 bp同源臂的供体DNA进行基因编辑时,增加供体DNA的量可以提高转化子的数量。
本研究构建的黑曲霉CRISPR/Cas9系统中,Cas9表达盒和sgRNA表达盒在同一个载体共表达,与sgRNA表达盒片段和Cas9表达质粒共转化宿主细胞相比,基因编辑效率更高,这可能是由于Cas9和sgRNA表达盒位于同一载体上时,保证它们同时转化入宿主细胞并实现高效表达,并防止sgRNA表达盒片段单独转化时的降解。黑曲霉转化相对来说比较困难,当sgRNA表达盒片段与携带有筛选标记的Cas9表达盒质粒共转化时,抗性平板只能筛选出转入了携带抗性标记的Cas9表达盒质粒,而无法保证sgRNA表达盒片段是否转入宿主细胞。这个表达盒片段是否转入到宿主菌株以及转入量的多少,都会影响基因编辑效率[9, 21]。此外,胞内存在大量的核酸酶,线性的sgRNA表达盒片段在胞内容易降解,导致基因编辑效率降低[22]。5S rRNA启动子是一个基因内启动子,5′末端融合有5S rRNA启动子的sgRNA模板序列可以被RNA聚合酶Ⅲ高效识别并大量转录[23],而且5S rRNA融合在sgRNA的5′端不影响Cas9-sgRNA复合物的形成与功能[9],这可能是5S rRNA启动子启动sgRNA的表达可有效提高基因编辑效率的关键因素之一。本系统还设计了一个含有BglⅡ和BamHⅠ同尾酶的中间质粒pUC57-sgRNA,通过这对同尾酶可以简单高效地进行多个sgRNA表达盒的串联,从而构建含有多个sgRNA表达盒的sgRNA和Cas9共表达质粒,实现多基因的同时编辑。
利用CRISPR/Cas9技术进行基因组编辑时,为了筛选出目的转化子,一般需要在编辑的基因位点插入选择标记基因以增加基因编辑的效率[8-9, 24]。由于真菌的选择标记有限,若要进行多个基因的连续编辑,标记的去除大大地增加了工作量和工作难度,且工业生产菌株的改造需尽量避免在基因组中累积选择标记或构建营养缺陷型菌株。本研究构建了一个高效率无选择标记的编辑系统,它既不需要在基因组中整合选择标记,也不产生营养缺陷型菌株,可轻松实现多个基因的连续编辑,适宜于工业生产菌株的构建。
| [1] |
Jin HX, OuYang XK, Hu ZC. Enhancement of epoxide hydrolase production by 60Co gamma and UV irradiation mutagenesis of Aspergillus niger ZJB-09103. Biotechnol Appl Biochem, 2017, 64(3): 392-399. DOI:10.1002/bab.1502
|
| [2] |
Wang SY, Jiang BL, Zhou X, et al. Study of a high-yield cellulase system created by heavy-ion irradiation-induced mutagenesis of Aspergillus niger and mixed fermentation with Trichoderma reesei. PLoS One, 2015, 10(12): e0144233. DOI:10.1371/journal.pone.0144233
|
| [3] |
Meyer V, Fiedler M, Nitsche B, et al. The cell factory Aspergillus enters the big data era: opportunities and challenges for optimising product formation. Adv Biochem Eng Biotechnol, 2015, 149: 91-132.
|
| [4] |
Behera BC. Citric acid from Aspergillus niger: a comprehensive overview. Crit Rev Microbiol, 2020, 46(6): 727-749. DOI:10.1080/1040841X.2020.1828815
|
| [5] |
郑小梅, 郑平, 孙际宾. 基于CRISPR/Cas系统的黑曲霉基因组编辑技术. 生物工程学报, 2021, 37(3): 980-990. Zheng XM, Zheng P, Sun JB. CRISPR/Cas-based genome editing in Aspergillus niger. Chin J Biotech, 2021, 37(3): 980-990 (in Chinese). |
| [6] |
郑小梅, 张晓立, 于建东, 等. CRISPR-Cas9介导的基因组编辑技术的研究进展. 生物技术进展, 2015, 5(1): 1-9, I0003, F0003. Zheng XM, Zhang XL, Yu JD, et al. CRISPR- Cas9-based genome engineering. Curr Biotechnol, 2015, 5(1): 1-9, I0003, F0003 (in Chinese). |
| [7] |
Gao YB, Zhao YD. Self-processing of ribozyme- flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. J Integr Plant Biol, 2014, 56(04): 343-349. DOI:10.1111/jipb.12152
|
| [8] |
Zheng XM, Zheng P, Sun JB, et al. Heterologous and endogenous U6 snRNA promoters enable CRISPR/ Cas9 mediated genome editing in Aspergillus niger. Fungal Biol Biotechnol, 2018, 5: 2. DOI:10.1186/s40694-018-0047-4
|
| [9] |
Zheng XM, Zheng P, Zhang K, et al. 5S rRNA promoter for guide RNA expression enabled highly efficient CRISPR/Cas9 genome editing in Aspergillus niger. ACS Synth Biol, 2019, 8(7): 1568-1574. DOI:10.1021/acssynbio.7b00456
|
| [10] |
Gems D, Johnstone IL, Clutterbuck AJ. An autonomously replicating plasmid transforms Aspergillus nidulans at high frequency. Gene, 1991, 98(1): 61-67. DOI:10.1016/0378-1119(91)90104-J
|
| [11] |
Verdoes JC, Punt PJ, Van Der Berg P, et al. Characterization of an efficient gene cloning strategy for Aspergillus niger based on an autonomously replicating plasmid: cloning of the nicB gene of A. niger. Gene, 1994, 146(2): 159-165. DOI:10.1016/0378-1119(94)90288-7
|
| [12] |
Aleksenko A, Clutterbuck AJ. Autonomous plasmid replication in Aspergillus nidulans: AMA1 and MATE elements. Fungal Genet Biol, 1997, 21(3): 373-387. DOI:10.1006/fgbi.1997.0980
|
| [13] |
Pohl C, Kiel JAKW, Driessen AJM, et al. CRISPR/Cas9 based genome editing of Penicillium chrysogenum. ACS Synth Biol, 2016, 5(7): 754-764. DOI:10.1021/acssynbio.6b00082
|
| [14] |
Van Leeuwe TM, Arentshorst M, Ernst T, et al. Efficient marker free CRISPR/Cas9 genome editing for functional analysis of gene families in filamentous fungi. Fungal Biol Biotechnol, 2019, 6: 13. DOI:10.1186/s40694-019-0076-7
|
| [15] |
Vanegas KG, Jarczynska ZD, Strucko T, et al. Cpf1 enables fast and efficient genome editing in Aspergilli. Fungal Biol Biotechnol, 2019, 6: 6. DOI:10.1186/s40694-019-0069-6
|
| [16] |
Meyer V, Arentshorst M, El-Ghezal A, et al. Highly efficient gene targeting in the Aspergillus niger kusA mutant. J Biotechnol, 2007, 128(4): 770-775. DOI:10.1016/j.jbiotec.2006.12.021
|
| [17] |
Krappmann S, Sasse C, Braus GH. Gene targeting in Aspergillus fumigatus by homologous recombination is facilitated in a nonhomologous end- joining-deficient genetic background. Eukaryot Cell, 2006, 5(1): 212-215. DOI:10.1128/EC.5.1.212-215.2006
|
| [18] |
Takahashi T, Masuda T, Koyama Y. Enhanced gene targeting frequency in Ku70 and Ku80 disruption mutants of Aspergillus sojae and Aspergillus oryzae. Mol Genet Genomics, 2006, 275(5): 460-470. DOI:10.1007/s00438-006-0104-1
|
| [19] |
Arentshorst M, Ram AF, Meyer V. Using non-homologous end-joining-deficient strains for functional gene analyses in filamentous fungi. Methods Mol Biol, 2012, 835: 133-150.
|
| [20] |
Nødvig C S, Nielsen JB, Kogle ME, et al. A CRISPR-Cas9 system for genetic engineering of filamentous fungi. PLoS One, 2015, 10(7): e0133085. DOI:10.1371/journal.pone.0133085
|
| [21] |
姚善泾, 蔡礼年, 林东强. 黑曲霉作为分泌蛋白细胞工厂的研究进展. 化工学报, 2019, 70(10): 3690-3703. Yao SJ, Cai LN, Lin DQ. Progress in Aspergillus niger as cell factory for secretory proteins. CIESC J, 2019, 70(10): 3690-3703 (in Chinese). |
| [22] |
Huang CY, Guo LW, Wang JG, et al. Efficient long fragment editing technique enables large-scale and scarless bacterial genome engineering. Appl Microbiol Biotechnol, 2020, 104(18): 7943-7956. DOI:10.1007/s00253-020-10819-1
|
| [23] |
Ciganda M, Williams N. Eukaryotic 5S rRNA biogenesis. Wiley Interdiscip Rev RNA, 2011, 2(4): 523-533. DOI:10.1002/wrna.74
|
| [24] |
Dong HZ, Zheng JW, Yu D, et al. Efficient genome editing in Aspergillus niger with an improved recyclable CRISPR-HDR toolbox and its application in introducing multiple copies of heterologous genes. J Microbiol Methods, 2019, 163: 105655.
|
 2022, Vol. 38
2022, Vol. 38